C18orf63
| Identyfikatory | |||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| C18orf63 | |||||||||||||||||||||||||||||||||||||||||||||||
| , DKFZP781G0119, chromosom 18 otwarta ramka odczytu 63 | |||||||||||||||||||||||||||||||||||||||||||||||
| identyfikatory zewnętrzne | |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| Wikidane | |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
Otwarta ramka odczytu 63 chromosomu 18 jest białkiem , które u ludzi jest kodowane przez gen C18orf63 . Białko to nie jest jeszcze dobrze poznane przez społeczność naukową. Przeprowadzono badania sugerujące, że C18orf63 może być potencjalnym biomarkerem wczesnego stadium raka trzustki i raka piersi .
Gen
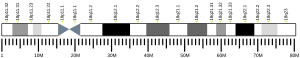
Gen ten znajduje się w prążku 22, podpasmo 3, na długim ramieniu chromosomu 18 . Składa się z 5065 par zasad rozciągających się od 74 315 875 do 74 359 187 pz na chromosomie 18. Gen ma łącznie 14 eksonów . C18orf63 jest również znany pod pseudonimem DKFZP78G0119. Dla tego genu nie istnieją żadne izoformy.
Wyrażenie
C18orf63 ma wysoką ekspresję w jądrach . Gen wykazuje niską ekspresję w nerkach, wątrobie, płucach i miednicy. Nie ma fenotypu związanego z tym genem.
Promotor
Region promotora dla C18orf63 ma długość 1163 pz, zaczynając od 74 314 813 pz i kończąc na 74 315 975 pz. Identyfikator promotora to GXP_4417391. Obecność wielu czynników transkrypcyjnych wiążących y-box i miejsc wiążących czynnik transkrypcyjny SRY sugeruje, że C18orf63 bierze udział w determinacji płci męskiej.
Białko
Białko C18orf63 składa się z 685 aminokwasów i ma masę cząsteczkową 77230,50 Da, z przewidywanym punktem izoelektrycznym 9,83. Dla tego białka nie izoformy . Białko to jest bogate w glutaminę , izoleucynę , lizynę i serynę w porównaniu do przeciętnego białka, ale brakuje mu kwasu asparaginowego i glicyny .
Struktura
W przewidywanej strukturze drugorzędowej tego białka występuje szereg skrętów beta , nici beta i helis alfa . Oczekuje się, że dla C18orf63 48,6% białka utworzy helisy alfa, a 28,6% struktury ma składać się z nici beta.
Domeny i motywy
Białko zawiera jedną domenę o nieznanej funkcji , DUF 4709, rozciągającą się od 7 do 280 aminokwasu. Przewiduje się, że motywy , które istnieją, obejmują motyw N-końcowy, motyw RxxL i motyw konserwujący KEN, które wszystkie sygnalizują degradację białka . Przewiduje się, że innym motywem , który istnieje, jest motyw Wxxx, który ułatwia wejście białek ładunkowych PTS1 do światła organelli, oraz motyw RVxPx, który umożliwia transport białka z sieci trans-Golgiego do błony plazmatycznej rzęsek . Na końcu sekwencji białkowej znajduje się również dwudzielny sygnał lokalizacji jądrowej . Nie ma domeny transbłonowej, co wskazuje, że C18orf63 nie jest białkiem transbłonowym.
Modyfikacje potranslacyjne
, że modyfikacje potranslacyjne, którym ulegnie białko, obejmują SUMOilację , fosforylację PKC i CK2 , N-glikozylację , amidację i cięcie. Istnieje sześć całkowitych miejsc fosforylacji PKC i 2 miejsca fosforylacji CK2, 2 miejsca SUMOilacji i 2 miejsca N-glikozylacji. W tej sekwencji nie ma peptydów sygnałowych.
Lokalizacja subkomórkowa
Ze względu na sygnał lokalizacji jądrowej na końcu sekwencji białkowej przewiduje się, że C18orf63 będzie jądrowy . Przewiduje się również, że C18orf63 będzie kierowany do mitochondriów oprócz jądra.
Homologia
ortologi
Ortologi znaleziono u większości eukariontów , z wyjątkiem klasy Amphibia . Nie istnieją żadne ludzkie paralogi dla C18orf63. Najdalszym wykrywalnym homologiem jest Mizuhopecten yessoensis , wykazujący 37% identyczności z sekwencją ludzkiego białka. Domena o nieznanej funkcji była jedyną domeną homologiczną obecną w sekwencji białka, stwierdzono, że jest wysoce konserwatywna we wszystkich ortologach. Poniższa tabela przedstawia kilka przykładów różnych ortologów tego białka.
| Rodzaj | Gatunek | Nazwa zwyczajowa | Numer dostępowy | Długość sekwencji | Tożsamość sekwencji | Podobieństwo sekwencji | |
| ssaki | Galeopter | różnorodny | Latający lemur | XP_008582575.1 | 677 | 78% | 87% |
| Fukomys | damarensis | Kretoszczur Damara | XP_019061329.1 | 654 | 70% | 81% | |
| Equus | przewalskiego | Koń Przewalskiego | XP_008534756.1 | 751 | 76% | 83% | |
| Loxodonta | afrykańska | Słoń afrykański | XP_023399495.1 | 676 | 73% | 83% | |
| Szynszyla | lanigera | Szynszyla długoogoniasta | XP_005373135.1 | 679 | 74% | 83% | |
| Ave | Korvus | Cornix | Wrona z kapturem | XP_019138065.2 | 743 | 52% | 69% |
| Sturnusa | pospolity | Szpak pospolity | XP_014726419.1 | 742 | 51% | 68% | |
| Strutio | kamelus | Południowy struś | XP_009668441.1 | 741 | 44% | 62% | |
| Phaethon | leptur | Białoogoniasty tropicbird | XP_010287785.1 | 740 | 44% | 60% | |
| Nestora | notatki | Kea | XP_010018784.1 | 741 | 43% | 60% | |
| gady | Ofiofag | Hanna | Kobra królewska | ETE73844.1 | 671 | 55% | 69% |
| Anolis | karolina | Anol Karolina | XP_008106943.1 | 719 | 48% | 66% | |
| Pogona | vitticeps | Środkowy brodaty smok | XP_020657479.1 | 676 | 52% | 70% | |
| Chrysemys | obraz | Malowany żółw | XP_008162704.1 | 770 | 45% | 60% | |
| Ryba | Callorhinchus | milicja | Australijski rekin widmo | XP_007901438.1 | 738 | 57% | 74% |
| Rhincodon | typu | Rekin wielorybi | XP_020370482.1 | 712 | 41% | 55% | |
| Salmo | wynagrodzenie | łosoś atlantycki | XP_0140366110.1 | 626 | 43% | 60% | |
| Bezkręgowce | stylofora | pistillata | Koral | XP_022802513.1 | 721 | 33% | 57% |
| Akantaster | plany | Rozgwiazda z korony cierniowej | XP_022082271.1 | 750 | 37% | 56% | |
| Mizuhopecten | taksoensis | Przegrzebek | OWF48219.1 | 260 | 37% | 57% |
Tempo ewolucji
C18orf63 jest wolno ewoluującym białkiem. Białko ewoluuje szybciej niż cytochorme C, ale wolniej niż betaglobina .
Białka oddziałujące
Czynniki transkrypcyjne będące przedmiotem zainteresowania, które mają wiązać się z sekwencją regulatorową, obejmują supresory guza p53 , czynniki determinujące jądra SRY , czynniki transkrypcyjne wiążące blok Y i elementy reagujące na glukokortykoidy . Stwierdzono, że białko JUN oddziałuje z C18orf63 poprzez koimmunoprecypitację antibait . Białko JUN wiąże się z promotorem USP28 w komórkach raka jelita grubego i bierze udział w aktywacji tych komórek rakowych.
Znaczenie kliniczne
Mutacje
W populacji ludzkiej występuje wiele mutacji zmiany sensu dla tego białka. W sekwencji regulatorowej mutacje zmiany sensu występują w dwóch miejscach wiązania czynnika transkrypcyjnego. Wpływ na czynniki transkrypcyjne to elementy reagujące na glukokortykoidy i regularne cykle komórkowe E2F-myc . Istnieje jedenaście powszechnych mutacji, które wpływają na samą sekwencję białka. Żadna z tych mutacji nie wpływa na przewidywane modyfikacje potranslacyjne, którym podlega sekwencja białka.
Związek chorobowy
C18orf63 został powiązany z zaburzeniami osobowości , otyłością i cukrzycą typu 2 poprzez badanie asocjacyjne całego genomu . Obecnie badania nie wykazały, czy C18orf63 odgrywa bezpośrednią rolę w którejkolwiek z tych chorób.