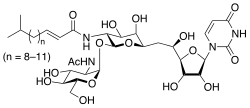
Tunikamycyna
|
|
| Nazwy | |
|---|---|
|
nazwa IUPAC
(E)-N-[(2S,3R,4R,5R,6R)-2-[(2R,3R,4R,5S,6R)-
3-acetamido-4,5-dihydroksy-6-(hydroksymetylo)oksan-2-ylo]oksy-6-[2-[(2R,3S,4R,5R)-5-(2,4-dioksopirymidyn-1- ylo)-3,4-dihydroksyoksolan-2-ylo]-2-hydroksyetylo]-4,5-dihydroksyoksan- 3-ylo]-5-metyloheks-2-enamid |
|
| Inne nazwy NSC 177382
|
|
| Identyfikatory | |
|
Model 3D ( JSmol )
|
|
| CHEMBL | |
| ChemSpider | |
| Karta informacyjna ECHA | 100.115.295 |
| Numer WE |
|
| Siatka | Tunikamycyna |
|
Identyfikator klienta PubChem
|
|
| UNII | |
|
Pulpit nawigacyjny CompTox ( EPA )
|
|
|
|
|
|
| Nieruchomości | |
| C 39 H 64 N 4 O 16 | |
| Masa cząsteczkowa | Nie dotyczy |
| Zagrożenia | |
| Oznakowanie GHS : | |

|
|
| Niebezpieczeństwo | |
| H300 | |
|
O ile nie zaznaczono inaczej, dane podano dla materiałów w stanie normalnym (przy 25°C [77°F], 100 kPa).
|
|
Tunikamycyna jest mieszaniną homologicznych antybiotyków nukleozydowych , która hamuje rodzinę enzymów UDP-HexNAc: poliprenol-P HexNAc-1-P. U eukariontów obejmuje to enzym fosfotransferazę GlcNAc (GPT), który katalizuje przeniesienie N-acetyloglukozamino-1-fosforanu z UDP-N-acetyloglukozaminy do fosforanu dolicholu w pierwszym etapie syntezy glikoproteiny . Tunikamycyna blokuje N-glikozylację (N-glikany) i traktowanie hodowanych komórek ludzkich tunikamycyną powoduje cykl komórkowy zatrzymanie w fazie G1 . Jest używany jako narzędzie eksperymentalne w biologii , np. do indukowania odpowiedzi niezwiniętego białka . Tunikamycyna jest wytwarzana przez kilka bakterii , w tym Streptomyces clavuligerus i Streptomyces lysosuperificus .
Homologi tunikamycyny mają różne masy cząsteczkowe ze względu na zmienność koniugatów łańcuchów bocznych kwasów tłuszczowych .
Biosynteza
Biosyntezę tunikamycyn badano w Streptomyces chartreusis i scharakteryzowano proponowany szlak biosyntezy. Bakterie wykorzystują enzymy w tun (TunA-N) do wytwarzania tunikamycyn.
TunA wykorzystuje jednostkę startową difosforanu urydyny-N-acetylo-glukozaminy (UDP-GlcNAc) i katalizuje odwodnienie grupy hydroksylowej 6'. Po pierwsze, reszta Tyr w TunA odrywa proton od grupy hydroksylowej 4', tworząc w tej pozycji keton. Wodorek jest następnie odrywany od węgla 4' przez NAD+, tworząc NADH. Keton jest stabilizowany przez wiązania wodorowe z reszty Tyr i pobliskiej reszty Thr. Następnie reszta glutaminianu odrywa proton od węgla 5', popychając elektrony w górę, tworząc podwójne wiązanie między węglem 5' i 6'. Pobliska cysteina przekazuje proton grupie hydroksylowej, gdy opuszcza ją jako woda. NADH przekazuje wodorek do węgla 4', reformując wodorotlenek w tej pozycji i tworząc UDP-6'-deoksy-5-6-en-GlcNAc. TunF następnie katalizuje epimeryzację związku pośredniego do UDP-6'-dezoksy-5-6-en-GalNAc, zmieniając 4' grupę hydroksylową z pozycji równikowej na osiową.
Inną jednostką startową tunikamycyny jest urydyna, która jest wytwarzana z trójfosforanu urydyny (UTP). TunN jest difosfatazą nukleotydów i katalizuje usuwanie pirofosforanu z UTP z wytworzeniem monofosforanu urydyny. Ostatni fosforan jest usuwany przez domniemaną monofosfatazę TunG.
Po wytworzeniu urydyny i UDP-6'-deoksy-5-6-en-GalNAc, TunB katalizuje ich wiązanie na węglu 6' UDP-6'-deoksy-5-6-en-GalNAc. TunB wykorzystuje S-adenylmetioninę (SAM) do utworzenia rodnika na węglu 5' rybozy na uracylu. Uważa się, że TunM katalizuje tworzenie nowego wiązania między węglem 5' urydyny a węglem 6' UDP-6'-deoksy-5-6-en-GalNAc przy użyciu elektronu z rodnika urydyny i jednego z elektronów z podwójnego wiązania UDP-6'-dezoksy-5-6-en-GalNAc. Rodnik na UDP-6'-dezoksy-5-6-en-GalNAc jest następnie gaszony przez oderwanie wodoru od SAM. Powstała cząsteczka to UDP-N-acetylo-tunikamina. TunH następnie katalizuje hydrolizę UDP z UDP-N-acetylo-tunikaminy. Wprowadza się kolejną cząsteczkę UDP-GlcNAc, a następnie tworzy się wiązanie β-1,1-glikozydowe, katalizowane przez TunD. Powstała cząsteczka jest deacetylowana przez TunE. TunL i tłuszczowa ligaza acylo-ACP są używane do ładowania metabolicznych kwasów tłuszczowych na acylowe białko nośnikowe, TunK. TunC następnie przyłącza kwas tłuszczowy do wolnej aminy, wytwarzając tunikamycynę.
Zobacz też
- Glikozylacja - tunikamycyna blokuje wszelką N-glikozylację białek
- glikoproteina
- rodzaju Streptomyces
Linki zewnętrzne
- Część książkowa Essentials in Glycobiology (1999) Tunicamycin: Hamowanie montażu DOL-PP-GlcNAc
- Karta charakterystyki tunikamycyny przygotowana przez studenta Open University w Wielkiej Brytanii