Kompleks połączeń egzonowych
Kompleks złącza eksonowego ( EJC ) jest kompleksem białkowym , który tworzy się na pre - informacyjnej nici RNA na połączeniu dwóch eksonów , które zostały połączone ze sobą podczas składania RNA . EJC ma duży wpływ na translację , nadzór i lokalizację splicingu mRNA . Najpierw osadza się na mRNA podczas składania, a następnie jest transportowany do cytoplazmy . Tam odgrywa główną rolę w potranskrypcyjnej regulacji mRNA. Uważa się, że kompleksy połączeń eksonów zapewniają specyficzną dla pozycji pamięć zdarzenia splicingu. EJC składa się ze stabilnego heterotetramerowego rdzenia, który służy jako platforma wiążąca dla innych czynników niezbędnych dla szlaku mRNA. Rdzeń EJC zawiera białkowy czynnik inicjacji eukariotycznej 4A-III ( eIF4A-III ; helikaza RNA typu DEAD-box ) związany z analogiem trifosforanu adenozyny ( ATP ), jak również dodatkowe białka Magoh i Y14 . Wiązanie tych białek z domenami nakrapianymi w jądrze zostało niedawno zmierzone i może być regulowane przez szlaki sygnałowe PI3K/AKT/mTOR . Aby doszło do związania kompleksu z mRNA, następuje zahamowanie czynnika eIF4AIII, co zatrzymuje hydrolizę ATP. To rozpoznaje EJC jako kompleks zależny od ATP. EJC oddziałuje również z dużą liczbą dodatkowych białek; przede wszystkim białka SR. Sugeruje się, że te interakcje są ważne dla zagęszczania mRNA. Rola EJC w eksporcie mRNA jest kontrowersyjna.
Składniki białkowe
EJC składa się z kilku kluczowych składników białkowych: między innymi RNPS1 , Y14, SRm160 , Aly/REF i Magoh. RNPS1 może działać jako koaktywator splicingu, ale wraz z Y14 bierze również udział w procesie rozpadu nonsensownego u eukariontów. SRm160 to kolejny koaktywator, który zaproponowano w celu wzmocnienia przetwarzania końca 3' mRNA. Uważa się, że składnik białkowy Magoh ułatwia subcytoplazmatyczną lokalizację mRNA, podczas gdy Aly jest zaangażowana w eksport jądrowego mRNA. Uważa się, że Aly jest rekrutowana do kompleksu połączenia egzonu przez białko UAP56 . UAP56 jest rozpoznawany jako helikaza RNA, ale działa jako czynnik splicingowy wymagany do wczesnego składania spliceosomu. Innym czynnikiem zaangażowanym w szlak EJC jest DEK . Wiadomo, że ten składnik bierze udział w różnych funkcjach, począwszy od splicingu, a skończywszy na regulacji transkrypcji i strukturze chromatyny .
Struktura
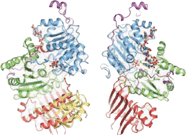
Krystalizacja kompleksu złącza egzonowego ujawniła strukturalną organizację jego składników białkowych . Rdzeń kompleksu jest wydłużony o całkowity wymiar 99Å na 67Å na 54Å. Jest zorganizowany wokół czynnika eIF4AIII. Sam czynnik składa się z dwóch różnych typów konformacji wokół mRNA: zamkniętej i otwartej. W stanie zamkniętym dwie domeny eIF4AIII tworzą złożone miejsca wiązania dla 5'-adenylilo-β,γ-imidodifosforanu (ADPNP) i mRNA. W konformacji otwartej obie domeny są obrócone o 160 stopni względem stanu zamkniętego18. Składniki białkowe Magoh i Y14 wiążą się ze sobą, tworząc heterodimer zlokalizowany na biegunie 5' EJC. Magoh wiąże się z domeną eIF4AIII poprzez interakcje między resztami z jej dwóch C-końcowych helis i jednym końcem dużego arkusza β . Konserwatywne reszty w łączniku między dwiema domenami eIF4AIII tworzą mostki solne lub wiązania wodorowe ze specyficznymi resztami w Magoh. Inne wiązanie zachodzi między drugą pętlą arkusza β Magoh a dwiema domenami eIF4AIII i ich łącznikiem. Istnieje tylko jedno wiązanie częściowe utworzone między Y14 i eIF4AIII. Składa się on z mostka solnego pomiędzy konserwowanymi resztami Y14 Arg108 i eIF4AIII Asp401 . Gdyby mutacje wystąpiły w obu tych resztach, połączenie Magoh-Y14 z EJC nie istniałoby.
Mechanizm
Podczas drugiego etapu splicingu w komórkach eukariotycznych, EJC jest odkładana około 20-24 nukleotydów od końca 5' powyżej połączenia splicingowego (gdzie łączą się dwa egzony), kiedy uformował się lariat i eksony zostały zligowane razem. Wiązanie EJC z mRNA zachodzi w sposób niezależny od sekwencji, tworząc dojrzałą matrycową rybonukleoproteinę (mRNP). EJC pozostaje stabilnie związana z tym mRNP, gdy jest eksportowana z jądra do cytoplazmy. Składniki białkowe są albo wiązane, albo uwalniane przez EJC podczas transportu. Aby doszło do translokacji mRNA przez kompleks porów jądrowych, heterodimer składający się z NXF1 /TAP i NXT1 / p15 musi związać się z transkryptami. NXF1/TAP jest głównym receptorem eksportu mRNA do cytoplazmy. Dzieje się tak, ponieważ oddziałuje zarówno z białkami adaptorowymi wiążącymi RNA, jak i składnikami kompleksu porów jądrowych .
Rozpoznanie przedwczesnego kodonu terminacyjnego następuje podczas translacji w cytoplazmie. Obraz pokazany poniżej sugeruje, że to zdarzenie jest jądrowe, w przeciwieństwie do ogólnego poglądu w tej dziedzinie. Czytelnicy powinni mieć świadomość, że tłumaczenie w jądrze jest wysoce kontrowersyjnym tematem, który nie jest dobrze poparty danymi. [ potrzebne źródło ]
W nonsensownym rozpadzie
Kompleksy połączeń egzonowych odgrywają główną rolę w nadzorze mRNA . Dokładniej, znajdują się one w rozpadu, w którym pośredniczy nonsens (NMD), w którym transkrypty mRNA z przedwczesnymi kodonami stop są degradowane. W normalnej translacji mRNA rybosom wiąże się z transkryptem i rozpoczyna wydłużanie łańcucha aminokwasów . Kontynuuje, aż dotrze do lokalizacji kompleksu połączenia eksonu, który następnie wypiera. Następnie translacja jest zakończona, gdy rybosom osiągnie kodon terminacji . W NMD transkrypt mRNA zawiera kodon przedwczesnej terminacji (PTC) z powodu mutacji nonsensownej . Jeśli ten kodon występuje przed miejscem EJC, EJC pozostanie związany, powodując rozpad mRNA. EJC i jego pozycja służą jako rodzaj regulatora, określającego, czy transkrypt jest wadliwy, czy nie.
Wiadomo również, że EJC biorą udział w NMD w inny sposób; rekrutację czynników nadzoru UPF1 , UPF2 i UPF3 . Białka te są najważniejszymi składnikami mechanizmu NMD. Białko EJC MAGOH, Y14 i eIF4AIII zapewniają wiązanie UPF3, które działa jako pomost między UPF2 i UPF1, tworząc trimeryczny kompleks. W obrębie tego kompleksu UPF2 i UPF3 działają wspólnie, promując ATPazę i helikazę RNA UPF1. Rdzeń EJC stabilnie zakotwicza kompleks UPF do mRNA i pomaga w regulacji niezbędnego białka UPF1. Rybosomy, które utknęły w martwym punkcie na PTC, rekrutują UPF1 poprzez interakcje z czynnikami uwalniającymi eRF1 i eRF3 . Wraz z białkiem SMG1 , eRF1, eRF3 i UPF1 tworzą kompleks SURF. Kompleks ten tworzy pomost między rybosomem a EJC w dół, który jest związany z UPF3 i UPF2. Ta interakcja wyzwala fosforylację UPF1 przez SMG1, powodując dysocjację eRF1 i eRF3. Wytworzony kompleks składa się z EJC, UPF3, UPF2, fosforylowanego UPF1 i SMG1, co z kolei powoduje degradację mRNA.