kokolithowirus

|
|
| Coccolithovirus | |
|---|---|
| Giant coccolithovirus, Emiliania huxleyi virus 86 (strzałka), infekujący Emiliania huxleyi coccolithophore | |
| Klasyfikacja wirusów | |
| (nierankingowe): | Wirus |
| królestwo : | Varidnaviria |
| Królestwo: | Bamfordvirae |
| Gromada: | Nucleocytoviricota |
| Klasa: | Megawirycety |
| Zamówienie: | Algavirales |
| Rodzina: | Phycodnaviridae |
| Rodzaj: | kokolithowirus |
| Szczepy | |
|
|
Coccolithovirus to rodzaj gigantycznego dwuniciowego wirusa DNA z rodziny Phycodnaviridae . Algi, w szczególności Emiliania huxleyi , gatunek coccolithophore , służą jako naturalni gospodarze. W rodzaju tym występuje tylko jeden opisany gatunek: Emiliania huxleyi virus 86 .
Struktura
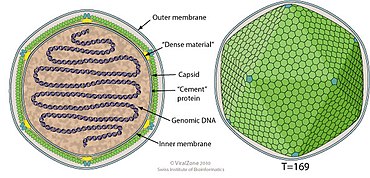
Kokolitowirusy są otoczkowe, dwudziestościenne i mają średnicę w zakresie 100–220 nm. Ich genomy są liniowe, mają długość od 410 do 415 kb i przewidują kodowanie około 472 białek.
| Rodzaj | Struktura | Symetria | Kapsyd | Układ genomowy | Segmentacja genomu |
|---|---|---|---|---|---|
| kokolithowirus | dwudziestościenny | T=169 | Koperta | Liniowy | jednoczęściowy |
Koło życia
Kokolitowirusy należą do rodziny Phycodnaviridae , jednej z pięciu rodzin należących do dużej i zróżnicowanej filogenetycznie grupy wirusów znanych jako wirusy nukleocytoplazmatyczne dużego dsDNA ( NCLDV ). Wirusy te albo replikują się wyłącznie w cytoplazmie komórki gospodarza, albo rozpoczynają swój cykl życiowy w jądrze gospodarza , ale kończą go w cytoplazmie. W przypadku EhV-86 strategia infekcji nie jest w pełni zrozumiała, ale Mackinder i in. (2009) zaproponowali następujący model: Wirus dostaje się do komórki gospodarza poprzez endocytozę , po której następuje fuzja jego błony lipidowej z błoną wakuolową gospodarza i uwolnienie rdzenia nukleoproteinowego do cytoplazmy. Alternatywnie błona wirusowa może łączyć się bezpośrednio z błoną plazmatyczną gospodarza. Genom wirusa jest następnie uwalniany z kapsydu do jądra, gdzie jest replikowany przez wirusową polimerazę DNA . Zreplikowany genom jest pakowany w zmontowane kapsydy w cytoplazmie i uważa się, że nowo utworzone (do 400–1000) wirionów są transportowane do błony plazmatycznej i uwalniane przez kontrolowany mechanizm pączkowania, co prowadzi do rozpadu komórkowego komórki gospodarza . [ potrzebne źródło ]
Podczas fazy G2 i M cyklu życiowego kokosfera jest niekompletna, a ekspozycja błony plazmatycznej na wirusa jest zwiększona. Nawet przy nienaruszonej kokosferze może wystąpić infekcja z powodu naturalnie występujących przerw między kokolitami.
E. huxleyi znana jest z tworzenia sezonowych zakwitów glonów, które mogą osiągnąć 250 000 km 2 , podczas których gęstość komórek w górnych 200 m wzrasta z 10 3 do 10 5 komórek na ml wody morskiej. Te zakwity glonów zanikają zwykle po 5–8 dniach, a kilka badań wykazało, że zakończenie kwitnienia jest nierozerwalnie związane z infekcją kokkolitynowirusami. Przenoszenie wirusów między żywicielami glonów odbywa się poprzez bierną dyfuzję. Co więcej, DNA EhV wykryto również u widłonogów , co prowadzi do wniosku, że wirusy są dalej rozprzestrzeniane przez przenoszący wirusy zooplankton .
Genom
Do tej pory w latach 1999-2008 wyizolowano 14 szczepów EhV, głównie z kanału La Manche (EC), ale także z wybrzeża Norwegii i Szkocji. Chociaż dostępne są częściowe sekwencje wszystkich tych 14 szczepów, EhV-86 jest jedynym szczepem, który został w pełni zsekwencjonowany ze względu na wysoce powtarzalny charakter genomu. Sekwencjonowanie EhV-86 ujawniło kolisty genom o długości 407 339 bp z 472 przewidywanymi sekwencjami kodującymi ( CDS ). Co ciekawe, 80% z tych domniemanych genów nie ma jak dotąd żadnych homologów w bazie danych. Te, którym można przypisać funkcję ze względu na podobieństwo sekwencji lub dopasowanie domen białkowych, obejmują polimerazy DNA i RNA , osiem proteaz, a także co najmniej cztery geny kodujące białka zaangażowane w biosyntezę sfingolipidów. Wykazano, że zostały one pozyskane od gospodarza poprzez poziomy transfer genów .
Ponadto genom EhV-86 ujawnił trzy różne rodziny (A, B, C) powtarzalnych regionów w genomie. Rodzina C składa się z powtórzeń bogatych w AT, które są niekodujące i które prawdopodobnie są częścią początku replikacji ( ORF ). Rodzina B to powtórzenia bogate w GC, które znajdują się w produktach białkowych ośmiu przewidywanych CDS. Regiony homologiczne rodziny A różnią się wielkością między 30–300 pz i znajdują się w części genomu o wielkości 104 kbp (200–304 kbp), która nie zawiera homologów genów o znanej funkcji w aktualnych bazach danych. Jednostki powtarzalne rodziny A są niekodujące i charakteryzują się nanomerem (GTTCCC(T/C)AA), który w sumie pojawia się w 106 miejscach w tym regionie. Ta sekwencja znajduje się bezpośrednio powyżej 86 CDS i prawdopodobnie odgrywa rolę w kontrolowaniu ekspresji powiązanych CDS. [ potrzebne źródło ]
Podczas infekcji opisano wyraźny wzorzec ekspresji genów wirusowych, który można podzielić na trzy fazy zgodnie z ekspresją CDS. Godzinę po zakażeniu transkrybowano 39 genów wirusowych, a następnie kolejne 194 geny po 2 godzinach i 71 genów po 4 godzinach. Wszystkie 39 genów, które ulegają ekspresji 1 godzinę po zakażeniu, znajdują się w regionie 104 kbp i mają nanomer bezpośrednio przed kodonem start. Ponieważ ekspresja wirusowej polimerazy RNA nie została wykryta 1 godzinę po zakażeniu, nie ustalono jeszcze, czy promotor jest rozpoznawany przez upakowaną wirusową polimerazę RNA, czy przez polimerazę RNA gospodarza. Analiza proteomiczna wirionu EhV-86 nie wykazała jednak żadnych głównych podjednostek polimerazy RNA.
Historia
Wilson i jego zespół z Marine Biological Association (MBA), University of East Anglia i Plymouth Marine Laboratory (PML) po raz pierwszy zaobserwowali wirusa w 1999 roku. Później latem 2005 roku naukowcy z Plymouth Marine Laboratory (Willie Wilson i in. ) oraz w Sanger Institute (Holden i in.) zsekwencjonowali genom szczepu EhV-86, stwierdzając, że ma on 472 geny kodujące białka, co czyni go „gigantycznym wirusem” i największym znanym wirusem morskim pod względem genomu.
Na podstawie wstępnych badań genomu Coccolithoviruses odkryto sekwencję genów odpowiedzialnych za produkcję ceramidu . Ceramid jest czynnikiem kontrolującym śmierć komórek i obecnie uważa się, że Coccolithovirus wykorzystuje go do przedłużenia życia Emilianii huxleyi , podczas gdy wykorzystuje komórkę gospodarza do replikacji. Jest to wyjątkowa zdolność niespotykana dotychczas w żadnym innym genomie wirusowym. [ potrzebne źródło ]
Zobacz też
- Mimivirus - największy gigantyczny wirus w historii według genomu
- Mycoplasma genitalium , Pelagibacter ubique – jedne z najmniejszych znanych bakterii
- Nanoarchaeum – najmniejsze znane archaeum
- Najmniejsze organizmy
- Parwowirusy – najmniejsza znana rodzina wirusów
- Phycodnaviridae – wirusy infekujące glony
Notatki
Dalsza lektura
- Wilson, WH, Schroeder, DC, Allen, MJ, Holden, MTG, Parkhill, J., Barrell, BG, Churcher, C., Hamlin, N., Mungall, K., Norbertczak, H., Quail, MA, Cena , C., Rabbinowitsch, E., Walker, D., Craigon, M., Roy, D. i Ghazal, P. (2005) Kompletna sekwencja genomu i profil transkrypcji fazy litycznej wirusa Coccolithovirus . Nauka 5737 , 1090-1092
- Allen, MJ, Schroeder, DC, Holden, MT i Wilson, WH (2006) Ewolucyjna historia Coccolithoviridae . Mol Biol Evol. 23 , 86–92 ( wymagane logowanie w Atenach )