Genotypowanie SNP
Genotypowanie SNP to pomiar zmienności genetycznej polimorfizmów pojedynczych nukleotydów (SNP) między członkami gatunku. Jest to forma genotypowania , która jest pomiarem bardziej ogólnej zmienności genetycznej. SNP są jednym z najczęstszych rodzajów zmienności genetycznej. SNP to mutacja pojedynczej pary zasad w określonym locus , zwykle składająca się z dwóch alleli (gdzie częstość rzadkich alleli wynosi > 1%). Stwierdzono, że SNP są zaangażowane w etiologię wielu chorób człowieka i stają się przedmiotem szczególnego zainteresowania farmakogenetyka . Ponieważ SNP są konserwowane podczas ewolucji, zaproponowano je jako markery do wykorzystania w analizie loci cech ilościowych ( QTL ) oraz w badaniach asocjacyjnych zamiast mikrosatelitów . Wykorzystanie SNP jest rozszerzane w ramach HapMap , którego celem jest dostarczenie minimalnego zestawu SNP potrzebnych do genotypowania ludzkiego genomu. SNP mogą również zapewnić genetyczny odcisk palca do wykorzystania w testach tożsamości. Wzrost zainteresowania SNP znalazł odzwierciedlenie w gwałtownym rozwoju różnorodnych metod genotypowania SNP.
Metody oparte na hybrydyzacji
Opracowano kilka aplikacji, które badają SNP poprzez hybrydyzację komplementarnych sond DNA z miejscem SNP. Wyzwaniem tego podejścia jest ograniczenie hybrydyzacji krzyżowej między sondami specyficznymi dla alleli. Wyzwanie to jest na ogół pokonywane przez manipulowanie rygorystycznymi warunkami hybrydyzacji.
Dynamiczna hybrydyzacja specyficzna dla alleli
Genotypowanie dynamicznej hybrydyzacji specyficznej dla alleli (DASH) wykorzystuje różnice w temperaturze topnienia DNA, które wynikają z niestabilności niedopasowanych par zasad. Proces ten można znacznie zautomatyzować i obejmuje on kilka prostych zasad. [ potrzebne źródło ]
W pierwszym etapie segment genomowy jest amplifikowany i przyłączany do kulki w reakcji PCR z biotynylowanym starterem. W drugim etapie zamplifikowany produkt dołącza się do ze streptawidyną i przemywa NaOH w celu usunięcia niebiotynylowanej nici. Następnie dodaje się oligonukleotyd specyficzny dla allelu w obecności cząsteczki, która fluoryzuje po związaniu z dwuniciowym DNA. Następnie mierzy się intensywność, gdy temperatura wzrasta, aż można określić temperaturę topnienia (Tm) . SNP spowoduje niższą niż oczekiwano Tm.
Ponieważ genotypowanie DASH mierzy wymierną zmianę w Tm, jest w stanie zmierzyć wszystkie typy mutacji, nie tylko SNP. Inne zalety DASH obejmują możliwość pracy z sondami bez etykiet oraz prostą konstrukcję i warunki działania. [ potrzebne źródło ]
Latarnie molekularne
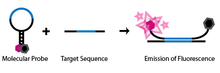
Wykrywanie SNP za pomocą latarni molekularnych wykorzystuje specjalnie zaprojektowaną jednoniciową sondę oligonukleotydową. Oligonukleotyd jest zaprojektowany tak, że na każdym końcu znajdują się regiony komplementarne, a pomiędzy nimi znajduje się sekwencja sondy. Taka konstrukcja pozwala sondzie przyjąć strukturę spinki do włosów lub pętelkowej struktury w jej naturalnym, izolowanym stanie. Do jednego końca sondy przymocowany jest fluorofor, a do drugiego wygaszacz fluorescencji. Ze względu na pętlową strukturę sondy, fluorofor znajduje się blisko wygaszacza, zapobiegając w ten sposób emisjom fluorescencji przez cząsteczkę. Cząsteczka jest również zaprojektowana w taki sposób, że tylko sekwencja sondy jest komplementarna do genomowego DNA, które będzie użyte w teście (Abravaya i in. 2003).
Jeśli sekwencja sondy latarni molekularnej napotka docelowy genomowy DNA podczas testu, nastąpi hybrydyzacja i hybrydyzacja. Ze względu na długość sekwencji sondy, segment spinki do włosów sondy zostanie zdenaturowany na korzyść utworzenia dłuższej, bardziej stabilnej hybrydy sonda-cel. Ta zmiana konformacyjna pozwala fluoroforowi i wygaszaczowi uwolnić się od ich ciasnej bliskości z powodu połączenia spinki do włosów, umożliwiając cząsteczce fluorescencję.
Z drugiej strony, jeśli sekwencja sondy napotka sekwencję docelową zawierającą zaledwie jeden niekomplementarny nukleotyd, latarnia molekularna preferencyjnie pozostanie w swoim naturalnym stanie spinki do włosów i nie będzie obserwowana żadna fluorescencja, ponieważ fluorofor pozostaje wygaszony.
Unikalna konstrukcja tych molekularnych sygnałów nawigacyjnych pozwala na prosty test diagnostyczny w celu identyfikacji SNP w danej lokalizacji. Jeśli latarnia molekularna jest zaprojektowana tak, aby pasowała do allelu typu dzikiego, a druga do mutanta allelu, można ich użyć do identyfikacji genotypu osobnika. Jeśli podczas testu zostanie wykryta tylko długość fali fluoroforu pierwszej sondy, osobnik jest homozygotyczny względem typu dzikiego. Jeśli wykryta zostanie tylko długość fali drugiej sondy, osobnik jest homozygotyczny względem zmutowanego allelu. Wreszcie, jeśli wykryte zostaną obie długości fal, wówczas oba sygnały molekularne muszą hybrydyzować z ich komplementarnymi, a zatem osobnik musi zawierać oba allele i być heterozygotyczny.
mikromacierze SNP
W macierzach SNP oligonukleotydów o dużej gęstości , setki tysięcy sond są rozmieszczone na małym chipie, co pozwala na jednoczesne przesłuchiwanie wielu SNP. Ponieważ allele SNP różnią się tylko jednym nukleotydem i ponieważ trudno jest osiągnąć optymalne warunki hybrydyzacji dla wszystkich sond w macierzy, docelowy DNA ma potencjał do hybrydyzacji z niedopasowanymi sondami. Jest to w pewnym stopniu rozwiązane przez użycie kilku redundantnych sond do przesłuchiwania każdego SNP. Sondy są zaprojektowane tak, aby miały miejsce SNP w kilku różnych lokalizacjach, a także zawierały niedopasowania do allelu SNP. Porównując zróżnicowaną wielkość hybrydyzacji docelowego DNA z każdą z tych zbędnych sond, możliwe jest określenie specyficznych alleli homozygotycznych i heterozygotycznych. Chociaż mikromacierze oligonukleotydowe mają stosunkowo niższą specyficzność i czułość, skala SNP, które można zbadać, jest główną korzyścią. The Affymetrix Human SNP 5.0 GeneChip przeprowadza badanie całego genomu, które może genotypować ponad 500 000 ludzkich SNP (Affymetrix 2007).
Metody oparte na enzymach
Szeroka gama enzymów, w tym ligaza DNA , polimeraza DNA i nukleazy, została wykorzystana do wygenerowania metod genotypowania SNP o wysokiej wierności.
Polimorfizm długości fragmentów restrykcyjnych
Polimorfizm długości fragmentów restrykcyjnych (RFLP) jest uważany za najprostszą i najwcześniejszą metodę wykrywania SNP. SNP-RFLP wykorzystuje wiele różnych endonukleaz restrykcyjnych i ich wysokie powinowactwo do unikalnych i specyficznych miejsc restrykcyjnych. Przeprowadzając trawienie próbki genomowej i określając długość fragmentów za pomocą testu żelowego, można stwierdzić, czy enzymy przecinają oczekiwane miejsca restrykcyjne. Niepowodzenie w cięciu próbki genomowej skutkuje identyfikowalnym większym niż oczekiwano fragmentem, co sugeruje, że w miejscu restrykcyjnym występuje mutacja, która chroni je przed aktywnością nukleazy.
Niestety, połączone czynniki wysokiej złożoności większości genomów eukariotycznych, wymagania dotyczące specyficznych endonukleaz, fakt, że dokładna mutacja niekoniecznie może zostać rozwiązana w jednym eksperymencie oraz powolny charakter testów żelowych sprawiają, że RFLP jest złym wyborem dla wysokiej przepustowości analiza.
Metody oparte na PCR
Refrakcyjny system mutacji amplifikacji tetra-primera lub ARMS-PCR wykorzystuje dwie pary starterów do amplifikacji dwóch alleli w jednej reakcji PCR. Startery są zaprojektowane w taki sposób, że dwie pary starterów zachodzą na siebie w miejscu SNP, ale każdy pasuje idealnie tylko do jednego z możliwych SNP. Podstawą wynalazku jest to, że nieoczekiwanie oligonukleotydy z niedopasowaną resztą 3' nie będą działać jako startery w PCR w odpowiednich warunkach. W rezultacie, jeśli dany allel jest obecny w reakcji PCR, para starterów specyficzna dla tego allelu wytworzy produkt, ale nie dla alternatywnego allelu o innym SNP. Dwie pary starterów są również zaprojektowane w taki sposób, że ich produkty PCR mają znacznie różną długość, co pozwala na łatwe rozróżnienie prążków za pomocą elektroforezy żelowej lub analizy temperatury topnienia. Badając wyniki, jeśli próbka genomowa jest homozygotyczna, produkty PCR, które zostaną uzyskane, będą pochodzić ze startera, który pasuje do lokalizacji SNP i zewnętrznego startera przeciwnej nici, a także z dwóch zewnętrznych starterów. Jeśli próbka genomowa jest heterozygotyczna, wówczas produkty powstaną ze startera każdego allelu i ich odpowiedników ze starterów zewnętrznych, jak również ze starterów zewnętrznych.
Alternatywną strategią jest przeprowadzenie wielu reakcji qPCR z różnymi zestawami starterów, które są ukierunkowane na każdy allel oddzielnie. Dobrze zaprojektowane startery wzmocnią swój docelowy SNP w znacznie wcześniejszym cyklu niż inne SNP. Pozwala to na rozróżnienie więcej niż dwóch alleli, chociaż dla każdego SNP wymagana jest indywidualna reakcja qPCR. Aby osiągnąć wystarczająco wysoką specyficzność, sekwencja startera może wymagać umieszczenia sztucznego niedopasowania w pobliżu jej 3'-końca, co jest podejściem ogólnie znanym jako Taq-MAMA.
Endonukleaza klapowa
Flap endonukleaza (FEN) jest endonukleazą, która katalizuje cięcie specyficzne dla struktury. To rozszczepienie jest bardzo wrażliwe na niedopasowania i może być używane do badania SNP o wysokim stopniu specyficzności
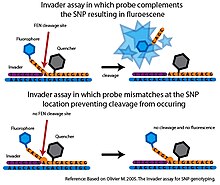
W podstawowym teście Invader, FEN zwany rozszczepieniem łączy się z dwiema specyficznymi sondami oligonukleotydowymi, które razem z docelowym DNA mogą tworzyć trójdzielną strukturę rozpoznawaną przez rozszczepianie. Pierwsza sonda, zwana Invader , jest komplementarna do końca 3' docelowego DNA. Ostatnia zasada Invader jest niedopasowaną zasadą, która zachodzi na nukleotyd SNP w docelowym DNA. Druga sonda jest sondą specyficzną dla allelu, która jest komplementarna do końca 5' docelowego DNA, ale rozciąga się również poza stronę 3' nukleotydu SNP. Sonda specyficzna dla allelu będzie zawierała zasadę komplementarną do nukleotydu SNP. Jeśli docelowy DNA zawiera pożądany allel, sonda Invader i specyficzna dla allelu zwiążą się z docelowym DNA, tworząc trójdzielną strukturę. Ta struktura jest rozpoznawana przez rozszczepienie, które rozszczepi i uwolni koniec 3' sondy specyficznej dla allelu. Jeśli nukleotyd SNP w docelowym DNA nie jest komplementarny do sondy specyficznej dla allelu, nie powstaje prawidłowa struktura trójdzielna i nie następuje cięcie. The najeźdźcy jest zwykle połączony z systemem transferu energii rezonansu fluorescencji (FRET) w celu wykrycia zdarzenia cięcia. W tej konfiguracji cząsteczka wygaszacza jest przyłączona do końca 3', a fluorofor do końca 5' sondy specyficznej dla allelu. Jeśli nastąpi rozszczepienie, fluorofor zostanie oddzielony od cząsteczki wygaszacza, generując wykrywalny sygnał.
W przypadku niedopasowanych sond następuje tylko minimalne rozszczepienie, co sprawia, że test Invader jest wysoce specyficzny. Jednak w oryginalnym formacie tylko jeden allel SNP mógł być badany na próbkę reakcyjną i wymagało to dużej ilości docelowego DNA, aby wygenerować wykrywalny sygnał w rozsądnych ramach czasowych. Kilka zmian rozszerzyło oryginalny Invader . Przeprowadzając wtórne reakcje rozszczepienia FEN, reakcja seryjnego inwazyjnego wzmocnienia sygnału (SISAR) umożliwia zbadanie obu alleli SNP w jednej reakcji. Najeźdźca SISAR wymaga również mniejszej ilości docelowego DNA, poprawiając czułość oryginalnego testu Invader . Test został również dostosowany na kilka sposobów do użytku w formacie o wysokiej przepustowości. Na jednej platformie sondy specyficzne dla alleli są zakotwiczone w mikrosferach. Gdy rozszczepienie przez FEN generuje wykrywalny sygnał fluorescencyjny, sygnał ten mierzy się za pomocą cytometrii przepływowej. Czułość cytometrii przepływowej eliminuje potrzebę amplifikacji PCR docelowego DNA (Rao et al. 2003). Te platformy o dużej przepustowości nie wyszły poza etap sprawdzania zasady i jak dotąd Invader system nie był używany w żadnych projektach genotypowania SNP na dużą skalę.
Przedłużenie podkładu
Wydłużenie startera to proces dwuetapowy, który najpierw obejmuje hybrydyzację sondy z zasadami bezpośrednio przed nukleotydem SNP, a następnie reakcję „minisekwencjonowania”, w której polimeraza DNA wydłuża zhybrydyzowany starter przez dodanie zasady, która jest komplementarna do nukleotyd SNP. Ta wbudowana zasada jest wykrywana i określa allel SNP (Goelet i wsp. 1999; Syvanen 2001). Ponieważ wydłużanie starterów opiera się na bardzo dokładnym enzymie polimerazy DNA, metoda ta jest generalnie bardzo niezawodna. Wydłużenie startera jest w stanie genotypować większość SNP w bardzo podobnych warunkach reakcji, co czyni go również bardzo elastycznym. Metoda wydłużania starterów jest stosowana w wielu formatach testów. Te formaty wykorzystują szeroki zakres technik wykrywania, które obejmują MALDI-TOF Spektrometria masowa (patrz Sequenom ) i metody podobne do ELISA .
Zasadniczo istnieją dwa główne podejścia, które wykorzystują włączenie albo znakowanych fluorescencyjnie dideoksynukleotydów (ddNTP), albo deoksynukleotydów znakowanych fluorescencyjnie (dNTP). W przypadku ddNTP sondy hybrydyzują z docelowym DNA bezpośrednio przed nukleotydem SNP, a pojedynczy ddNTP komplementarny do allelu SNP jest dodawany do końca 3' sondy (brakujący 3'-hydroksyl w didioksynukleotydzie zapobiega dodawaniu kolejnych nukleotydów ). Każdy ddNTP jest znakowany innym sygnałem fluorescencyjnym, co pozwala na wykrycie wszystkich czterech alleli w tej samej reakcji. W przypadku dNTP sondy specyficzne dla alleli mają zasady 3', które są komplementarne do każdego z badanych alleli SNP. Jeśli docelowy DNA zawiera allel komplementarny do zasady 3' sondy, docelowy DNA całkowicie zhybrydyzuje z sondą, umożliwiając polimerazie DNA rozciąganie się od końca 3' sondy. Jest to wykrywane przez włączenie znakowanych fluorescencyjnie dNTP na końcu sondy. Jeśli docelowy DNA nie zawiera allelu komplementarnego do zasady 3' sondy, docelowy DNA wytworzy niedopasowanie na końcu 3' sondy i polimeraza DNA nie będzie mogła wydłużyć się od końca 3' sondy. Zaletą drugiego podejścia jest to, że kilka znakowanych dNTP może zostać włączonych do rosnącej nici, co pozwala na zwiększenie sygnału. Jednak polimeraza DNA w niektórych rzadkich przypadkach może rozciągać się od niedopasowanych sond 3', dając wynik fałszywie dodatni.
Inne podejście stosuje metoda genotypowania iPLEX SNP firmy Sequenom , która wykorzystuje spektrometr masowy MassARRAY. Sondy przedłużające są zaprojektowane w taki sposób, że 40 różnych testów SNP można amplifikować i analizować w koktajlu PCR. Reakcja wydłużania wykorzystuje ddNTP jak powyżej, ale wykrycie allelu SNP zależy od rzeczywistej masy produktu wydłużania, a nie od cząsteczki fluorescencyjnej. Ta metoda jest przeznaczona do przepustowości od niskiej do średniej i nie jest przeznaczona do skanowania całego genomu.
Elastyczność i specyficzność wydłużania starterów sprawiają, że nadaje się on do analizy o wysokiej przepustowości. Sondy wydłużające startery można układać na szkiełkach, co pozwala na jednoczesne genotypowanie wielu SNP. Technologia ta, ogólnie nazywana wydłużaniem starterów w macierzach (APEX), ma kilka zalet w porównaniu z metodami opartymi na różnicowej hybrydyzacji sond. Dla porównania metody APEX mają większą moc dyskryminacyjną niż metody wykorzystujące tę hybrydyzację różnicową, ponieważ często niemożliwe jest uzyskanie optymalnych warunków hybrydyzacji dla tysięcy sond na mikromacierzach DNA (zwykle jest to rozwiązywane przez posiadanie wysoce nadmiarowych sond). Jednak w metodach APEX nie można uzyskać takiej samej gęstości sond, co przekłada się na niższą wydajność na cykl.
Infinium firmy Illumina Incorporated jest przykładem potoku genotypowania całego genomu, który jest oparty na metodzie wydłużania starterów. W teście Infinium można genotypować ponad 100 000 SNP. Test wykorzystuje nukleotydy znakowane haptenem w reakcji wydłużania startera. Znacznik haptenowy jest rozpoznawany przez przeciwciała, które z kolei są sprzężone z wykrywalnym sygnałem (Gunderson i in. 2006).
APEX-2 to metoda genotypowania wydłużania starterów z macierzą, która jest w stanie zidentyfikować setki SNP lub mutacji równolegle przy użyciu wydajnego jednorodnego multipleksowego PCR (do 640-pleksu) i czterokolorowego wydłużania jednozasadowego na mikromacierzy. Multipleksowy PCR wymaga dwóch oligonukleotydów na amplikony generujące SNP/mutacje, które zawierają badaną parę zasad. Te same oligonukleotydy stosuje się w następnym etapie jako immobilizowane startery przedłużające o pojedynczej zasadzie na mikromacierzy (Krjutskov et al. 2008).
5'- nukleaza
Aktywność 5'-nukleazy polimerazy Taq DNA jest wykorzystywana w teście TaqMan do genotypowania SNP. Test TaqMan przeprowadza się równolegle z reakcją PCR, a wyniki można odczytać w czasie rzeczywistym w miarę postępu reakcji PCR (McGuigan & Ralston 2002). Test wymaga starterów PCR do przodu i do tyłu, które będą amplifikować region zawierający miejsce polimorficzne SNP. Rozróżnienie alleli uzyskuje się za pomocą FRET w połączeniu z jedną lub dwiema sondami specyficznymi dla alleli, które hybrydyzują z miejscem polimorficznym SNP. Sondy będą miały fluorofor połączony z ich końcem 5' i cząsteczkę wygaszacza połączoną z końcem 3'. Gdy sonda jest nienaruszona, wygaszacz pozostanie w pobliżu fluoroforu, eliminując sygnał fluoroforu. Podczas etapu amplifikacji PCR, jeśli sonda specyficzna dla allelu jest doskonale komplementarna do allelu SNP, zwiąże się z docelową nicią DNA, a następnie zostanie zdegradowana przez aktywność 5'-nukleazy polimerazy Taq, ponieważ wydłuża DNA z PCR podkłady. Degradacja sondy powoduje oddzielenie fluoroforu od cząsteczki wygaszacza, generując wykrywalny sygnał. Jeśli sonda specyficzna dla allelu nie jest doskonale komplementarna, będzie miała niższą temperaturę topnienia i nie będzie wiązać się tak skutecznie. Zapobiega to oddziaływaniu nukleazy na sondę (McGuigan i Ralston 2002).
Ponieważ test TaqMan jest oparty na PCR, jest stosunkowo prosty do wdrożenia. Test TaqMan można zwielokrotnić, łącząc wykrywanie do siedmiu SNP w jednej reakcji. Jednakże, ponieważ każdy SNP wymaga odrębnej sondy, TaqMan jest ograniczony przez to, jak blisko mogą znajdować się SNP. Skalę testu można drastycznie zwiększyć, przeprowadzając wiele równoczesnych reakcji na płytkach do mikromiareczkowania. Generalnie TaqMan ogranicza się do zastosowań, które obejmują badanie niewielkiej liczby SNP, ponieważ dla każdego SNP należy zaprojektować optymalne sondy i warunki reakcji (Syvanen 2001).
Test ligacji oligonukleotydów
Ligaza DNA katalizuje ligację końca 3' fragmentu DNA z końcem 5' bezpośrednio przylegającego fragmentu DNA. Mechanizm ten można wykorzystać do zbadania SNP przez hybrydyzację dwóch sond bezpośrednio nad miejscem polimorficznym SNP, dzięki czemu może nastąpić ligacja, jeśli sondy są identyczne z docelowym DNA. W teście ligazy oligonukleotydowej zaprojektowano dwie sondy; sondę specyficzną dla allelu, która hybrydyzuje z docelowym DNA, tak że jej zasada 3' znajduje się bezpośrednio nad nukleotydem SNP, oraz drugą sondę, która hybrydyzuje z matrycą w górę (w dół w nici komplementarnej) miejsca polimorficznego SNP, zapewniając koniec 5' do reakcji ligacji. Jeśli sonda specyficzna dla allelu pasuje do docelowego DNA, w pełni hybrydyzuje z docelowym DNA i może nastąpić ligacja. Ligacja na ogół nie zachodzi w obecności niedopasowanej zasady 3'. Zligowane lub nieligowane produkty można wykrywać za pomocą elektroforezy żelowej, spektrometrii masowej MALDI-TOF lub elektroforezy kapilarnej do zastosowań na dużą skalę. Dzięki odpowiednim sekwencjom i znacznikom na oligonukleotydach można wygenerować wysokowydajne dane dotyczące sekwencji z ligowanych produktów i określić genotypy (Curry i in., 2012). Zastosowanie dużej liczby indeksów próbek umożliwia generowanie danych sekwencji o wysokiej przepustowości dla setek SNP w tysiącach próbek w niewielkiej części przebiegu sekwencjonowania o wysokiej przepustowości. Jest to masowe genotypowanie za pomocą technologii sekwencjonowania (MGST). [ potrzebne źródło ]
Inne metody poamplifikacyjne oparte na właściwościach fizycznych DNA
Charakterystyczne właściwości DNA, takie jak temperatura topnienia i konformacja pojedynczej nici, zostały wykorzystane w kilku zastosowaniach do rozróżnienia alleli SNP. Metody te bardzo często osiągają wysoką specyficzność, ale wymagają wysoce zoptymalizowanych warunków, aby uzyskać jak najlepsze wyniki.
Polimorfizm konformacji pojedynczej nici
Jednoniciowy DNA (ssDNA) składa się w strukturę trzeciorzędową. Konformacja jest zależna od sekwencji i większość mutacji pojedynczych par zasad zmienia kształt struktury. Po nałożeniu na żel trzeciorzędowy kształt określi ruchliwość ssDNA, zapewniając mechanizm różnicowania alleli SNP. Ta metoda obejmuje najpierw amplifikację PCR docelowego DNA. Dwuniciowe produkty PCR są denaturowane przy użyciu ciepła i formaldehydu w celu wytworzenia ssDNA. ssDNA nakłada się na niedenaturujący żel do elektroforezy i pozostawia do fałdowania w strukturę trzeciorzędową. Różnice w sekwencji DNA zmienią konformację trzeciorzędową i zostaną wykryte jako różnica w ruchliwości nici ssDNA (Costabile i in. 2006). Metoda ta jest szeroko stosowana, ponieważ jest prosta technicznie, stosunkowo niedroga i wykorzystuje powszechnie dostępny sprzęt. Jednak w porównaniu z innymi metodami genotypowania SNP czułość tego testu jest niższa. Stwierdzono, że konformacja ssDNA jest wysoce zależna od temperatury i ogólnie nie jest jasne, jaka jest idealna temperatura. Bardzo często test będzie prowadzony przy użyciu kilku różnych temperatur. Istnieje również ograniczenie długości fragmentu, ponieważ czułość spada, gdy stosuje się sekwencje dłuższe niż 400 bp (Costabile i in. 2006).
Elektroforeza żelowa w gradiencie temperatury
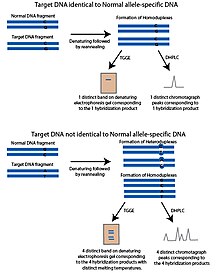
elektroforezy żelowej z gradientem temperatury (TGGE) lub elektroforezy kapilarnej z gradientem temperatury (TGCE) opiera się na zasadzie, że częściowo zdenaturowany DNA jest bardziej ograniczony i porusza się wolniej w materiale porowatym, takim jak żel. Właściwość ta pozwala na rozdział DNA na podstawie temperatury topnienia. Aby dostosować te metody do wykrywania SNP, stosuje się dwa fragmenty; docelowy DNA, który zawiera badane miejsce polimorficzne SNP i sekwencję DNA specyficzną dla allelu, określaną jako normalny fragment DNA. Normalny fragment jest identyczny z docelowym DNA, z wyjątkiem potencjalnego miejsca polimorficznego SNP, które jest nieznane w docelowym DNA. Fragmenty są denaturowane, a następnie ponownie wyżarzane. Jeśli docelowy DNA ma ten sam allel co normalny fragment, utworzą się homodupleksy o tej samej temperaturze topnienia. Podczas pracy na żelu z gradientem temperatury pojawi się tylko jeden prążek. Jeśli docelowy DNA ma odrębny allel, po etapie ponownego wygrzewania utworzą się cztery produkty; homodupleksy składające się z docelowego DNA, homodupleksy składające się z normalnego DNA i dwa heterodupleksy z każdej nici docelowego DNA zhybrydyzowanej z normalną nicią DNA. Te cztery produkty będą miały różne temperatury topnienia i pojawią się jako cztery pasma w denaturującym żelu.
Denaturująca wysokosprawna chromatografia cieczowa
Denaturująca wysokosprawna chromatografia cieczowa (DHPLC) wykorzystuje HPLC z odwróconą fazą przesłuchiwać SNP. Kluczem do DHPLC jest faza stała, która ma zróżnicowane powinowactwo do jedno- i dwuniciowego DNA. W DHPLC fragmenty DNA są denaturowane przez ogrzewanie, a następnie pozostawiane do ponownego połączenia. Temperatura topnienia ponownie połączonych fragmentów DNA określa długość czasu, przez jaki są one przetrzymywane w kolumnie. Za pomocą PCR generowane są dwa fragmenty; docelowy DNA zawierający miejsce polimorficzne SNP i sekwencję DNA specyficzną dla allelu, określaną jako normalny fragment DNA. Ten normalny fragment jest identyczny z docelowym DNA, z wyjątkiem potencjalnego miejsca polimorficznego SNP, które jest nieznane w docelowym DNA. Fragmenty są denaturowane, a następnie pozostawiane do stopniowego ponownego wyżarzania. Ponownie przetworzone produkty dodaje się do kolumny DHPLC. Jeśli allel SNP w docelowym DNA pasuje do normalnego fragmentu DNA, podczas etapu ponownego wygrzewania utworzą się tylko identyczne homodupleksy. Jeśli docelowy DNA zawiera inny allel SNP niż normalny fragment DNA, oprócz homodupleksów utworzą się heterodupleksy docelowego DNA i normalnego DNA zawierające niedopasowane miejsce polimorficzne. Niedopasowane heterodupleksy będą miały inną temperaturę topnienia niż homodupleksy i nie będą zatrzymywane w kolumnie tak długo. Generuje to wzór chromatografu, który różni się od wzoru, który zostałby wygenerowany, gdyby docelowy fragment DNA i normalne fragmenty DNA były identyczne. Eluowany DNA jest wykrywany przez absorpcję UV.
DHPLC można łatwo zautomatyzować, ponieważ nie jest wymagane znakowanie ani oczyszczanie fragmentów DNA. Metoda jest również stosunkowo szybka i charakteryzuje się wysoką specyficznością. Jedną z głównych wad DHPLC jest to, że temperatura kolumny musi być optymalizowana dla każdego celu, aby osiągnąć właściwy stopień denaturacji.
Topienie całego amplikonu w wysokiej rozdzielczości
Analiza topnienia w wysokiej rozdzielczości jest najprostszą do zrozumienia metodą opartą na PCR. Zasadniczo te same właściwości termodynamiczne, które pozwoliły na działanie technik żelowych, mają zastosowanie tutaj iw czasie rzeczywistym. Fluorymetr monitoruje denaturację całego amplikonu dsDNA po PCR. Tworzysz startery specyficzne dla miejsca, które chcesz wzmocnić. „Malujesz” amplikon barwnikiem specyficznym dla podwójnej nici, zawartym w mieszaninie PCR. Specyficzny ds barwnik integruje się z produktem PCR. W istocie cały amplikon staje się sondą. Otwiera to nowe możliwości odkrywania. Albo umieszczasz startery bardzo blisko obu stron danego SNP (genotypowanie małych amplikonów, Liew, 2004) albo amplifikujesz większy region (o długości 100-400 pz) do celów skanowania. W przypadku prostego genotypowania SNP łatwiej jest po prostu zrobić mały amplikon, aby zminimalizować szanse pomylenia jednego SNP z innym. Określa się temperaturę topnienia (Tm) całego amplikonu i większość homozygot wystarczająco różni się (w lepszych instrumentach) Tm od genotypu. Heterozygoty są jeszcze łatwiejsze do rozróżnienia, ponieważ mają wygenerowane heterodupleksy (patrz wyjaśnienia na bazie żelu), co poszerza przejście w stanie stopionym i zwykle daje dwa dostrzegalne piki. Opisano topienie amplikonu przy użyciu startera znakowanego fluorescencyjnie (Gundry i in., 2003), ale jest to mniej praktyczne niż stosowanie barwników specyficznych dla ds ze względu na koszt fluorogennego startera.
Skanowanie większych amplikonów opiera się na tych samych zasadach, co opisane powyżej. Jednak temperatura topnienia i ogólny kształt krzywej topnienia stają się pouczające. W przypadku amplikonów > ok. 150 bp występują często > 2 piki topnienia, z których każdy może się różnić w zależności od składu matrycy DNA. Wielu badaczom udało się z powodzeniem wyeliminować większość ich sekwencjonowania poprzez skanowanie oparte na stopieniu, umożliwiając dokładne genotypowanie dużej liczby osobników w oparciu o locus. Wielu badaczy odkryło, że skanowanie mutacji przy użyciu topnienia w wysokiej rozdzielczości jest realnym i praktycznym sposobem badania całych genów.
Wykorzystanie białek wiążących niedopasowanie DNA
Białka wiążące niedopasowanie DNA mogą rozróżnić niedopasowania pojedynczych nukleotydów, a tym samym ułatwić analizę różnicową SNP. Na przykład białko MutS z Thermus aquaticus wiąże różne niedopasowania pojedynczych nukleotydów z różnymi powinowactwami i może być stosowane w elektroforezie kapilarnej do rozróżnienia wszystkich sześciu zestawów niedopasowań (Drabovich & Krylov 2006).
SNPlex
SNPlex to zastrzeżona platforma do genotypowania sprzedawana przez Applied Biosystems .
Test nukleazy Surveyor
Nukleaza Surveyor jest enzymem endonukleazy niedopasowanych zasad, który rozpoznaje wszystkie podstawienia zasad i małe insercje/delecje (indele) oraz rozszczepia stronę 3 'niedopasowanych miejsc w obu niciach DNA.
Sekwencjonowanie
sekwencjonowania nowej generacji, takie jak pirosekwencjonowanie, sekwencjonują mniej niż 250 zasad w odczycie, co ogranicza ich zdolność do sekwencjonowania całych genomów. Jednak ich zdolność do generowania wyników w czasie rzeczywistym i ich potencjał do masowego skalowania czyni je realną opcją do sekwencjonowania małych regionów w celu przeprowadzenia genotypowania SNP. W porównaniu z innymi metodami genotypowania SNP, sekwencjonowanie jest w szczególności odpowiednie do identyfikacji wielu SNP w małym regionie, takim jak wysoce polimorficzny głównego kompleksu zgodności tkankowej genomu.
Dalsza lektura
- Abravaya K, Huff J, Marshall R, Kupiec B, Mullen C, Schneider G, Robinson J (kwiecień 2003). „Latarnie molekularne jako narzędzia diagnostyczne: technologia i zastosowania”. Chemia kliniczna i medycyna laboratoryjna . 41 (4): 468–474. doi : 10.1515/CCLM.2003.070 . PMID 12747588 . S2CID 36220311 .
- Costabile M, Quach A, Ferrante A (grudzień 2006). „Podejścia molekularne w diagnostyce pierwotnych niedoborów odporności” . Ludzka mutacja . 27 (12): 1163–1173. doi : 10.1002/humu.20412 . PMC 7165860 . PMID 16960849 .
- Drabowicz AP, Kryłow SN (marzec 2006). „Identyfikacja par zasad w polimorfizmie pojedynczego nukleotydu za pomocą elektroforezy kapilarnej za pośrednictwem białka MutS”. Chemia analityczna . 78 (6): 2035–2038. doi : 10.1021/ac0520386 . PMID 16536443 .
- Gundry CN, Vandersteen JG, Reed GH, Pryor RJ, Chen J, Wittwer CT (marzec 2003). „Analiza topnienia amplikonu ze znakowanymi starterami: metoda w zamkniętej probówce do różnicowania homozygot i heterozygot” . Chemia kliniczna . 49 (3): 396–406. doi : 10.1373/49.3.396 . PMID 12600951 .
- Gunderson KL, Steemers FJ, Ren H, Ng P, Zhou L, Tsan C i in. (2006). „Genotypowanie całego genomu”. Metody w enzymologii . 410 : 359–376. doi : 10.1016/S0076-6879(06)10017-8 . ISBN 9780121828158 . PMID 16938560 .
- Liew M, Pryor R, Palais R, Meadows C, Erali M, Lyon E, Wittwer C (lipiec 2004). „Genotypowanie polimorfizmów pojedynczych nukleotydów przez topienie małych amplikonów w wysokiej rozdzielczości” . Chemia kliniczna . 50 (7): 1156–1164. doi : 10.1373/clinchem.2004.032136 . PMID 15229148 .
- McGuigan FE, Ralston SH (wrzesień 2002). „Wykrywanie polimorfizmu pojedynczego nukleotydu: dyskryminacja alleli przy użyciu TaqMan”. Genetyka psychiatryczna . 12 (3): 133–136. doi : 10.1097/00041444-200209000-00003 . PMID 12218656 .
- Syvänen AC (grudzień 2001). „Dostęp do zmienności genetycznej: genotypowanie polimorfizmów pojedynczych nukleotydów”. Recenzje przyrody. Genetyka . 2 (12): 930–942. doi : 10.1038/35103535 . PMID 11733746 . S2CID 15411761 .
Linki zewnętrzne
- Międzynarodowy projekt HapMap
- Delaware Valley Personalized Medicine Project Wykorzystuje SNP, aby uczynić medycynę osobistą
- Genotypowanie za pomocą testu ligacji oligonukleotydów (OLA)
- Dlaczego SNP testuje myszy?