Metylaza adeninowa DNA
| Specyficzne dla | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| miejsca identyfikatory metylotransferazy DNA (specyficzne dla adeniny). | |||||||||
| nr WE | 2.1.1.72 | ||||||||
| nr CAS | 69553-52-2 | ||||||||
| Bazy danych | |||||||||
| IntEnz | Widok IntEnz | ||||||||
| BRENDA | Wpis BRENDY | ||||||||
| ExPASy | Widok NiceZyme | ||||||||
| KEGG | Wpis KEGG | ||||||||
| MetaCyc | szlak metaboliczny | ||||||||
| PRYM | profil | ||||||||
| Struktury PDB | RCSB PDB PDBe PDB suma | ||||||||
| |||||||||
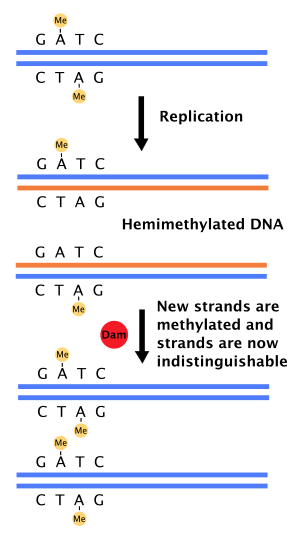
Metylaza adeniny DNA , (metylaza Dam) (również specyficzna dla miejsca metylotransferaza DNA (specyficzna dla adeniny) , EC 2.1.1.72 , metylaza modyfikacji , system modyfikacji restrykcji ) jest enzymem , który dodaje grupę metylową do adeniny o sekwencji 5 „-GATC-3” w nowo zsyntetyzowanym DNA . Bezpośrednio po syntezie DNA nić potomna pozostaje przez krótki czas niemetylowana. Jest to sieroca metylotransferaza, która nie jest częścią systemu modyfikacji restrykcji i reguluje ekspresję genów. Enzym ten katalizuje następującą reakcję chemiczną
- S-adenozylo-L-metionina + adenina DNA S-adenozylo-L-homocysteina + DNA 6-metyloaminopuryna
Jest to duża grupa enzymów unikalnych dla prokariontów i bakteriofagów.
Enzym metylotransferazy DNA E. coli (Dam) jest szeroko stosowany w technice profilowania chromatyny, DamID . W którym Dam jest połączona z interesującym białkiem wiążącym DNA i wyrażana jako transgen w genetycznie możliwym organizmie modelowym w celu identyfikacji miejsc wiązania białka.
Rola w naprawie niedopasowania DNA
Gdy polimeraza DNA popełni błąd powodujący niedopasowanie pary zasad lub małą insercję lub delecję podczas syntezy DNA , komórka naprawi DNA na drodze zwanej naprawą niedopasowania . Jednak komórka musi być w stanie odróżnić nić matrycową od nici nowo zsyntetyzowanej. W niektórych bakteriach nici DNA są metylowane przez metylazę Dam, dlatego bezpośrednio po replikacji DNA będzie hemimetylowane . Enzym naprawczy, MutS , wiąże się z niedopasowaniami w DNA i rekrutuje MutL, który następnie aktywuje endonukleazę MutH. MutH wiąże hemimetylowane miejsca GATC i po aktywacji selektywnie rozszczepia niemetylowaną nić potomną, umożliwiając helikazie i egzonukleazom wycięcie powstającej nici w regionie otaczającym niedopasowanie. Nić jest następnie ponownie syntetyzowana przez polimerazę DNA III .
Rola w regulacji replikacji
Odpalanie początku replikacji (oriC) w komórkach bakteryjnych jest ściśle kontrolowane, aby zapewnić, że replikacja DNA zachodzi tylko raz podczas każdego podziału komórki. Częściowo można to wytłumaczyć powolną hydrolizą ATP przez DnaA, białko, które wiąże się z powtórzeniami w oriC, inicjując replikację. Metylaza kobieca również odgrywa pewną rolę, ponieważ oriC ma 11 sekwencji 5'-GATC-3' (w E. coli ). Natychmiast po replikacji DNA oriC jest hemimetylowany i sekwestrowany przez pewien czas. Dopiero po tym oriC zostaje uwolniony i musi zostać w pełni zmetylowany przez metylazę Dam, zanim nastąpi wiązanie DnaA.
Rola w regulacji ekspresji białek
Dam odgrywa również rolę w promocji i represji transkrypcji RNA . W E. coli dalsze sekwencje GATC są metylowane , co sprzyja transkrypcji. Na przykład zmiana fazy pilusów związanych z odmiedniczkowym zapaleniem nerek (PAP) w uropatogennych E. coli jest kontrolowana przez Dam poprzez metylację dwóch miejsc GATC proksymalnie i dystalnie od promotora PAP . Biorąc pod uwagę jego rolę w regulacji białek w E. coli , gen metylazy Dam jest nieistotny, ponieważ nokaut genu nadal pozostawia bakterie żywe. Zachowanie żywotności pomimo nokautu matki obserwuje się również u Salmonelli i Aggregatibacter actinomycetemcomitans . Jednak w organizmach takich jak Vibrio cholerae i Yersinia pseudotuberculosis gen damy jest niezbędny do przeżycia. Nokaut dam w Aggregatibacter actinomycetemcomitans spowodował rozregulowanie poziomu białka, leukotoksyny, a także zmniejszył zdolność drobnoustroju do inwazji komórek nabłonka jamy ustnej. Dodatkowo badanie nad Streptococcus mutans z niedoborem metylazy Dam , patogenem dentystycznym, ujawniło dysregulację 103 genów, z których niektóre obejmują potencjał próchnicotwórczy.
Cechy konstrukcyjne
Podobieństwo domen katalitycznych metylotransferaz C5-cytozyny oraz metylotransferaz N6 i N4-adeniny wzbudziło duże zainteresowanie w zrozumieniu podstaw podobieństw i różnic funkcjonalnych. Metylotransferazy lub metylazy są podzielone na trzy grupy (grupy α, β i γ) w oparciu o kolejność określonych 9 motywów i domenę rozpoznawania celu (TRD). Motyw I składa się z tripeptydu Gly-X-Gly i jest określany jako pętla G i bierze udział w wiązaniu kofaktora S-adenozylometioniny . Motif II jest wysoce konserwatywny wśród metalaz N4 i N6-adeninowych i zawiera ujemnie naładowany aminokwas, po którym następuje hydrofobowy łańcuch boczny w ostatnich pozycjach nici β2 do wiązania AdoMet . Motyw III jest również zamieszany w wiązanie Adometu. Motyw IV jest szczególnie ważny i dobrze znany w charakteryzowaniu metylazy. Składa się ze składnika dirolilowego i jest wysoce konserwatywny wśród metylotransferaz N6-adeninowych jako motyw DPPY, jednak motyw ten może się różnić dla metylotransferaz N4-adeninowych i C5-cytozynowych. Stwierdzono, że motyw DPPY jest niezbędny do wiązania AdoMet. Motywy IV-VIII odgrywają rolę w aktywności katalitycznej, podczas gdy motywy 1-III i X odgrywają rolę w wiązaniu kofaktora. W przypadku metylaz N6-adeninowych kolejność tych motywów jest następująca: N-końcowy - X - I - II - III - TRD - IV - V - VI - VII - VIII - C-końcowy i E . coli Metylaza Dam jest zgodna z tą sekwencją strukturalną. Eksperyment krystalograficzny z 2015 roku wykazał, że E. coli Dam methylase była zdolna do wiązania DNA innego niż GATC z tą samą sekwencją omawianych motywów; autorzy uważają, że uzyskana struktura może służyć jako podstawa do represji transkrypcji, która nie jest oparta na metylacji.
Sieroce melazy bakteryjne i bakteriofagowe
Metylaza dam jest sierocą metylotransferazą, która nie jest częścią systemu modyfikacji restrykcji, ale działa niezależnie, regulując ekspresję genów, naprawę niedopasowań i replikację bakterii wśród wielu innych funkcji. Nie jest to jedyny przykład metylotransferazy sierocej, ponieważ istnieje metylotransferaza regulowana cyklem komórkowym (CcrM), która metyluje 5'-GANTC-'3 hemi-metylowany DNA w celu kontrolowania cyklu życiowego Caulobacter crescentus i innych pokrewnych gatunków.
W odróżnieniu od swoich bakteryjnych odpowiedników, fagowe sieroce metylotransferazy również istnieją, a przede wszystkim w bakteriofagach T2, T4 i innych T-parzystych, które infekują E. coli. W badaniu stwierdzono, że pomimo wspólnej homologii sekwencji, sekwencje aminokwasowe metylazy E. coli i T4 Dam mają identyczność sekwencji do 64% w czterech regionach o długości od 11 do 33 reszt, co sugeruje wspólne ewolucyjne pochodzenie bakterii i geny metylazy fagowej. Metylazy T2 i T4 różnią się od E. coli nie tylko zdolnością do metylacji 5-hydroksymetylocytozyny, ale także do metylacji niekanonicznych miejsc DNA. Pomimo obszernej in vitro tych kilku wybranych metylotransferaz fagowych sierocych, ich biologiczne przeznaczenie wciąż nie jest jasne.