Retrowirusowa rybonukleaza H
| Retrowirusowa rybonukleaza H | |||||||||
|---|---|---|---|---|---|---|---|---|---|
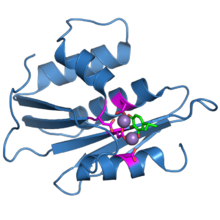 Domena rybonukleazy H z białka odwrotnej transkryptazy HIV-1. Cztery reszty karboksylanowe miejsca aktywnego są pokazane w kolorze magenta. Dwa związane jony manganu są pokazane jako fioletowe kulki. Związana cząsteczka inhibitora, beta-tujaplicynol, jest zaznaczona na zielono.
| |||||||||
| Identyfikatory | |||||||||
| nr WE | 3.1.26.13 | ||||||||
| nr CAS | 9050-76-4 | ||||||||
| Bazy danych | |||||||||
| IntEnz | Widok IntEnz | ||||||||
| BRENDA | Wpis BRENDY | ||||||||
| ExPASy | Widok NiceZyme | ||||||||
| KEGG | Wpis KEGG | ||||||||
| MetaCyc | szlak metaboliczny | ||||||||
| PRYM | profil | ||||||||
| Struktury PDB | RCSB PDB PDBe PDB suma | ||||||||
| |||||||||
Retrowirusowa ( rybonukleaza H (retrowirusowa RNaza H) jest domeną katalityczną enzymu retrowirusowej odwrotnej transkryptazy RT). Enzym RT jest używany do generowania komplementarnego DNA ( cDNA ) z genomu retrowirusowego RNA . Ten proces nazywa się odwrotną transkrypcją . Aby zakończyć ten złożony proces, retrowirusowe enzymy RT muszą przyjąć wielofunkcyjną naturę. Dlatego posiadają 3 z następujących biochemicznych : polimerazy DNA zależnej od RNA , rybonukleazy H i polimerazy DNA zależnej od DNA . Podobnie jak wszystkie enzymy RNazy H, domena retrowirusowej RNazy H rozszczepia dupleksy DNA/RNA i nie degraduje DNA ani niezhybrydyzowanego RNA.
Struktura

Chociaż struktury RT ludzkich , mysich i ptasich retrowirusów wykazują różne podjednostki, względne rozmiary, orientacja i połączenie domen polimerazy DNA i RNazy H są uderzająco podobne. Domena RNazy H zajmuje ~ 25% C-końca białka RT . Domena polimerazy DNA zajmuje ~ 55% N-końca białka RT. Domeny RNazy H MMLV i HIV-1 RT są strukturalnie bardzo podobne do Escherichia coli i Bacillus halodurans RNazy H, jak również ludzką RNazę H1 . Na ogół pofałdowane struktury domen retrowirusowej RNazy H mają postać 5-niciowych mieszanych arkuszy beta otoczonych czterema helisami alfa w rozkładzie asymetrycznym. Godną uwagi różnicą między różnymi białkami RNazy H jest obecność lub brak C-helisy (obecnej w E. coli, MLV i ludzkich RNazach H, nieobecnej w HIV-1, B. halodurany i ASLV RNazy H), dodatnio naładowanej helisa alfa zwana również podstawową pętlą lub występem. Uważa się, że odgrywa rolę w wiązaniu substratu.

Funkcjonować
Podczas odwrotnej transkrypcji wirusowego genomowego RNA do cDNA powstaje hybryda RNA/DNA. Nić RNA jest następnie hydrolizowana przez domenę RNazy H, aby umożliwić syntezę drugiej nici DNA przez funkcję polimerazy DNA enzymu RT. Ponadto wiriony retrowirusowe pakują pojedynczą cząsteczkę tRNA , której używają jako startera podczas odwrotnej transkrypcji wirusowego genomowego RNA. Retrowirusowa RNaza H jest potrzebna do strawienia cząsteczki tRNA, gdy nie jest już potrzebna. Procesy te zachodzą w sposób zależny od Mg2+.
Retrowirusowe RNazy H rozszczepiają swoje substraty na 3 różne sposoby:
- specyficzne dla sekwencji wewnętrzne cięcie RNA [1-4]. Enzymy ludzkiego wirusa niedoboru odporności typu 1 i mysiego wirusa białaczki Moloney'a preferują rozszczepianie nici RNA o jeden nukleotyd od połączenia RNA-DNA.
- Skierowane na 5'-końcu RNA cięcie 13-19 nukleotydów od końca RNA.
- Skierowane na 3'-koniec DNA cięcie 15-20 nukleotydów od końca startera.
Dwa tryby skierowane na koniec są unikalne dla retrowirusowych RNaz H ze względu na szereg efektów związanej z domeną polimerazy retrowirusowej RT. W bardziej uniwersalnym trybie rozszczepiania wewnętrznego, RNazy H zachowują się jak typowe endonukleazy i rozszczepiają RNA wzdłuż hybrydowego substratu DNA/RNA przy braku jakichkolwiek efektów końcowych.
Linki zewnętrzne
- Retrowirus + rybonukleaza + H w US National Library of Medicine Medical Subject Headings (MeSH)
