Icerudiwirus

|
|
| Icerudivirus | |
|---|---|
| Rudiviridae virion | |
| Klasyfikacja wirusów | |
| (nierankingowe): | Wirus |
| królestwo : | Adnaviria |
| Królestwo: | Zilligvirae |
| Gromada: | Taleaviricota |
| Klasa: | Tokiviricetes |
| Zamówienie: | więzadła wirusowe |
| Rodzina: | Rudiviridae |
| Rodzaj: | Icerudiwirus |
Icerudivirus to rodzaj wirusów z rodziny Rudiviridae . Wirusy te są bezotoczkowymi wirusami w kształcie sztywnych pręcików z liniowymi genomami dsDNA, które infekują hipertermofilne archeony z gatunku Sulfolobus islandicus . W rodzaju występują trzy gatunki.
Taksonomia
Następujące gatunki są przypisane do rodzaju:
- Icerudivirus SIRV1 , nazwa zwyczajowa Sulfolobus islandicus pałeczkowaty wirus 1 (SIRV1)
- Icerudivirus SIRV2 , nazwa zwyczajowa Sulfolobus islandicus pręcikowaty wirus 2 (SIRV2)
- Icerudivirus SIRV3
Icerudivirus był wcześniej nazwany Rudivirus , ale został przemianowany w 2020 roku.
Odkrycie
SIRV1 i SIRV2 były wytwarzane przez sklonowane kolonie szczepów Sulfolobus islandicus . Te dwa szczepy zostały wyizolowane z próbek pobranych w 1994 roku z różnych pól solfatarycznych w Islandii , Kverkfjöll i Hveragerði , które są oddalone od siebie o 250 km. Te islandzkie kwaśne gorące źródła solfatarowe osiągają temperaturę 88°C i pH 2,5.
Struktura
Wiriony są bezotoczkowe i składają się z podobnej do rurki superhelisy utworzonej przez dsDNA i główne białko strukturalne, z czopami na każdym końcu, do których zakotwiczone są trzy włókna ogonka. Wydaje się, że te włókna ogonka biorą udział w adsorpcji na powierzchni komórki gospodarza i są tworzone przez jedno z mniejszych białek strukturalnych. [ potrzebne źródło ]
SIRV1 i SIRV2 to sztywne pręciki o szerokości około 23 nm, ale różniące się długością — SIRV1 ma około 830 nm, a SIRV2 około 900 nm. Przedstawiają kanał centralny o długości ok. 6 nm, która otacza genom DNA. Na każdym końcu pręta znajduje się korek o długości ok. 48 nm długości i 6 nm średnicy, która wypełnia końcową część wnęki, wraz z trzema włóknami ogonka o długości ok. długości 28 nm. [ potrzebne źródło ]
Trójwymiarową rekonstrukcję wirionu SIRV2 w rozdzielczości ~ 4 angstremów uzyskano za pomocą mikroskopii krioelektronowej. Struktura ujawniła wcześniej nieznaną formę organizacji wirionu, w której alfa-helikalne główne białko kapsydu SIRV2 owija się wokół DNA, czyniąc je niedostępnym dla rozpuszczalnika. Stwierdzono, że wirusowe DNA jest całkowicie w formie A, co sugeruje wspólny mechanizm ochrony DNA w najbardziej niesprzyjających środowiskach z przetrwalnikami bakteryjnymi.
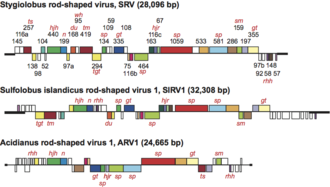
Genom
Genom składa się z liniowego dsDNA i waha się od 24 kb (ARV1) do 35 kb (SIRV2). Dwie nici liniowych genomów są połączone kowalencyjnie, a na obu końcach genomu znajdują się odwrócone końcowe powtórzenia. Sulfolobus z odwróconymi końcowymi powtórzeniami o długości 2029 pz na końcach liniowego genomu. Zawartość G+C w obu genomach jest niezwykle niska i wynosi zaledwie 25%, podczas gdy genom Sulfolobus solfataricus (sekwencjonowany genom najbliższy gospodarzowi wirusa) osiąga 37%.
Chociaż sekwencje odwróconych powtórzeń końcowych rudiwirusów są różne, wszystkie one niosą motyw AATTTAGGAATTTAGGAATTT w pobliżu końców genomu, co może stanowić sygnał dla resolwazy złącza Hollidaya i replikacji DNA .
Wzorce transkrypcyjne i regulacja transkrypcji
Wzory transkrypcji SIRV1 i SIRV2 są stosunkowo proste, z kilkoma różnicami w ekspresji czasowej. W przeciwieństwie do tego, przewidywano, że co najmniej 10% jego genów ma różne motywy wiążące DNA w kodowanych przez nie białkach i przypisano je jako domniemane regulatory transkrypcji . Wysoki odsetek genów wirusowych kodujących białka wiążące DNA z motywami wiążącymi DNA wstęga-helisa-helisa (RHH) Zaproponowano. Obfitość genów kodujących białka należące do nadrodziny RHH obecnych w genomach crenarchaea i ich wirusów może podkreślać ważną rolę tych białek w regulacji transkrypcji genów gospodarza i wirusów w trudnych warunkach.
Białko SvtR było pierwszym szczegółowo scharakteryzowanym regulatorem RHH crenarchae, a także pierwszymi wirusowymi regulatorami transkrypcji w domenie Archaeal . Silnie hamuje transkrypcję mniejszego białka strukturalnego i, w mniejszym stopniu, własnego genu. Struktura jest bardzo podobna do bakteryjnych białek RHH pomimo niskiego podobieństwa sekwencji, takich jak CopG, bakteryjny regulator liczby kopii plazmidu.
Wykazano również, że aktywator transkrypcji kodowany przez Sulfolobus islandicus, Sta1, aktywuje transkrypcję kilku genów wirusowych .
Cykl życiowy wirusa
SIRV2 rozpoznaje swojego gospodarza przez wiązanie się z pilusami typu 4 obficie obecnymi na powierzchni komórki. Wirus początkowo wiąże się z czubkiem pilusa, a następnie przesuwa się wzdłuż pilusa na powierzchnię komórki, gdzie wirion rozkłada się, a genom SIRV2 jest internalizowany przez nieznany mechanizm. SIRV2 jest litycznym , który zabija komórkę gospodarza w wyniku opracowanych mechanizmów zaaranżowanych przez wirusa. Masowa degradacja chromosomów gospodarza następuje z powodu infekcji wirusowej, a w cytoplazmie zachodzi składanie wirionów . Wiriony są uwalniane z komórki gospodarza poprzez mechanizm, który obejmuje tworzenie specyficznych struktur komórkowych.
Potencjalne zastosowania w nanotechnologii
SIRV2 może działać jako szablon do selektywnej względem miejsca i kontrolowanej przestrzennie modyfikacji chemicznej. Zarówno końce, jak i ciało wirusa, lub tylko końce, mogą być adresowane chemicznie, dlatego SIRV2 można uznać za strukturalnie unikalny blok nanobudulcowy.