Reduktaza rybonukleotydowa
| reduktaza rybonukleozydodifosforanowa | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 reduktaza rybonukleozydodifosforanowa heterooktamer,
| |||||||||
| identyfikatory E.Coli | |||||||||
| nr WE | 1.17.4.1 | ||||||||
| nr CAS | 9047-64-7sy | ||||||||
| Bazy danych | |||||||||
| IntEnz | Widok IntEnz | ||||||||
| BRENDA | Wpis BRENDY | ||||||||
| ExPASy | Widok NiceZyme | ||||||||
| KEGG | Wpis KEGG | ||||||||
| MetaCyc | szlak metaboliczny | ||||||||
| PRYM | profil | ||||||||
| Struktury PDB | RCSB PDB PDBe PDB suma | ||||||||
| Ontologia genów | AmiGO / QuickGO | ||||||||
| |||||||||
Reduktaza rybonukleotydowa ( RNR ), znana również jako reduktaza difosforanowa rybonukleozydu ( rNDP ), jest enzymem , który katalizuje tworzenie dezoksyrybonukleotydów z rybonukleotydów . Katalizuje to tworzenie, usuwając grupę 2'-hydroksylową z pierścienia rybozy difosforanów nukleozydów. Ta redukcja wytwarza dezoksyrybonukleotydy. Z kolei dezoksyrybonukleotydy są wykorzystywane w syntezie DNA . Reakcja katalizowana przez RNR jest ściśle konserwatywna we wszystkich żywych organizmach. Ponadto RNR odgrywa kluczową rolę w regulowaniu całkowitej szybkości syntezy DNA, dzięki czemu stosunek DNA do masy komórkowej jest utrzymywany na stałym poziomie podczas podziału komórki i naprawy DNA . Dość niezwykłą cechą enzymu RNR jest to, że katalizuje reakcję przebiegającą poprzez mechanizm działania wolnych rodników . Substratami dla RNR są ADP , GDP , CDP i UDP . dTDP (difosforan dezoksytymidyny) jest syntetyzowany przez inny enzym ( kinaza tymidylanowa ) z dTMP (monofosforan dezoksytymidyny).
Struktura
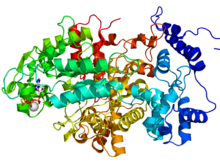
Reduktazy rybonukleotydowe dzielą się na trzy klasy. Enzymy RNR klasy I są zbudowane z dużej podjednostki alfa i małych podjednostek beta, które łączą się, tworząc aktywny heterodimeryczny tetramer . Redukując NDP do 2'-dNDP, enzym katalizuje syntezę de novo dezoksyrybonukleotydów (dNTP), które są prekursorami syntezy DNA i są niezbędne do proliferacji komórek . RNR klasy II wytwarzają rodnik 5'-dezoksyadenozylowy przez homolityczne rozszczepienie wiązania C-Co w adenozylokobalaminie. Ponadto RNR klasy III zawierają stabilny rodnik glicylowy.
Ludzie są nosicielami RNR klasy I. Podjednostka alfa jest kodowana przez gen RRM1, podczas gdy istnieją dwie izoformy podjednostki beta, kodowane przez geny RRM2 i RRM2B:
|
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
monomer alfa klasy I składa się z trzech domen :
- jedna domena głównie helikalna zawierająca 220 N-końcowych reszt,
- drugą dużą dziesięcioniciową strukturę α/β zawierającą 480 reszt,
- i trzecią małą pięcioniciową strukturę α/β zawierającą 70 reszt.
W Pfam druga domena została zinterpretowana jako dwie oddzielne domeny:
- krótsza całkowicie alfa N-końcowa domena,
- i dłuższą beczkową domenę C-końcową.
|
|
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Podjednostka beta klasy I zwykle zawiera centrum dimetaliczne i stabilny rodnik tyrozylowy . U ludzi podjednostka beta opiera się na kofaktorze diżelaza. W E. coli rodnik tyrozylowy znajduje się w pozycji 122 (Y122), zapewniając stabilny rodnik dla podjednostek RNR2 klasy I. W Aegypti , ten rodnik tyrozylowy znajduje się w pozycji 184 (Y184). Rodnik tyrozylowy jest głęboko zakopany w białku w środowisku hydrofobowym, zlokalizowanym blisko centrum żelaza, które jest wykorzystywane do stabilizacji rodnika tyrozylowego. Struktura dwóch μ-okso-połączonych żelaz jest zdominowana przez ligandy, które służą jako miejsca wiązania żelaza: cztery karboksylany [ asparaginian (D146), glutaminian (E177, E240 i E274)] oraz dwie histydyny (H180 i H277). Powiązanie występuje między C-końcem RNR2 a C-końcem z RNR1. Aktywność enzymatyczna zależy od połączenia podjednostek RNR1 i RNR2. Miejsce aktywne składa się z aktywnych grup ditiolowych z RNR1 oraz centrum diżelazowego i rodnika tyrozylowego z podjednostki RNR2.
Inne reszty RNR2, takie jak asparaginian (D273), tryptofan (W48) i tyrozyna (Y356) dodatkowo stabilizują rodnik tyrozylowy w miejscu aktywnym, umożliwiając w ten sposób przenoszenie elektronów. Reszty te pomagają w przeniesieniu elektronu rodnikowego z tyrozyny (Y122) RNR2 do cysteiny (C439) RNR1. Transfer elektronów rozpoczyna się na tyrozynie RNR2 (Y122) i trwa w RNR2 do tryptofanu (W48), który jest oddalony od tyrozyny RNR1 (Y731) o 2,5 nanometra . Transfer elektronów z RNR2 do RNR1 zachodzi przez tyrozynę (Y356 do Y731) i dalej przez tyrozynę (Y730) do cysteiny (C439) w miejscu aktywnym. Ukierunkowane mutacje struktury pierwszorzędowej RNR wskazują, że wszystkie cytowane powyżej reszty uczestniczą w przenoszeniu wolnych rodników na duże odległości do miejsca aktywnego.
U komarów A. aegypti RNR1 zachowuje większość kluczowych reszt aminokwasowych, w tym asparaginian (D64) i walinę (V292 lub V284), które są niezbędne w regulacji allosterycznej ; reszty proliny (P210 i P610), leucyny (L453 i L473) i metioniny (M603), które znajdują się w hydrofobowym miejscu aktywnym; reszty cysteiny (C225, C436 i C451), które biorą udział w usuwaniu atomu wodoru i przenoszeniu elektronu rodnikowego w miejscu aktywnym; cysteina (C225 i C436), asparagina (N434) i reszty glutaminianu (E441), które wiążą substrat rybonukleotydowy; reszty tyrozyny (Y723 i Y743), które dyktują przeniesienie rodników; i reszty cysteiny (C838 i C841), które są stosowane w regeneracji grup ditiolowych w miejscu aktywnym.
Funkcjonować
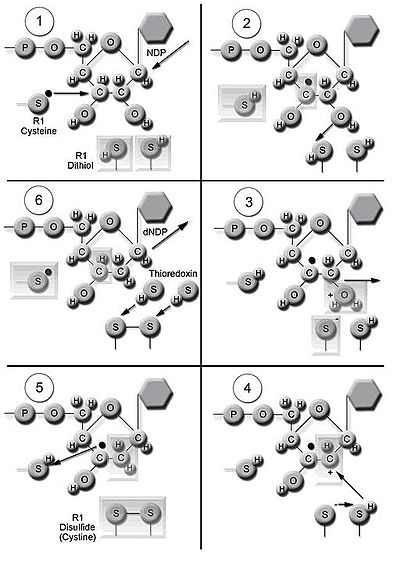
Enzym reduktaza rybonukleotydowa (RNR) katalizuje syntezę de novo dNDP. Kataliza 5'-difosforanów rybonukleozydu (NDP) obejmuje redukcję przy węglu 2' 5-fosforanu rybozy z wytworzeniem 5'-difosforanów 2'-dezoksyrybonukleozydu o zredukowanej pochodnej 2'-dezoksyrybonukleozydu (dNDP). Redukcja ta rozpoczyna się wraz z wytworzeniem wolnego rodnika. Po pojedynczej redukcji RNR wymaga elektronów przekazanych z grup ditiolowych białka tioredoksyny . Regeneracja tioredoksyny zachodzi , gdy fosforan dinukleotydu nikotynoamidoadeninowego ( NADPH ) dostarcza dwa atomy wodoru , które są wykorzystywane do zmniejszenia grupy dwusiarczkowe tioredoksyny.
Trzy klasy RNR mają podobne mechanizmy redukcji NDP, ale różnią się domeną generującą wolne rodniki, specyficznym metalem w strukturze metaloproteiny i donorami elektronów. Wszystkie zajęcia wykorzystują chemię wolnorodnikową. Reduktazy klasy I wykorzystują centrum żelaza z konwersją żelaza do żelaza w celu wytworzenia wolnego rodnika tyrozylowego. Redukcja substratów NDP zachodzi w warunkach tlenowych. Reduktazy klasy I dzielą się na IA i IB ze względu na różnice w regulacji. Reduktazy klasy IA występują u eukariontów , eubakterii , bakteriofagów i wirusy . Reduktazy klasy IB znajdują się w eubakteriach. Reduktazy klasy IB mogą również wykorzystywać rodnik generowany podczas stabilizacji dwujądrowego manganu . Reduktazy klasy II wytwarzają wolny rodnik 5'-deoksyadenozylowy z kobalaminy (koenzym B12) i mają prostszą budowę niż reduktazy klasy I i klasy III. Redukcja NDP lub 5'-trifosforanów rybonukleotydów (NTP) zachodzi w warunkach tlenowych lub beztlenowych. Reduktazy klasy II są rozmieszczone w archebakteriach , eubakterie i bakteriofagi. Reduktazy klasy III wykorzystują rodnik glicyny generowany za pomocą S-adenozylometioniny i centrum siarki żelaza. Redukcja NTP jest ograniczona do warunków beztlenowych. Reduktazy klasy III są rozmieszczone w archebakteriach, eubakteriach i bakteriofagach. Organizmy nie są ograniczone do posiadania jednej klasy enzymów. Na przykład E. coli ma RNR zarówno klasy I, jak i klasy III.
Mechanizm redukcji katalitycznej
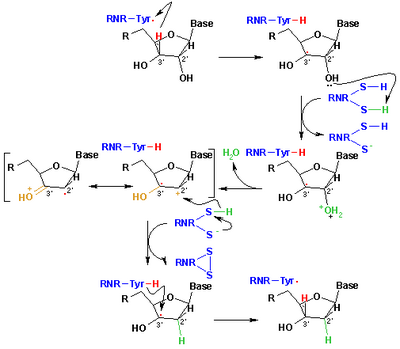
Mechanizm, który jest obecnie akceptowany dla redukcji rybonukleotydów do dezoksyrybonukleotydów, przedstawiono na poniższym schemacie. Pierwszy etap obejmuje abstrakcję 3'-H substratu 1 przez rodnik Cys439. Następnie reakcja polega na eliminacji jednej cząsteczki wody z węgla C-2' rybonukleotydu, katalizowanej przez Cys225 i Glu441. W trzecim etapie następuje przeniesienie atomu wodoru z Cys225 na węgiel C-2' rodnika 2'-ketylowego 3, po uprzednim przeniesieniu protonu z Cys462 na Cys225. Pod koniec tego etapu otrzymuje się rodnikowy anionowy mostek dwusiarczkowy i ketonowy związek pośredni 4 o zamkniętej powłoce. Ten związek pośredni został zidentyfikowany podczas konwersji kilku 2'-podstawionych analogów substratów, jak również z naturalnym substratem oddziałującym z mutantami enzymów. Kolejnym etapem jest utlenienie anionowego mostka dwusiarczkowego, z jednoczesną redukcją substratu, generując 5. Gęstość spinów przesuwa się od atomów siarki do atomu C-3' substratu, z jednoczesnym przeniesieniem protonu z Glu441 na węgiel C -3'. Ostatni etap jest odwrotnością pierwszego etapu i obejmuje przeniesienie wodoru z Cys439 do C-3', regenerację początkowego rodnika i uzyskanie końcowego produktu 6.
Teoretyczne modele niektórych etapów tych mechanizmów z wykorzystaniem pełnego modelu białka R1 można znaleźć w badaniach przeprowadzonych przez Cerqueira i in. .
Rozporządzenie
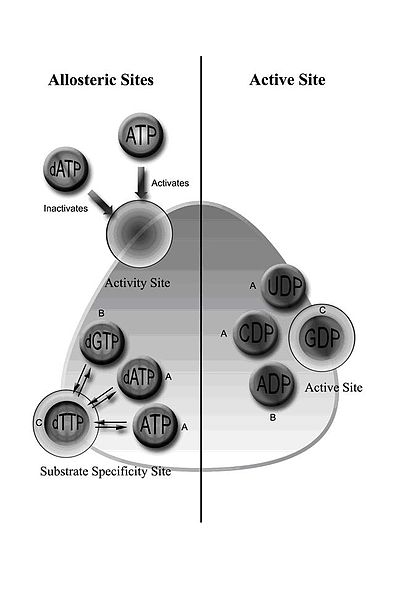
RNR klasy I obejmuje podjednostki RNR1 i RNR2, które mogą łączyć się, tworząc heterodimeryczny tetramer. RNR1 zawiera oba miejsca allosteryczne, pośredniczące w regulacji specyficzności i aktywności substratu. W zależności od konfiguracji allosterycznej, jeden z czterech rybonukleotydów wiąże się z miejscem aktywnym.
Regulacja RNR ma na celu utrzymanie zrównoważonych ilości dNTP. Wiązanie cząsteczek efektorowych zwiększa lub zmniejsza aktywność RNR. Kiedy ATP wiąże się z miejscem aktywności allosterycznej, aktywuje RNR. W przeciwieństwie do tego, gdy dATP wiąże się z tym miejscem, dezaktywuje RNR. Oprócz kontrolowania aktywności, mechanizm allosteryczny reguluje również specyficzność substratu i zapewnia, że enzym wytwarza równe ilości każdego dNTP do syntezy DNA. We wszystkich klasach wiązanie ATP lub dATP z miejscem allosterycznym indukuje redukcję 5'-difosforanu cytydyny (CDP) i 5'-difosforanu urydyny (UDP); 2'-deoksyguanozyno-5'-trifosforan (dGTP) indukuje redukcję adenozyno-5'-difosforanu (ADP); a 5'-trifosforan 2'-dezoksytymidyny (dTTP) indukuje redukcję 5'-difosforanu guanozyny (GDP) (ryc. 1).
Reduktazy klasy IB nie są hamowane przez dATP, ponieważ brakuje im około 50 N-końcowych aminokwasów wymaganych do allosterycznej aktywności. Ponadto ważne jest, aby aktywność reduktazy rybonukleotydowej znajdowała się pod kontrolą transkrypcyjną i potranskrypcyjną, ponieważ synteza wolnego od uszkodzeń DNA opiera się na zrównoważonej puli dezoksyrybonukleotydów. Komórki eukariotyczne z reduktazami klasy IA mają mechanizm kontroli negatywnej, który wyłącza syntezę dNTP w miarę ich gromadzenia. Mechanizm ten chroni komórkę przed toksycznymi i mutagennymi efektami, które mogą wynikać z nadprodukcji dNTP, ponieważ zmiany w zrównoważonych pulach dNTP prowadzą do uszkodzenia DNA i śmierci komórki. Chociaż nadprodukcja dNTP lub niezrównoważona ich podaż może prowadzić do błędnego włączenia nukleotydów do DNA, podaż dNTP może pozwolić na naprawę DNA. p53R2 jest małą podjednostką reduktazy rybonukleotydowej, która może indukować taką naprawę. Zmiany w tym indukowanym przez p53 homologu R2 mogą powodować wyczerpanie mitochondrialnego DNA iw konsekwencji p53R2 służy jako główny czynnik w dostarczaniu dNTP.
RNR może wykorzystywać morfeeinowy model regulacji allosterycznej .
Inhibitory RNR1 i RNR2
Ogólnie inhibitory RNR klasy I można podzielić na trzy główne grupy: inhibitory translacji, które blokują syntezę enzymu; inhibitory dimeryzacji, które zapobiegają asocjacji dwóch podjednostek RNR (R1 i R2); oraz inhibitory katalityczne, które inaktywują podjednostkę R1 i/lub podjednostkę R2.
RNR klasy I może być hamowany przez peptydy podobne do C-końca RNR2. Peptydy te mogą konkurować z RNR2 o wiązanie z RNR1, w wyniku czego RNR1 nie tworzy enzymatycznie aktywnego kompleksu z RNR2. Chociaż C-koniec białek RNR2 jest różny u różnych gatunków, RNR2 może wchodzić w interakcje z RNR1 u różnych gatunków. Gdy mysi C-koniec RNR2 został zastąpiony C-końcowymi (7 lub 33) resztami aminokwasowymi E. coli RNR2, chimeryczna podjednostka RNR2 nadal wiąże się z mysimi podjednostkami RNR1. Brakuje im jednak aktywności enzymatycznej, prawdopodobnie ze względu na eliminację reszt biorących udział w przenoszeniu elektronu wolnorodnikowego z podjednostki RNR2 na podjednostkę RNR1.
Małe peptydy mogą specyficznie hamować wiązanie podjednostek RNR2 z RNR1, gdy mają one znaczące podobieństwo do normalnego C-końca RNR2. To hamowanie wiązania RNR2 z RNR1 zostało pomyślnie przetestowane w RNR wirusa opryszczki pospolitej (HSV). Gdy oligomer składający się z 7 aminokwasów (GAVVNDL) obcięty z C-końca podjednostki RNR2 został użyty w testach kompetycyjnych, uniemożliwił on normalnemu RNR2 tworzenie aktywnego enzymatycznie kompleksu z RNR1. Inne małe inhibitory peptydowe podobne do C-końca RNR2 były również z powodzeniem stosowane do hamowania aktywności enzymatycznej RNR HSV, a tym samym replikacji HSV. W mysich modelach zapalenia rogówki zrębu i neowaskularyzacji rogówki ( choroba oka HSV ), doniesiono, że mały C-końcowy analog RNR2 BILD 1263 hamuje RNR i jest skuteczny w zapobieganiu tym chorobom. W niektórych przypadkach, chociaż leczenie małymi analogami C-końcowymi może nie zatrzymać rozprzestrzeniania się choroby, nadal mogą one pomóc w leczeniu. W acyklowirze oporny HSV (PAAr5), doniesiono, że mały inhibitor peptydowy BILD 1633 jest od 5 do 10 razy silniejszy niż BILD 1263 przeciwko skórnej infekcji PAAr5. Terapia skojarzona (BILD 1633 i acyklowir) jest skuteczniejsza w leczeniu miejscowych zmian chorobowych u myszy. Dane te sugerują, że małe inhibitory peptydowe, które konkurują z RNR2 o wiązanie z RNR1, są przydatne w zapobieganiu rozprzestrzenianiu się HSV.
Gal hamuje RNR2, zastępując Fe 3+ w miejscu aktywnym. Maltolan galu jest biodostępną po podaniu doustnym formą galu, która wykorzystuje tę hamującą aktywność do leczenia raka, infekcji i innych chorób.
Leki hydroksymocznik i Motexafin gadolin zakłócają działanie tego enzymu.
Linki zewnętrzne
- Rybonukleotydy + reduktazy w US National Library of Medicine Medical Subject Headings (MeSH)
- Baza danych reduktazy rybonukleotydowej (RNRDb)