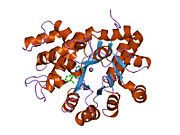
Deaminaza adenozynowa
| ADA | |||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| Identyfikatory | |||||||||||||||||||||||||||||||||||||||||||||||
| , entrez:100, deaminaza adenozyny, | |||||||||||||||||||||||||||||||||||||||||||||||
| identyfikatory zewnętrzne ADA1 | |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| Wikidane | |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| deaminazy adenozyny/AMP deaminazy | |||||||||
|---|---|---|---|---|---|---|---|---|---|
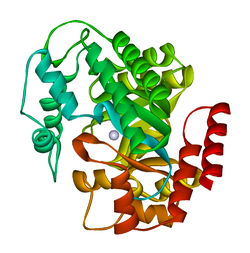
 adenozyny Plasmodium yoelii (py02076)
| |||||||||
| Identyfikatory | |||||||||
| Symbol | A_deaminaza | ||||||||
| Pfam | PF00962 | ||||||||
| Klan Pfam | CL0034 | ||||||||
| InterPro | IPR001365 | ||||||||
| PROZYTA | PDOC00419 | ||||||||
| SCOP2 | 1dodaj / ZAKRES / SUPFAM | ||||||||
| CDD | cd01320 | ||||||||
| |||||||||
| Identyfikatory | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| domeny deaminazy adenozyny (edytazy). | |||||||||
| Symbol | A_deamin | ||||||||
| Pfam | PF02137 | ||||||||
| InterPro | IPR002466 | ||||||||
| PROZYTA | PDOC00419 | ||||||||
| SCOP2 | 1dodaj / ZAKRES / SUPFAM | ||||||||
| |||||||||
| identyfikatory | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| deaminazy adenozyny/AMP | |||||||||
| Symbol | A_deaminaza_N | ||||||||
| Pfam | PF08451 | ||||||||
| InterPro | IPR013659 | ||||||||
| |||||||||
Deaminaza adenozyny (znana również jako aminohydrolaza adenozyny lub ADA ) jest enzymem ( EC 3.5.4.4 ) zaangażowanym w metabolizm puryn . Jest potrzebna do rozkładu adenozyny z pożywienia i do obrotu kwasów nukleinowych w tkankach.
Jego podstawową funkcją u ludzi jest rozwój i utrzymanie układu odpornościowego. Jednak pełna fizjologiczna rola ADA nie jest jeszcze w pełni poznana.
Struktura
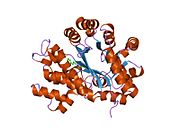
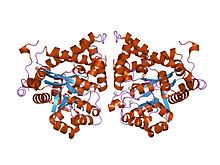
ADA występuje zarówno w małej postaci (jako monomer), jak iw dużej postaci (jako kompleks dimeru). W postaci monomeru enzym jest łańcuchem polipeptydowym złożonym w osiem pasm równoległych beczek α/β, które otaczają centralną głęboką kieszeń, która jest miejscem aktywnym. Oprócz ośmiu centralnych beczek β i ośmiu peryferyjnych helis α , ADA zawiera również pięć dodatkowych helis: reszty 19-76-krotne w trzech helisach, zlokalizowane między fałdami β1 i α1; a dwie antyrównoległe helisy na końcu karboksylowym znajdują się w poprzek końca aminowego beczki β.
Centrum aktywne ADA zawiera jon cynku, który znajduje się w najgłębszym zagłębieniu miejsca aktywnego i jest koordynowany przez pięć atomów z His15, His17, His214, Asp295 i podłoża. Cynk jest jedynym niezbędnym do działania kofaktorem .
Substrat, adenozyna, jest stabilizowany i związany z miejscem aktywnym dziewięcioma wiązaniami wodorowymi. Grupa karboksylowa Glu217, z grubsza współpłaszczyznowa z pierścieniem purynowym substratu, jest w stanie utworzyć wiązanie wodorowe z N1 substratu. Grupa karboksylowa Asp296, również współpłaszczyznowa z pierścieniem purynowym substratu, tworzy wiązanie wodorowe z N7 substratu. Grupa NH Gly184 jest w stanie utworzyć wiązanie wodorowe z N3 substratu. Asp296 tworzy wiązania zarówno z jonem Zn 2+ , jak iz 6-OH podłoża. His238 również wiąże wodór z substratem 6-OH. 3'-OH rybozy substratu tworzy wiązanie wodorowe z Asp19, podczas gdy 5'-OH tworzy wiązanie wodorowe z His17. Dwa dalsze wiązania wodorowe są tworzone w cząsteczkach wody, przy otwarciu miejsca aktywnego, przez 2'-OH i 3'-OH substratu.
Ze względu na cofnięcie się miejsca aktywnego do wnętrza enzymu, po związaniu substrat jest prawie całkowicie odseparowany od rozpuszczalnika. Ekspozycja powierzchniowa podłoża na działanie rozpuszczalnika po związaniu wynosi 0,5% ekspozycji powierzchniowej podłoża w stanie wolnym.
Reakcje
ADA nieodwracalnie dezaminuje adenozynę, przekształcając ją w pokrewną inozynę nukleozydową poprzez zastąpienie grupy aminowej grupą ketonową.
Inozyna może być następnie derybozylowana (usunięta z rybozy ) przez inny enzym zwany fosforylazą nukleozydów purynowych (PNP), przekształcając ją w hipoksantynę .
Mechanizm katalizy
Proponowany mechanizm deaminacji katalizowanej przez ADA to stereospecyficzna addycja-eliminacja przez tetraedryczny związek pośredni. W obu mechanizmach Zn 2+ jako silny elektrofil aktywuje cząsteczkę wody, która jest deprotonowana przez podstawowy Asp295, tworząc atakujący wodorotlenek. His238 orientuje cząsteczkę wody i stabilizuje ładunek atakującego wodorotlenku. Glu217 jest protonowany, aby przekazać proton N1 substratu.
Reakcja jest stereospecyficzna ze względu na położenie reszt cynku, Asp295 i His238, które wszystkie są skierowane w stronę B pierścienia purynowego substratu.
Kompetycyjne hamowanie zaobserwowano w przypadku ADA, gdzie produkt inozyny działa na kompetycyjny inhibitor aktywności enzymatycznej.
Funkcjonować
ADA jest uważana za jeden z kluczowych enzymów metabolizmu puryn. Enzym został znaleziony w bakteriach, roślinach, bezkręgowcach, kręgowcach i ssakach, z wysokim zachowaniem sekwencji aminokwasów . Wysoki stopień zachowania sekwencji aminokwasów sugeruje kluczową naturę ADA w szlaku odzyskiwania puryn.
Przede wszystkim ADA u ludzi bierze udział w rozwoju i utrzymaniu układu odpornościowego. Jednak związek ADA zaobserwowano również z różnicowaniem komórek nabłonka , neuroprzekaźnictwem i utrzymaniem ciąży . Sugerowano również, że ADA, oprócz rozpadu adenozyny, stymuluje uwalnianie aminokwasów pobudzających i jest niezbędna do sprzęgania receptorów adenozynowych A1 i heterotrimerycznych białek G. Niedobór deaminazy adenozyny prowadzi do zwłóknienia płuc, co sugeruje, że przewlekła ekspozycja na wysokie poziomy adenozyny może raczej zaostrzać reakcje zapalne niż je tłumić. Stwierdzono również, że białko i aktywność deaminazy adenozynowej są regulowane w górę w sercach myszy z nadekspresją HIF-1α , co częściowo wyjaśnia obniżone poziomy adenozyny w sercach wykazujących ekspresję HIF-1α podczas stresu niedokrwiennego .
Patologia
Niektóre mutacje w genie deaminazy adenozyny powodują, że nie ulega ona ekspresji. Wynikający z tego niedobór jest jedną z przyczyn ciężkiego złożonego niedoboru odporności (SCID), zwłaszcza dziedziczenia autosomalnego recesywnego. Niedobór ADA jest również związany z zapaleniem płuc, śmiercią komórek grasicy i wadliwą sygnalizacją receptora komórek T.
I odwrotnie, mutacje powodujące nadekspresję tego enzymu są jedną z przyczyn niedokrwistości hemolitycznej .
Istnieją pewne dowody na to, że inny allel (ADA2) może prowadzić do autyzmu .
Podwyższony poziom ADA jest również związany z AIDS .
izoformy
Istnieją 2 izoformy ADA: ADA1 i ADA2.
- ADA1 znajduje się w większości komórek ciała, zwłaszcza w limfocytach i makrofagach , gdzie jest obecna nie tylko w cytozolu i jądrze, ale także w postaci ektoformy na błonie komórkowej przyłączonej do peptydazy dipeptydylowej-4 (inaczej CD26). ADA1 bierze udział głównie w aktywności wewnątrzkomórkowej i występuje zarówno w postaci małej (monomer), jak i dużej (dimer). Wzajemna konwersja form małych do dużych jest regulowana przez „czynnik konwersji” w płucach.
- ADA2 został po raz pierwszy zidentyfikowany w ludzkiej śledzionie. Następnie znaleziono go w innych tkankach, w tym w makrofagach, gdzie współistnieje z ADA1. Te dwie izoformy regulują stosunek adenozyny do deoksyadenozyny, wzmacniając zabijanie pasożytów. ADA2 znajduje się głównie w ludzkim osoczu i surowicy i występuje wyłącznie jako homodimer.
Znaczenie kliniczne
ADA2 jest dominującą formą obecną w ludzkim osoczu krwi , a jej stężenie wzrasta w wielu chorobach, szczególnie tych związanych z układem odpornościowym: na przykład reumatoidalnym zapaleniu stawów , łuszczycy i sarkoidozie . Izoforma ADA2 w osoczu jest również zwiększona w większości nowotworów. ADA2 nie jest wszechobecna, ale współistnieje z ADA1 tylko w monocytach-makrofagach. [ potrzebne źródło ]
Całkowite stężenie ADA w osoczu można mierzyć za pomocą wysokosprawnej chromatografii cieczowej lub technik enzymatycznych lub kolorymetrycznych. Być może najprostszym systemem jest pomiar amoniaku uwalnianego z adenozyny po rozbiciu na inozynę. Po inkubacji osocza z buforowanym roztworem adenozyny amoniak poddaje się reakcji z odczynnikiem Berthelota w celu uzyskania niebieskiego zabarwienia, które jest proporcjonalne do aktywności enzymu. Aby zmierzyć ADA2, erytro-9-(2-hydroksy-3-nonylo)adeninę (EHNA), aby zahamować aktywność enzymatyczną ADA1. To brak ADA1 powoduje SCID .
ADA można również stosować w badaniu limfocytarnego wysięku opłucnowego lub wodobrzusza otrzewnej , ponieważ takie próbki z niskimi poziomami ADA zasadniczo wykluczają gruźlicę.
gruźlicy można teraz dokładnie zdiagnozować na podstawie zwiększonego poziomu deaminazy adenozyny w płynie opłucnowym, powyżej 40 jednostek na litr.
Kladrybina i pentostatyna to leki przeciwnowotworowe stosowane w leczeniu białaczki włochatokomórkowej ; ich mechanizm działania polega na hamowaniu deaminazy adenozynowej.
Zobacz też
Dalsza lektura
- da Cunha JG (1992). „[Deaminaza adenozyny. Enzym pluridyscyplinarny]”. Acta Médica Portuguesa . 4 (6): 315–23. PMID 1807098 .
- Franco R, Casadó V, Ciruela F, Saura C, Mallol J, Canela EI, Lluis C (lipiec 1997). „Deaminaza adenozyny na powierzchni komórki: znacznie więcej niż ektoenzym”. Postęp w neurobiologii . 52 (4): 283–94. doi : 10.1016/S0301-0082(97)00013-0 . PMID 9247966 . S2CID 40318396 .
- Valenzuela A, Blanco J, Callebaut C, Jacotot E, Lluis C, Hovanessian AG, Franco R (1997). „Gp120 otoczki wirusa HIV-1 i cząsteczki wirusa blokują wiązanie deaminazy adenozyny z ludzkim CD26” . Postępy w medycynie eksperymentalnej i biologii . 421 : 185–92. doi : 10.1007/978-1-4757-9613-1_24 . ISBN 978-1-4757-9615-5 . PMID 9330696 .
- Moriwaki Y, Yamamoto T, Higashino K (październik 1999). „Enzymy zaangażowane w metabolizm puryn - przegląd lokalizacji histochemicznej i implikacji funkcjonalnych”. Histologia i histopatologia . 14 (4): 1321–40. PMID 10506947 .
- Hirschhorn R. (1993). „Identyfikacja dwóch nowych mutacji zmiany sensu (R156C i S291L) u dwóch pacjentów z ADA-SCID nietypowych dla odpowiedzi na terapię częściowymi transfuzjami wymiennymi”. Ludzka mutacja . 1 (2): 166–8. doi : 10.1002/humu.1380010214 . PMID 1284479 . S2CID 44617309 .
- Berkvens TM, van Ormondt H, Gerritsen EJ, Khan PM, van der Eb AJ (sierpień 1990). „Identyczna delecja 3250 pz między dwoma powtórzeniami AluI w genach ADA niespokrewnionych pacjentów z ADA-SCID”. Genomika . 7 (4): 486–90. doi : 10.1016/0888-7543(90)90190-6 . PMID 1696926 .
- Aran JM, Colomer D, Matutes E, Vives-Corrons JL, Franco R (sierpień 1991). „Obecność deaminazy adenozynowej na powierzchni jednojądrzastych komórek krwi: lokalizacja immunochemiczna za pomocą mikroskopii świetlnej i elektronowej” . The Journal of Histochemistry and Cytochemistry . 39 (8): 1001–8. doi : 10.1177/39.8.1856451 . PMID 1856451 .
- Bielat K, Tritsch GL (kwiecień 1989). „Aktywność ektoenzymatyczna deaminazy adenozyny ludzkich erytrocytów”. Biochemia molekularna i komórkowa . 86 (2): 135–42. doi : 10.1007/BF00222613 . PMID 2770711 . S2CID 20850552 .
- Hirschhorn R, Tzall S, Ellenbogen A, Orkin SH (luty 1989). „Identyfikacja mutacji punktowej powodującej termolabilną deaminazę adenozynową (ADA) u dwóch niespokrewnionych dzieci z częściowym niedoborem ADA” . Dziennik badań klinicznych . 83 (2): 497–501. doi : 10.1172/JCI113909 . PMC 303706 . PMID 2783588 .
- Murray JL, Perez-Soler R, Bywaters D, Hersh EM (styczeń 1986). „Zmniejszona aktywność deaminazy adenozyny (ADA) i 5'nukleotydazy (5NT) w limfocytach T krwi obwodowej w chorobie Hodgkina”. American Journal of Hematology . 21 (1): 57–66. doi : 10.1002/ajh.2830210108 . PMID 3010705 . S2CID 25540139 .
- Wiginton DA, Kaplan DJ, Stany JC, Akeson AL, Perme CM, Bilyk IJ, Vaughn AJ, Lattier DL, Hutton JJ (grudzień 1986). „Pełna sekwencja i struktura genu ludzkiej deaminazy adenozynowej”. Biochemia . 25 (25): 8234–44. doi : 10.1021/bi00373a017 . PMID 3028473 .
- Akeson AL, Wiginton DA, Dusing MR, Zjednoczone JC, Hutton JJ (listopad 1988). „Zmutowane ludzkie allele deaminazy adenozynowej i ich ekspresja przez transfekcję do fibroblastów” . Journal of Biological Chemistry . 263 (31): 16291-6. doi : 10.1016/S0021-9258(18)37591-4 . PMID 3182793 .
- Glader BE, Backer K (luty 1988). „Podwyższona aktywność deaminazy adenozyny w krwinkach czerwonych: marker zaburzonej erytropoezy w niedokrwistości Diamonda-Blackfana i innych chorobach hematologicznych”. Brytyjski Dziennik Hematologii . 68 (2): 165–8. doi : 10.1111/j.1365-2141.1988.tb06184.x . PMID 3348976 . S2CID 44789636 .
- Petersen MB, Tranebjaerg L, Tommerup N, Nygaard P, Edwards H (luty 1987). „Nowe przypisanie locus genu deaminazy adenozyny do chromosomu 20q13 X 11 w badaniu pacjenta z delecją śródmiąższową 20q” . Dziennik genetyki medycznej . 24 (2): 93–6. doi : 10.1136/jmg.24.2.93 . PMC 1049896 . PMID 3560174 .
- Orkin SH, Goff SC, Kelley WN, Daddona PE (kwiecień 1985). „Przejściowa ekspresja cDNA ludzkiej deaminazy adenozynowej: identyfikacja niefunkcjonalnego klonu wynikającego z podstawienia pojedynczego aminokwasu” . Biologia molekularna i komórkowa . 5 (4): 762–7. doi : 10.1128/mcb.5.4.762 . PMC 366780 . PMID 3838797 .
- Valerio D, Duyvesteyn MG, Dekker BM, Weeda G, Berkvens TM, van der Voorn L, van Ormondt H, van der Eb AJ (luty 1985). „Deaminaza adenozyny: charakterystyka i ekspresja genu z niezwykłym promotorem” . Dziennik EMBO . 4 (2): 437–43. doi : 10.1002/j.1460-2075.1985.tb03648.x . PMC 554205 . PMID 3839456 .
- Bonthron DT, Markham AF, Ginsburg D, Orkin SH (sierpień 1985). „Identyfikacja mutacji punktowej w genie deaminazy adenozynowej odpowiedzialnej za niedobór odporności” . Dziennik badań klinicznych . 76 (2): 894–7. doi : 10.1172/JCI112050 . PMC 423929 . PMID 3839802 .
- Daddona PE, Shewach DS, Kelley WN, Argos P, Markham AF, Orkin SH (październik 1984). „Ludzka deaminaza adenozynowa. cDNA i kompletna sekwencja pierwszorzędowych aminokwasów” . Journal of Biological Chemistry . 259 (19): 12101–6. doi : 10.1016/S0021-9258(20)71325-6 . PMID 6090454 .
- Valerio D, Duyvesteyn MG, Meera Khan P, Geurts van Kessel A, de Waard A, van der Eb AJ (listopad 1983). „Izolacja klonów cDNA dla ludzkiej deaminazy adenozynowej”. gen . 25 (2–3): 231–40. doi : 10.1016/0378-1119(83)90227-5 . PMID 6198240 .
Linki zewnętrzne
- ADA w przeglądarce genomu UCSC .
- ADA w przeglądarce genomu UCSC .
- PDBe-KB zawiera przegląd wszystkich informacji o strukturze dostępnych w PDB dla ludzkiej deaminazy adenozynowej
- PDBe-KB zawiera przegląd wszystkich informacji o strukturze dostępnych w PDB dla mysiej deaminazy adenozynowej