Sygnatury mutacyjne
Sygnatury mutacji to charakterystyczne kombinacje typów mutacji wynikających z określonych procesów mutagenezy , takich jak nieścisłość replikacji DNA , ekspozycja na egzogenne i endogenne genotoksyny , wadliwe ścieżki naprawy DNA i edycja enzymatyczna DNA.
Termin ten jest używany dla dwóch różnych pojęć, często mylonych: sygnatur mutagenów i sygnatur guzów. Jego pierwotne użycie, sygnatura mutagenu, odnosiło się do wzorca mutacji wykonanych w laboratorium przez znany mutagen, a nie przez inne mutageny - unikalny dla mutagenu, tak jak ludzki podpis jest unikalny dla sygnatariusza. Wyjątkowość umożliwia wywnioskowanie mutagenu na podstawie mutacji komórki. Później wyrażenie to odnosiło się do wzorca mutacji charakterystycznych dla typu guza, chociaż zwykle nie jest unikalne dla typu guza ani mutagenu. Jeśli sygnatura mutacji guza pasuje do unikalnej sygnatury mutacji mutagenu, ważne jest wydedukowanie ekspozycji na czynnik rakotwórczy lub procesu mutagenezy, który miał miejsce w odległej przeszłości pacjenta. Coraz bardziej wyrafinowane sygnatury guzów stają się możliwe do przypisania do sygnatur mutagenów.
Rozszyfrowanie sygnatur mutacji w raku zapewnia wgląd w mechanizmy biologiczne zaangażowane w karcynogenezę i normalną mutagenezę somatyczną . Sygnatury mutacji wykazały ich przydatność w leczeniu raka i profilaktyce raka. Postępy w dziedzinie onkogenomiki umożliwiły rozwój i stosowanie terapii ukierunkowanych molekularnie , ale takie terapie historycznie koncentrowały się na hamowaniu czynników onkogennych ( np . rak jelita grubego ). Niedawno profilowanie sygnatur mutacji okazało się skuteczne w kierowaniu postępowaniem onkologicznym i stosowaniem terapii celowanych (np. immunoterapia w naprawie niedopasowanych zasad w różnych typach nowotworów, platyna i inhibitor PARP w celu wykorzystania syntetycznej śmiertelności w raku piersi z niedoborem rekombinacji homologicznej ).
Pojęcia ogólne

Mechanizmy – przegląd
Biologiczne mechanizmy mutagenezy leżące u podstaw sygnatur mutacji (np. sygnatury COSMIC od 1 do 30) obejmują między innymi:
-
Niewierność replikacji DNA
- Korekta DNA to proces, w którym polimeraza DNA wycina nieprawidłowo włączony nukleotyd poprzez reakcję enzymatyczną egzonukleazy . Niezdolność polimerazy DNA do korygowania tych błędów replikacji prowadzi do stopniowej akumulacji mutacji w wyniku kolejnych mitoz komórek .
-
Genotoksyny
- Endogenne mutacje komórkowe (np. spontaniczna deaminacja 5-metylocytozyny prowadzi do przejścia C>T (genetyka) ) (patrz uszkodzenie DNA (występujące naturalnie) )
- Egzogenne/ rakotwórcze
- ultrafioletowe : promieniowanie UVB powoduje bezpośrednie uszkodzenie DNA i jest znanym czynnikiem ryzyka raka skóry (np. czerniaka )
- Alkilujące środki przeciwnowotworowe : ta grupa chemioterapeutyków dodaje grupę alkilową do DNA, co powoduje sieciowanie DNA i zakłóca replikację DNA i naprawę DNA . Komórki rakowe są najbardziej dotknięte ze względu na ich wysoki wskaźnik mitozy .
- Tytoń : Tytoń zawiera kilka czynników rakotwórczych , które są szkodliwe dla DNA, w tym wielopierścieniowe węglowodory aromatyczne , akroleinę , nitrozoaminy , cyjanki i inne (patrz wpływ tytoniu na zdrowie )
-
Niedobór naprawy DNA
- rekombinacji homologicznej (HRD): pęknięcie podwójnej nici DNA wymaga mechanizmu rekombinacji homologicznej w celu dokładnej naprawy punktów przerwania.
- naprawy niedopasowania DNA (MMR): mechanizm naprawy niedopasowania rozpoznaje i naprawia błędne wstawienie, usunięcie lub nieprawidłowe włączenie par zasad.
-
Enzymatyczna edycja DNA
- Enzymy deaminazy cytydynowej: ta rodzina enzymów jest częścią wrodzonego układu odpornościowego i bierze udział w kontroli retrowirusów i elementów transpozonów (w tym endogennych retrowirusów ). Enzymy te ( deaminaza cytydyny / CDA, deaminaza cytydyny indukowana aktywacją i rodzina białek APOBEC ) aktywnie powodują deaminację cytydyny , a tym samym wprowadzają mutacje przejścia C>T (genetyczne) .
-
Niewierność replikacji DNA
Dane genomowe
Analizy sygnatur mutacji raka wymagają danych genomowych z sekwencjonowania genomu raka z sekwencjonowaniem sparowanego normalnego DNA w celu stworzenia katalogu mutacji guza (typy mutacji i liczby) określonego guza. Różne rodzaje mutacji (np. warianty pojedynczych nukleotydów, indele, warianty strukturalne) mogą być stosowane pojedynczo lub w połączeniu do modelowania sygnatur mutacji w raku.
Rodzaje mutacji: podstawienia zasad
Istnieje sześć klas podstawienia zasad: C>A, C>G, C>T, T>A, T>C, T>G. Substytucja G>T jest uważana za równoważną substytucji C>A, ponieważ nie jest możliwe rozróżnienie, w której nici DNA (przód czy tył) substytucja wystąpiła początkowo. Zarówno podstawienia C>A, jak i G>T są zatem liczone jako część klasy „C>A”. Z tego samego powodu mutacje G>C, G>A, A>T, A>G i A>C są liczone jako część „C>G”, „C>T”, „T>A”, „ klasy T>C” i „T>G”.
Pobranie informacji z sąsiednich zasad 5' i 3' (zwanych także flankującymi parami zasad lub kontekstem trinukleotydowym) prowadzi do 96 możliwych typów mutacji (np. A[C>A]A, A[C>A]T, itp.). Katalog mutacji guza jest tworzony poprzez kategoryzowanie każdego wariantu pojedynczego nukleotydu (SNV) (synonimy: podstawienie pary zasad lub mutacja punktowa podstawienia ) w jednym z 96 typów mutacji i zliczenie całkowitej liczby podstawień dla każdego z tych 96 typów mutacji (patrz rysunek).
Katalog mutacji nowotworowych

Po uzyskaniu katalogu mutacji (np. liczby dla każdego z 96 typów mutacji) guza, istnieją dwa podejścia do rozszyfrowania wkładu różnych sygnatur mutacji w krajobraz genomowy guza:
- Katalog mutacji guza jest porównywany z referencyjnym katalogiem mutacji lub referencyjnym zbiorem danych sygnatur mutacji, takim jak 21 Signatures of Mutation Processes in Human Cancer z bazy danych Catalog of Somatic Mutation In Cancer (COSMIC ) .
- de novo można przeprowadzić przy użyciu metod statystycznych, takich jak nieujemna faktoryzacja macierzy, w celu zidentyfikowania potencjalnych nowych procesów mutacyjnych.
Zidentyfikowanie wkładu różnych sygnatur mutacji w karcynogenezę zapewnia wgląd w biologię nowotworów i może zaoferować możliwości terapii celowanej .
Rodzaje mutacji: indele
Sygnatura 3, obserwowana w guzie z niedoborem rekombinacji homologicznej (HR), jest związana ze zwiększonym obciążeniem dużymi indelami (do 50 nukleotydów) z nakładającą się mikrohomologią w punktach przerwania. W takich nowotworach pęknięcia dwuniciowe DNA są naprawiane przez nieprecyzyjne mechanizmy naprawcze łączenia niehomologicznych końców (NHEJ) lub łączenia końców za pośrednictwem mikrohomologii (MMEJ) zamiast naprawy HR o wysokiej wierności.
Sygnatura 6, obserwowana w guzach z niestabilnością mikrosatelitarną , również zawiera wzbogacenie indeli o 1 pz w regionach powtórzeń nukleotydów.
Rodzaje mutacji: warianty strukturalne
rekombinacji homologicznej prowadzi do wzorca podstawienia Signature 3, ale także do zwiększenia obciążenia wariantami strukturalnymi. W przypadku braku rekombinacji homologicznej łączenie niehomologicznych końców prowadzi do dużych wariantów strukturalnych, takich jak translokacje chromosomów , inwersje chromosomów i warianty liczby kopii .
Sygnatury mutacyjne
Krótki opis wybranych procesów mutacyjnych i związanych z nimi sygnatur mutacyjnych w raku zostanie zawarty w poniższych sekcjach. Niektóre sygnatury są wszechobecne w różnych raka (np. sygnatura 1), podczas gdy inne mają tendencję do kojarzenia się z określonymi nowotworami (np. sygnatura 9 i nowotwory układu limfatycznego) .
Niektóre sygnatury mutacji charakteryzują się silnym odchyleniem transkrypcyjnym z substytucjami preferencyjnie wpływającymi na jedną z nici DNA, transkrybowaną lub nietranskrybowaną nić (sygnatury 5, 7, 8, 10, 12, 16).
Podpis 1 charakteryzuje się przewagą przejścia C>T (genetyka) w kontekstach trinukleotydowych Np[C>T]G i koreluje z wiekiem pacjenta w momencie rozpoznania raka . Podstawowym proponowanym mechanizmem biologicznym jest spontaniczna deaminacja 5-metylocytozyny .
Sygnatura 5 ma przewagę podstawień T>C w kontekście trinukleotydu ApTpN z tendencją do transkrypcji nici.
Niedobór rekombinacji homologicznej
Signature 3 wykazuje wysoką liczbę mutacji w wielu klasach mutacji i jest powiązany z mutacjami zarodkowymi i somatycznymi (biologicznymi) BRCA1 i BRCA2 w kilku typach raka (np. piersi, trzustki, jajnika, prostaty). Ta sygnatura wynika z niedoboru naprawy pęknięć dwuniciowych DNA (lub niedoboru rekombinacji homologicznej ). Sygnatura 3 wiąże się z dużym obciążeniem indeli z mikrohomologią w punktach przerwania.
Enzymy APOBEC
Rodzina enzymów deaminazy cytydynowej APOBEC3 odpowiada na infekcje wirusowe poprzez edycję genomu wirusowego, ale stwierdzono również, że aktywność enzymatyczna APOBEC3A i APOBEC3B powoduje niepożądaną edycję genomu gospodarza, a nawet może uczestniczyć w onkogenezie w nowotworach związanych z wirusem brodawczaka ludzkiego .
Signature 2 i Signature 13 są wzbogacone o podstawienia C>T i C>G i uważa się, że wynikają z aktywności deaminazy cytydynowej rodziny enzymów AID/ APOBEC .
Polimorfizm delecji linii zarodkowej obejmujący APOBEC3A i APOBEC3B jest związany z dużym obciążeniem mutacjami Signature 2 i Signature 13. Uważa się, że ten polimorfizm ma umiarkowaną penetrację (dwukrotnie przewyższa ryzyko tła) dla ryzyka raka piersi. Dokładne role i mechanizmy leżące u podstaw APOBEC nie zostały jeszcze w pełni określone, ale uważa się , że indukowana aktywacją deaminaza cytydynowa (AID) / kompleks APOBEC bierze udział w odpowiedzi immunologicznej gospodarza na infekcje wirusowe i metabolizmie lipidów.
Zarówno Signature 2, jak i Signature 13 charakteryzują się podstawieniami cytozyny na uracyl z powodu deaminaz cytydynowych. Signature 2 ma wyższy odsetek podstawień C[T>C]N, a Signature 13 wyższy odsetek podstawień T[C>G]N. APOBEC3A i APOBEC3B preferencyjnie obejmuje opóźnioną nić DNA podczas replikacji.
Niedobór naprawy niedopasowania
Cztery sygnatury mutacji COSMIC zostały powiązane z niedoborem naprawy niedopasowania DNA i znalezione w guzach z niestabilnością mikrosatelitarną : Sygnatura 6, 15, 20 i 26. Utrata funkcji genów MLH1 , MSH2 , MSH6 lub PMS2 powoduje wadliwą naprawę niedopasowania DNA .
Korekta DNA
Sygnatura 10 ma skłonność do transkrypcji i jest wzbogacona o podstawienia C>A w kontekście TpCpT, jak również podstawienia T>G w kontekście TpTpTp. Sygnatura 10 jest związana ze zmienioną funkcją polimerazy DNA epsilon , co skutkuje niedostateczną aktywnością korekty DNA . Zarówno mutacje domeny egzonukleazy linii germinalnej, jak i somatycznej POLE (genu) są związane z sygnaturą 10.
Naprawa z wycięciem podstawy
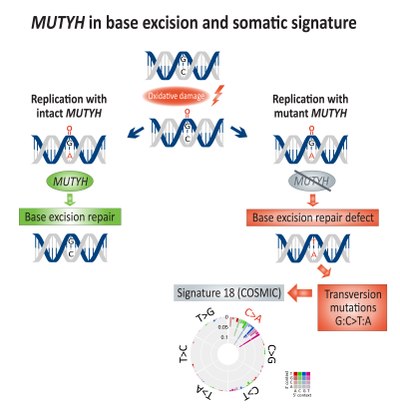
Wzbogacenie somatyczne w mutacje transwersyjne (G: C> T: A) zostało powiązane z niedoborem naprawy przez wycięcie zasady (BER) i powiązane z wadliwym MUTYH , glikozylazą DNA , w raku jelita grubego. Bezpośrednie DNA przez utlenianie prowadzi do powstania 8-oksoguaniny , która, jeśli nie zostanie naprawiona, doprowadzi do włączenia adeniny zamiast cytozyny podczas replikacji DNA. MUTYH koduje adeninę mutY enzym glikozylazy, który wycina niedopasowaną adeninę z 8-oksoguaniny : parowanie zasad adeniny , umożliwiając w ten sposób mechanizmy naprawy DNA z udziałem OGG1 (glikozylaza oksoguaniny) i NUDT1 (hydrolaza Nudix 1, znana również jako MTH1 , MutT homolog 1) w celu usunięcia uszkodzonej 8-oksoguaniny .
Ekspozycje na egzogenne genotoksyny
Wybrane egzogenne genotoksyny/ czynniki rakotwórcze oraz wywołane przez nie mutagenami mechanizmy uszkodzeń i naprawy DNA zostały powiązane z określonymi sygnaturami molekularnymi.
Promieniowanie ultrafioletowe (UV)
- Sygnatura 7 ma przewagę podstawień C>T w miejscach sąsiadujących pirymidyn (sąsiadujące C lub T), ze szczególnie diagnostycznym podzbiorem będącym mutacją dinukleotydu CC>TT. Ten wzór powstaje, ponieważ główne fotoprodukty DNA indukowane promieniowaniem UV łączą dwie sąsiednie pirymidyny; fotoproduktem jest zazwyczaj dimer cyklobutanu pirymidyny (CPD). Wydaje się, że specyficzność dla C> T wynika z milionowego przyspieszenia deaminacji C, gdy jest częścią CPD, przy czym powstały uracyl działa jak T. CPD są naprawiane poprzez naprawę przez wycięcie nukleotydów sprzężonych z transkrypcją , powodując silne odchylenie dla podstawień C>T wzbogaconych na nietranskrybowanej nici DNA. Regiony białka supresorowego guza, które są inaktywowane przez mutację w nowotworach skóry związanych ze światłem słonecznym, są takie same jak w rakach narządów nienarażonych na światło słoneczne, ale zmutowany nukleotyd jest często przesunięty o kilka zasad w miejsce, w którym może powstać CPD. Ultrafioletowy ekspozycja na promieniowanie jest zatem proponowanym mechanizmem mutagennym leżącym u podstaw tej sygnatury. UV ilustruje również subtelność w interpretacji sygnatury guza jako sygnatury mutagenu: tylko trzy czwarte mutacji indukowanych przez UV w laboratorium to mutacje sygnatury UV, ponieważ UV również wyzwala komórkowe procesy oksydacyjne. Dlatego nawet jeśli wszystkie mutacje w guzie były spowodowane promieniowaniem UV ze światła słonecznego, oczekuje się, że jedna czwarta mutacji nie będzie mutacjami sygnatur UV. Nie trzeba przywoływać drugiego czynnika rakotwórczego, aby wyjaśnić te mutacje, ale wymagany jest drugi proces mutacji. Identyfikacja sygnatury UV w guzie o nieznanej pierwotnej lokalizacji jest klinicznie ważna, ponieważ sugeruje rozpoznanie przerzutowego raka skóry i ma ważne implikacje terapeutyczne.
Środki alkilujące
- Signature 11 zidentyfikowano w nowotworach wcześniej narażonych na temozolamid, czynnik alkilujący . Ta sygnatura jest wzbogacona o podstawienia C> T na guaniny z powodu naprawy wycinania nukleotydów sprzężonej z transkrypcją . W tej sygnaturze występuje silne odchylenie nici transkrypcyjnej.
Tytoń
- Zarówno Signature 4 ( palenie tytoniu , rak płuc ), jak i Signature 29 ( żucie tytoniu , rak płaskonabłonkowy jamy ustnej dziąseł i policzków) wykazują błąd transkrypcji nici i wzbogacenie dla podstawień C> A, ale ich odpowiedni skład i wzorce (proporcja każdego typu mutacji ) nieznacznie się różnią.
- Proponowanym mechanizmem leżącym u podstaw Signature 4 jest usuwanie adduktów DNA ( benzo(a)piren tytoniowy kowalencyjnie związany z guaniną ) poprzez naprawę wycinania nukleotydów sprzężonych z transkrypcją (NER) maszyny.
Hipermutacja genu immunoglobuliny
Signature 9 został zidentyfikowany w przewlekłej białaczce limfocytowej i złośliwym chłoniaku z komórek B oraz wzbogaceniu funkcji dla zdarzeń transwersji T> G. Uważa się, że jest wynikiem mutagenezy związanej z podatną na błędy polimerazą η ( gen POLH ) .
Ostatnio sygnatura syntezy podatnej na błędy polimerazy η została powiązana z nowotworami niehematologicznymi (np. rakiem skóry ) i postawiono hipotezę, że przyczynia się do mutagenezy motywu YCG i może częściowo wyjaśniać wzrost substytucji dinukleotydów TC.
Historia
W latach 90-tych Curtis Harris z US National Cancer Institute i Bert Vogelstein z Johns Hopkins Oncology Center w Baltimore dokonali przeglądu danych pokazujących, że różne typy raka mają swój własny, unikalny zestaw mutacji w p53 , które prawdopodobnie były spowodowane różnymi czynników, takich jak substancje chemiczne w dymie tytoniowym lub światło ultrafioletowe ze słońca. Wraz z pojawieniem się sekwencjonowania nowej generacji , Michael Stratton dostrzegł potencjał technologii do zrewolucjonizowania naszego rozumienia zmian genetycznych w poszczególnych guzach, ustanawiając Wellcome Sanger Institute ogromne banki maszyn do sekwencjonowania DNA w ruchu, aby odczytać każdą pojedynczą literę DNA w guzie. Do 2009 roku Stratton i jego zespół stworzyli pierwsze pełne sekwencje genomu raka. Były to szczegółowe mapy przedstawiające wszystkie zmiany genetyczne i mutacje, które wystąpiły w obrębie dwóch pojedynczych nowotworów — czerniaka skóry i guza płuc. Genomy czerniaka i raka płuc były mocnym dowodem na to, że odciski palców konkretnych winowajców można zobaczyć w nowotworach o jednej głównej przyczynie. Guzy te nadal zawierały wiele mutacji, których nie można było wytłumaczyć światłem ultrafioletowym ani paleniem tytoniu. Praca detektywistyczna stała się znacznie bardziej skomplikowana w przypadku nowotworów o złożonym, wielokrotnym lub nawet zupełnie nieznanym pochodzeniu. Na zasadzie analogii wyobraź sobie naukowca zajmującego się kryminalistyką, który szuka odcisków palców na miejscu zbrodni. Technik medycyny sądowej może mieć szczęście i znaleźć zestaw doskonałych odcisków na szybie okiennej lub klamce drzwi, które pasują do znanego zabójcy. Jednak znacznie bardziej prawdopodobne jest, że odkryją mieszankę odcisków palców należących do całego szeregu osób - od ofiary i potencjalnych podejrzanych po niewinne osoby i policyjnych śledczych - wszystkie ułożone jeden na drugim na wszelkiego rodzaju powierzchniach. Jest to bardzo podobne do genomów raka, w których wiele wzorców mutacji często nakłada się na siebie, co powoduje, że dane są niezrozumiałe. Na szczęście doktorant Stratton's, Ludmił Aleksandrow wymyślił sposób matematycznego rozwiązania tego problemu. Aleksandrow wykazał, że wzorce mutacji z poszczególnych mutagenów znalezionych w guzie można odróżnić od siebie za pomocą matematycznego podejścia zwanego ślepą separacją źródeł . Nowo rozplątane wzorce mutacji nazwano sygnaturami mutacyjnymi. W 2013 roku Alexandrov i Stratton opublikowali pierwszą strukturę obliczeniową do rozszyfrowywania sygnatur mutacji z genomiki raka dane. Następnie zastosowali te ramy do ponad siedmiu tysięcy genomów raka, tworząc pierwszą kompleksową mapę sygnatur mutacji w ludzkim raku. Obecnie w repertuarze ludzkich nowotworów zidentyfikowano ponad sto sygnatur mutacji. W kwietniu 2022 roku opisano 58 nowych sygnatur mutacji.