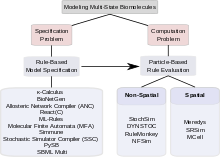
Wielostanowe modelowanie biomolekuł
Wielostanowe modelowanie biomolekuł odnosi się do szeregu technik stosowanych do przedstawiania i obliczania zachowania biologicznych cząsteczek lub kompleksów , które mogą przyjąć dużą liczbę możliwych stanów funkcjonalnych.
Biologiczne systemy sygnalizacyjne często opierają się na kompleksach makrocząsteczek biologicznych , które mogą podlegać kilku funkcjonalnie istotnym modyfikacjom, które są wzajemnie kompatybilne. Mogą więc istnieć w bardzo dużej liczbie funkcjonalnie różnych stanów. Modelowanie takich systemów wielostanowych stwarza dwa problemy: problem, jak opisać i określić system wielostanowy („problem specyfikacji”) oraz problem, jak użyć komputera do symulacji postępu systemu w czasie (tzw. „problem obliczeniowy”). Aby rozwiązać problem specyfikacji, modelarze odeszli w ostatnich latach od jawnej specyfikacji wszystkich możliwych stanów i skierowali się w stronę modelowanie oparte na regułach , które pozwala na niejawną specyfikację modelu, w tym rachunek κ, BioNetGen, Allosteric Network Compiler i inne. Aby rozwiązać problem obliczeniowy, zwrócili się ku metodom opartym na cząstkach, które w wielu przypadkach okazały się bardziej wydajne obliczeniowo niż metody oparte na populacji oparte na równaniach różniczkowych zwyczajnych , równaniach różniczkowych cząstkowych lub stochastycznym algorytmie symulacji Gillespiego . Biorąc pod uwagę obecną technologię komputerową, metody oparte na cząstkach są czasami jedyną możliwą opcją. Symulatory oparte na cząstkach dzielą się dalej na dwie kategorie: przestrzenne , takie jak StochSim, DYNSTOC, RuleMonkey i NFSim oraz symulatory przestrzenne, w tym Meredys, SRSim i MCell. Modelarze mogą zatem wybierać spośród różnych narzędzi; najlepszy wybór w zależności od konkretnego problemu. Trwają prace nad szybszymi i potężniejszymi metodami, które dają nadzieję na symulowanie w przyszłości coraz bardziej złożonych procesów sygnalizacyjnych.
Wstęp
Biomolekuły wielostanowe w transdukcji sygnału
W żywych komórkach sygnały są przetwarzane przez sieci białek , które mogą działać jako złożone urządzenia obliczeniowe. Sieci te opierają się na zdolności pojedynczych białek do istnienia w różnych funkcjonalnie różnych stanach, osiąganych poprzez wiele mechanizmów, w tym modyfikacje potranslacyjne , wiązanie ligandów , zmiany konformacyjne lub tworzenie nowych kompleksów . Podobnie kwasy nukleinowe może podlegać różnym transformacjom, w tym wiązaniu białek, wiązaniu innych kwasów nukleinowych, zmianom konformacyjnym i metylacji DNA .
Ponadto może współistnieć kilka rodzajów modyfikacji, wywierając łączny wpływ na biologiczną makrocząsteczkę w dowolnym momencie. Zatem biocząsteczka lub kompleks biomolekuł może często przyjmować bardzo dużą liczbę funkcjonalnie odrębnych stanów. Liczba stanów rośnie wykładniczo wraz z liczbą możliwych modyfikacji, co jest zjawiskiem znanym jako „ eksplozja kombinatoryczna ”. Jest to niepokojące dla biologów obliczeniowych , którzy modelują lub symulują takie biomolekuły, ponieważ rodzi to pytania o to, w jaki sposób można reprezentować i symulować tak dużą liczbę stanów.
Przykłady eksplozji kombinatorycznej
Biologiczne sieci sygnalizacyjne obejmują szeroki wachlarz odwracalnych interakcji , modyfikacji potranslacyjnych i zmian konformacyjnych . Ponadto często zdarza się, że białko składa się z kilku identycznych lub nieidentycznych podjednostek , a kilka białek i/lub rodzajów kwasów nukleinowych łączy się w większe kompleksy. Gatunki molekularne o kilku z tych cech mogą zatem istnieć w wielu możliwych stanach.
Na przykład oszacowano, że drożdżowe białko rusztowania Ste5 może być częścią 25666 unikalnych kompleksów białkowych. W E. coli cztery różne rodzaje receptorów chemotaksji oddziałują w grupach po trzy, a każdy pojedynczy receptor może istnieć w co najmniej dwóch możliwych konformacjach i ma do ośmiu miejsc metylacji , co daje miliardy potencjalnych stanów. Kinaza białkowa CaMKII jest dodekamerem dwunastu podjednostek katalitycznych , ułożonych w dwie heksameryczne pierścienie. Każda podjednostka może istnieć w co najmniej dwóch różnych konformacjach, a każda podjednostka ma różne fosforylacji i wiązania liganda. Niedawny model obejmował stany konformacyjne, dwa fosforylacji i dwa tryby wiązania wapnia/kalmoduliny , w sumie około miliarda możliwych stanów na pierścień heksameryczny. Model sprzężenia receptora EGF z kaskadą kinazy MAP przedstawiony przez Danosa i współpracowników odpowiada za różne gatunki molekularne, jednak autorzy zauważają kilka punktów, w których model można dalej rozszerzyć. Nowszy model ErbB uwzględnia nawet więcej niż jeden googol ( ) odrębny gatunek molekularny. Problem eksplozji kombinatorycznej jest również istotny dla biologii syntetycznej , z najnowszym modelem stosunkowo prostego syntetycznego obwodu genu eukariotycznego obejmującego 187 gatunków i 1165 reakcji .
Oczywiście nie wszystkie możliwe stany wielostanowej cząsteczki lub kompleksu będą koniecznie wypełnione. Rzeczywiście, w systemach, w których liczba możliwych stanów jest znacznie większa niż liczba cząsteczek w przedziale (np. komórce), nie może ich być. W niektórych przypadkach informacje empiryczne mogą być wykorzystane do wykluczenia pewnych stanów, jeśli na przykład niektóre kombinacje cech są niezgodne. W przypadku braku takich informacji należy jednak rozważyć a priori wszystkie możliwe stany . W takich przypadkach można zastosować modelowanie komputerowe, aby odkryć, w jakim stopniu różne stany są zaludnione.
Istnienie (lub potencjalne istnienie) tak dużej liczby gatunków molekularnych jest zjawiskiem kombinatorycznym : powstaje ze stosunkowo niewielkiego zestawu cech lub modyfikacji (takich jak modyfikacja potranslacyjna lub tworzenie kompleksów), które wspólnie decydują o stanie całego molekuły lub kompleksu, w taki sam sposób, jak istnienie zaledwie kilku wyborów w kawiarni (mała, średnia lub duża, z mlekiem lub bez, bezkofeinowa lub bez, dodatkowa porcja espresso ) szybko prowadzi do dużej liczby możliwych napojów (w tym przypadku 24; każdy dodatkowy wybór binarny podwaja tę liczbę). Chociaż trudno jest nam uchwycić całkowitą liczbę możliwych kombinacji, zwykle nie jest koncepcyjnie trudne zrozumienie (znacznie mniejszego) zestawu cech lub modyfikacji oraz wpływu, jaki każda z nich ma na funkcję biomolekuły. Szybkość, z jaką cząsteczka przechodzi określoną reakcję, będzie zwykle zależeć głównie od pojedynczej cechy lub niewielkiego podzbioru cech. To obecność lub brak tych cech decyduje o szybkości reakcji . Szybkość reakcji jest taka sama dla dwóch cząsteczek różniących się jedynie cechami, które nie mają wpływu na tę reakcję. Zatem liczba parametrów będzie znacznie mniejsza niż liczba reakcji. (W przykładzie z kawiarni dodanie dodatkowej porcji espresso będzie kosztować 40 centów, bez względu na wielkość napoju i to, czy zawiera mleko). To właśnie takie „lokalne zasady” są zwykle odkrywane w eksperymentach laboratoryjnych. Zatem model wielostanowy można konceptualizować w kategoriach kombinacji cech modułowych i lokalnych reguł. Oznacza to, że nawet model, który może wyjaśnić ogromną liczbę gatunków i reakcji molekularnych, niekoniecznie jest koncepcyjnie złożony.
Specyfikacja a obliczenia
Kombinatoryczna złożoność systemów sygnalizacyjnych obejmujących białka wielostanowe stwarza dwa rodzaje problemów. Pierwszy problem dotyczy tego, jak można określić taki system; tj. w jaki sposób modelarz może określić wszystkie kompleksy, wszystkie zmiany, jakim podlegają te kompleksy, oraz wszystkie parametry i warunki rządzące tymi zmianami w solidny i wydajny sposób. Ten problem nazywa się „problemem specyfikacji”. Drugi problem dotyczy obliczeń . Zadaje pytania o to, czy złożony kombinatorycznie model, raz określony, jest wykonalny obliczeniowo, biorąc pod uwagę dużą liczbę stanów i jeszcze większą liczbę możliwych przejść między stanami, czy można go przechowywać elektronicznie i czy można go ocenić w rozsądny sposób. ilość czasu obliczeniowego. Ten problem nazywa się „problemem obliczeniowym”. Wśród podejść, które zostały zaproponowane w celu rozwiązania złożoności kombinatorycznej w modelowaniu wielostanowym, niektóre dotyczą głównie rozwiązania problemu specyfikacji, inne koncentrują się na znalezieniu skutecznych metod obliczeniowych. Niektóre narzędzia dotyczą zarówno specyfikacji, jak i obliczeń. W poniższych sekcjach omówiono podejścia oparte na regułach do problemu specyfikacji i podejścia oparte na cząstkach do rozwiązania problemu obliczeniowego. Istnieje szeroka gama narzędzi obliczeniowych do modelowania wielostanowego.
Kwestia specyfikacji
Wyraźna specyfikacja
Najbardziej naiwnym sposobem określania np. białka w modelu biologicznym jest wyraźne określenie każdego z jego stanów i użycie każdego z nich jako cząsteczki molekularnej w ramach symulacji, która umożliwia przejścia ze stanu do stanu . Na przykład, jeśli białko może być ligandem lub nie, istnieć w dwóch stanach konformacyjnych (np. otwarte lub zamknięte) i znajdować się w dwóch możliwych obszarach subkomórkowych (np. cytozolowych lub związanych z błoną ), wówczas osiem możliwych stanów wynikowych może być wyraźnie wyliczone jako:
- związany, otwarty, cytozol
- związany, otwarty, membrana
- związany, zamknięty, cytozol
- związany, zamknięty, membrana
- niezwiązany, otwarty, cytozol
- niezwiązany, otwarty, membranowy
- niezwiązany, zamknięty, cytozol
- niezwiązany, zamknięty, membranowy
Wyliczanie wszystkich możliwych stanów jest procesem długotrwałym i potencjalnie podatnym na błędy. W przypadku kompleksów makrocząsteczkowych, które mogą przyjmować wiele stanów, wyliczanie każdego stanu szybko staje się żmudne, jeśli nie niemożliwe. Co więcej, dodanie pojedynczej dodatkowej modyfikacji lub cechy do modelu badanego kompleksu podwoi liczbę możliwych stanów (jeśli modyfikacja jest binarna) i ponad dwukrotnie zwiększy liczbę przejść, które należy określić.
Specyfikacja modelu oparta na regułach
Oczywiste jest, że szczegółowy opis, który wymienia wszystkie możliwe rodzaje cząsteczek (w tym wszystkie ich możliwe stany), wszystkie możliwe reakcje lub przemiany, którym mogą podlegać te gatunki, oraz wszystkie parametry rządzące tymi reakcjami, bardzo szybko staje się nieporęczny, ponieważ złożoność systemu biologicznego wzrasta. Modelarze szukali zatem niejawnych , a nie jawnych sposobów określania biologicznego systemu sygnalizacyjnego. Niejawny opis to taki, który grupuje reakcje i parametry, które mają zastosowanie do wielu typów cząsteczek w jednej matrycy reakcyjnej. Może również dodać zestaw warunków regulujących parametry reakcji, tj. prawdopodobieństwo lub szybkość zajścia reakcji lub to, czy w ogóle zachodzi. Tylko właściwości cząsteczki lub kompleksu, które mają znaczenie dla danej reakcji (albo wpływają na reakcję, albo na nią wpływają), są wyraźnie wymienione, a wszystkie inne właściwości są ignorowane w specyfikacji reakcji.
Na przykład szybkość dysocjacji ligandu z białka może zależeć od stanu konformacyjnego białka, ale nie od jego lokalizacji subkomórkowej. Niejawny opis wymieniałby zatem dwa procesy dysocjacji (z różnymi szybkościami, w zależności od stanu konformacyjnego), ale ignorowałby atrybuty odnoszące się do lokalizacji subkomórkowej, ponieważ nie wpływają one na szybkość dysocjacji ligandu, ani nie mają na nie wpływu. Ta zasada specyfikacji została podsumowana jako „Nie przejmuj się, nie pisz”.
Ponieważ nie jest napisany w kategoriach reakcji, ale bardziej ogólnych „reguł reakcji” obejmujących zestawy reakcji, ten rodzaj specyfikacji jest często nazywany „opartym na regułach” . Ten opis systemu w kategoriach zasad modułowych opiera się na założeniu, że tylko podzbiór cech lub atrybutów jest istotny dla określonej reguły reakcji. Tam, gdzie to założenie jest spełnione, zestaw reakcji można gruboziarniście podzielić na jedną regułę reakcji. To gruboziarniste ziarno zachowuje ważne właściwości podstawowych reakcji. Na przykład, jeśli reakcje są oparte na kinetyce chemicznej, wywodzą się z nich również reguły.
Istnieje wiele metod specyfikacji opartych na regułach. Generalnie specyfikacja modelu jest zadaniem odrębnym od wykonania symulacji. Dlatego wśród istniejących systemów specyfikacji modeli opartych na regułach niektóre koncentrują się wyłącznie na specyfikacji modelu, umożliwiając następnie użytkownikowi wyeksportowanie określonego modelu do dedykowanego silnika symulacyjnego. Jednak wiele rozwiązań problemu specyfikacji zawiera również metodę interpretacji określonego modelu. Odbywa się to poprzez dostarczenie metody symulacji modelu lub metody przekształcenia go do postaci, która może być wykorzystana do symulacji w innych programach.
Wczesną metodą specyfikacji opartą na regułach jest rachunek κ, algebra procesów , której można używać do kodowania makrocząsteczek ze stanami wewnętrznymi i miejscami wiązania oraz do określania reguł, według których wchodzą w interakcje. Rachunek κ zajmuje się jedynie zapewnieniem języka do kodowania modeli wielostanowych, a nie interpretacją samych modeli. Symulatorem kompatybilnym z Kappa jest KaSim.
BioNetGen to pakiet oprogramowania, który zapewnia zarówno możliwości specyfikacji, jak i symulacji. Modele oparte na regułach można zapisać przy użyciu określonej składni, języka BioNetGen (BNGL). Podstawową koncepcją jest przedstawienie systemów biochemicznych jako wykresów , gdzie cząsteczki są reprezentowane jako węzły (lub zbiory węzłów), a wiązania chemiczne jako krawędzie. Reguła reakcji odpowiada zatem regule przepisywania grafu. BNGL zapewnia składnię do określania tych wykresów i powiązanych reguł jako łańcuchów strukturalnych. BioNetGen może następnie wykorzystać te reguły do generowania równań różniczkowych zwyczajnych (ODE) w celu opisania każdej reakcji biochemicznej. Alternatywnie może wygenerować listę wszystkich możliwych gatunków i reakcji w SBML , który można następnie wyeksportować do pakietów oprogramowania symulacyjnego, które mogą odczytywać SBML . Można również skorzystać z własnego oprogramowania symulacyjnego BioNetGen opartego na ODE i jego zdolności do generowania reakcji w locie podczas symulacji stochastycznej. Ponadto model określony w BNGL może być odczytywany przez inne oprogramowanie symulacyjne, takie jak DYNSTOC, RuleMonkey i NFSim.
Innym narzędziem, które generuje pełne sieci reakcji na podstawie zestawu reguł, jest Allosteric Network Compiler (ANC). Koncepcyjnie ANC postrzega cząsteczki jako urządzenia allosteryczne z Monod-Wyman-Changeux (MWC), których interakcje są regulowane przez ich stan wewnętrzny, jak również zewnętrzne modyfikacje. Bardzo przydatną cechą ANC jest to, że automatycznie oblicza zależne parametry, narzucając w ten sposób termodynamiczną .
Rozszerzenie rachunku κ zapewnia React(C) . Autorzy React C pokazują, że może on wyrażać stochastyczny rachunek π. Zapewniają również algorytm symulacji stochastycznej oparty na algorytmie stochastycznym Gillespiego dla modeli określonych w React(C) .
ML-Rules jest podobny do React(C), ale zapewnia dodatkową możliwość zagnieżdżania: gatunek składowy modelu, ze wszystkimi jego atrybutami, może być częścią gatunku składowego wyższego rzędu. Dzięki temu ML-Rules może uchwycić wielopoziomowe modele, które mogą wypełnić lukę między, na przykład, serią procesów biochemicznych a makroskopowym zachowaniem całej komórki lub grupy komórek. Na przykład model podziału komórki w drożdżach rozszczepieniach obejmuje wiązanie i aktywację cykliny / cdc2 , wydzielanie i dyfuzję feromonów , podział komórek i ruchu komórek. Modele określone w ML-Rules mogą być symulowane przy użyciu struktury symulacji Jamesa II. Podobny zagnieżdżony język do reprezentowania wielopoziomowych systemów biologicznych został zaproponowany przez Oury i Plotkin. Formalizm specyfikacji oparty na strukturze molekularnych automatów skończonych (MFA) może być następnie wykorzystany do generowania i symulowania systemu ODE lub do symulacji stochastycznej przy użyciu kinetycznego algorytmu Monte Carlo .
Niektóre systemy specyfikacji oparte na regułach i powiązane z nimi narzędzia do generowania i symulacji sieci zostały zaprojektowane w celu uwzględnienia heterogeniczności przestrzennej, aby umożliwić realistyczną symulację interakcji w przedziałach biologicznych. Na przykład projekt Simmune obejmuje komponent przestrzenny: użytkownicy mogą określać swoje wielostanowe biomolekuły i interakcje w obrębie membran lub przedziałów o dowolnym kształcie. Objętość reakcji jest następnie dzielona na połączone woksele i generowana jest oddzielna sieć reakcji dla każdego z tych podobjętości.
Stochastic Simulator Compiler (SSC) umożliwia opartą na regułach, modułową specyfikację oddziałujących biomolekuł w regionach o dowolnie złożonej geometrii. Ponownie, system jest reprezentowany za pomocą wykresów, z interakcjami chemicznymi lub zdarzeniami dyfuzji sformalizowanymi jako reguły przepisywania wykresów. Następnie kompilator generuje całą sieć reakcji przed uruchomieniem stochastycznego algorytmu dyfuzji reakcji.
Inne podejście stosuje PySB, gdzie specyfikacja modelu jest osadzona w języku programowania Python . Model (lub część modelu) jest reprezentowany jako program w Pythonie. Pozwala to użytkownikom przechowywać procesy biochemiczne wyższego rzędu, takie jak kataliza lub polimeryzacja , jako makra i ponownie wykorzystywać je w razie potrzeby. Modele można symulować i analizować za pomocą bibliotek Pythona, ale modele PySB można również eksportować do formatów BNGL, kappa i SBML.
Modele obejmujące gatunki wielostanowe i wieloskładnikowe można również określić na poziomie 3 języka znaczników biologii systemów (SBML) przy użyciu pakietu multi. Dostępny jest projekt specyfikacji.
Tak więc, biorąc pod uwagę tylko stany i cechy ważne dla konkretnej reakcji, specyfikacja modelu oparta na regułach eliminuje potrzebę wyraźnego wyliczenia każdego możliwego stanu cząsteczkowego, który może ulec podobnej reakcji, a tym samym pozwala na wydajną specyfikację.
Kwestia obliczeń
Podczas przeprowadzania symulacji na modelu biologicznym każde oprogramowanie symulacyjne ocenia zestaw reguł, zaczynając od określonego zestawu warunków początkowych i zwykle przechodząc przez serię kroków czasowych, aż do określonego czasu końcowego. Jednym ze sposobów klasyfikowania algorytmów symulacyjnych jest przyjrzenie się poziomowi analizy, na którym stosowane są reguły: mogą one być oparte na populacji, oparte na pojedynczych cząsteczkach lub hybrydowe.
Ocena reguł oparta na populacji
W ocenie reguł na podstawie populacji reguły są stosowane do populacji. Wszystkie cząsteczki tego samego gatunku w tym samym stanie są połączone razem. Zastosowanie określonej reguły zmniejsza lub zwiększa wielkość jednej puli, być może kosztem innej.
Niektóre z najbardziej znanych klas podejść symulacyjnych w biologii obliczeniowej należą do rodziny opartej na populacji, w tym te oparte na numerycznym całkowaniu równań różniczkowych zwyczajnych i cząstkowych oraz algorytm symulacji stochastycznej Gillespiego.
Równania różniczkowe opisują zmiany stężeń cząsteczek w czasie w sposób deterministyczny. Symulacje oparte na równaniach różniczkowych zwykle nie próbują rozwiązać tych równań analitycznie, ale wykorzystują odpowiedni solwer numeryczny .
Algorytm stochastyczny Gillespiego zmienia skład puli cząsteczek poprzez progresję losowych zdarzeń reakcji, których prawdopodobieństwo jest obliczane na podstawie szybkości reakcji i liczby cząsteczek, zgodnie ze stochastycznym równaniem głównym .
W podejściach opartych na populacji można pomyśleć, że modelowany system znajduje się w danym stanie w dowolnym punkcie czasowym, gdzie stan jest zdefiniowany zgodnie z naturą i rozmiarem zaludnionych pul cząsteczek. Oznacza to, że przestrzeń wszystkich możliwych stanów może stać się bardzo duża. W przypadku niektórych metod symulacji wykorzystujących całkowanie numeryczne równań różniczkowych zwyczajnych i cząstkowych lub algorytm stochastyczny Gillespiego wszystkie możliwe pule cząsteczek i reakcje, którym podlegają, są definiowane na początku symulacji, nawet jeśli są puste. Takie metody „najpierw generuj” słabo skalują się wraz ze wzrostem liczby stanów molekularnych. Na przykład niedawno oszacowano, że nawet w przypadku prostego modelu CaMKII z zaledwie 6 stanami na podjednostki i 10 podjednostkami, wygenerowanie całej sieci reakcyjnej na procesorze Intel 2,54 GHz zajęłoby 290 lat. Procesor Xeon . Ponadto etap generowania modelu w metodach generujących najpierw niekoniecznie się kończy, na przykład gdy model obejmuje składanie białek w kompleksy o dowolnie dużych rozmiarach, takie jak włókna aktynowe . W takich przypadkach użytkownik musi określić warunek zakończenia.
Nawet jeśli można pomyślnie wygenerować duży system reakcji, jego symulacja przy użyciu oceny reguł opartej na populacji może przekroczyć granice obliczeniowe. W niedawnym badaniu wykazano, że potężny komputer nie jest w stanie symulować białka z więcej niż 8 ) przy użyciu zwykłych równań różniczkowych.
Zaproponowano metody zmniejszania rozmiaru przestrzeni stanów. Jednym z nich jest rozważenie tylko stanów sąsiadujących ze stanem obecnym (tj. stanów, które można osiągnąć w następnej iteracji) w każdym punkcie czasowym. Eliminuje to konieczność wyliczania wszystkich możliwych stanów na początku. Zamiast tego reakcje są generowane „w locie” przy każdej iteracji. Metody te są dostępne zarówno dla algorytmów stochastycznych, jak i deterministycznych. Metody te nadal opierają się na definicji (choć zredukowanej) sieci reakcji - w przeciwieństwie do metod „bez sieci” omówionych poniżej.
Nawet przy generowaniu sieci „w locie”, sieci generowane do oceny reguł opartych na populacji mogą stać się dość duże, a przez to trudne – jeśli nie niemożliwe – do obsługi obliczeniowej. Alternatywnym podejściem jest ocena reguł oparta na cząstkach.
Ocena reguł oparta na cząstkach
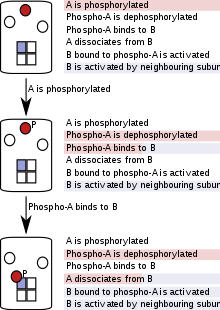
W symulacjach opartych na cząstkach (czasami nazywanych „opartymi na agentach”) białka, kwasy nukleinowe, kompleksy makrocząsteczkowe lub małe cząsteczki są reprezentowane jako pojedyncze obiekty oprogramowania , a ich postępy są śledzone w trakcie całej symulacji. Ponieważ ocena reguł oparta na cząstkach śledzi pojedyncze cząstki, a nie populacje, wiąże się to z wyższymi kosztami obliczeniowymi podczas modelowania systemów z dużą całkowitą liczbą cząstek, ale niewielką liczbą rodzajów (lub pul) cząstek. Jednak w przypadku złożoności kombinatorycznej modelowanie pojedynczych cząstek jest zaletą, ponieważ w dowolnym punkcie symulacji należy wziąć pod uwagę tylko istniejące cząsteczki, ich stany i reakcje, którym mogą podlegać. Ocena reguł oparta na cząstkach nie wymaga generowania pełnych lub częściowych sieci reakcji na początku symulacji ani w żadnym innym punkcie symulacji i dlatego jest nazywana „bez sieci”.
Metoda ta zmniejsza złożoność modelu na etapie symulacji, a tym samym oszczędza czas i moc obliczeniową. Symulacja podąża za każdą cząstką, a na każdym etapie symulacji cząsteczka „widzi” tylko reakcje (lub zasady), które jej dotyczą. Zależy to od stanu cząstki oraz, w niektórych implementacjach, od stanów jej sąsiadów w holoenzymie lub kompleksie. W miarę postępu symulacji stany cząstek są aktualizowane zgodnie z regułami, które są wystrzeliwane.
Niektóre pakiety symulacji oparte na cząstkach wykorzystują formalizm ad-hoc do specyfikacji reagentów, parametrów i reguł. Inni mogą odczytywać pliki w uznanym formacie specyfikacji opartym na regułach, takim jak BNGL.
Metody oparte na cząstkach nieprzestrzennych
StochSim to cząsteczkowy symulator stochastyczny , służący głównie do modelowania reakcji chemicznych i innych przemian molekularnych. Algorytm zastosowany w StochSim różni się od bardziej znanego algorytmu stochastycznego Gillespiego tym, że działa na pojedynczych jednostkach, a nie na pulach jednostek, co czyni go bardziej opartym na cząsteczkach niż na populacji.
W StochSim każdy gatunek molekularny może być wyposażony w szereg binarnych flag stanu reprezentujących określoną modyfikację. Reakcje można uzależnić od zestawu flag stanu ustawionych na określone wartości. Ponadto wynikiem reakcji może być zmiana flagi stanu. Co więcej, jednostki można układać w tablice geometryczne (na przykład dla holoenzymów składających się z kilku podjednostek), a reakcje mogą być „wrażliwe na sąsiada”, tj. na prawdopodobieństwo reakcji dla danej jednostki wpływa wartość flagi stanu na jednostce sąsiedniej. Te właściwości sprawiają, że StochSim idealnie nadaje się do modelowania cząsteczek wielostanowych ułożonych w holoenzymy lub kompleksy o określonej wielkości. Rzeczywiście, StochSim został wykorzystany do modelowania klastrów bakteryjnych receptorów chemotaktycznych i holoenzymów CaMKII.
Rozszerzenie do StochSim zawiera oparty na cząsteczkach symulator DYNSTOC, który wykorzystuje algorytm podobny do StochSim do symulacji modeli określonych w języku BioNetGen (BNGL) i poprawia obsługę cząsteczek w kompleksach makromolekularnych .
Innym symulatorem stochastycznym opartym na cząstkach, który może odczytywać pliki wejściowe BNGL, jest RuleMonkey. Jego algorytm symulacji różni się od algorytmów leżących u podstaw zarówno StochSim, jak i DYNSTOC tym, że krok czasowy symulacji jest zmienny.
Bezsieciowy symulator stochastyczny (NFSim) różni się od tych opisanych powyżej tym, że umożliwia definiowanie szybkości reakcji jako dowolnych wyrażeń matematycznych lub warunkowych, a tym samym ułatwia selektywne, gruboziarniste modelowanie . RuleMonkey i NFsim implementują różne, ale powiązane algorytmy symulacji. Szczegółowy przegląd i porównanie obu narzędzi podają Yang i Hlavacek.
Łatwo sobie wyobrazić system biologiczny, w którym niektóre składniki są złożonymi cząsteczkami wielostanowymi, podczas gdy inne mają kilka możliwych stanów (lub nawet tylko jeden) i występują w dużych ilościach. Zaproponowano podejście hybrydowe do modelowania takich systemów: w ramach hybrydowej struktury cząstek/populacji (HPP) użytkownik może określić model oparty na regułach, ale może wyznaczyć niektóre gatunki, które mają być traktowane jako populacje (a nie cząstki) w kolejnych symulacja. Ta metoda łączy w sobie zalety obliczeniowe modelowania opartego na cząstkach dla systemów wielostanowych o stosunkowo małej liczbie cząsteczek oraz modelowania opartego na populacji dla systemów o dużej liczbie cząsteczek i małej liczbie możliwych stanów. Specyfikacja modeli HPP jest obsługiwana przez BioNetGen, a symulacje można przeprowadzać za pomocą NFSim.
Metody oparte na cząstkach przestrzennych

Metody oparte na cząstkach przestrzennych różnią się od metod opisanych powyżej ich jawną reprezentacją przestrzeni.
Jednym z przykładów symulatora opartego na cząstkach, który pozwala na reprezentację przedziałów komórkowych, jest SRSim. SRSim jest zintegrowany z symulatorem dynamiki molekularnej LAMMPS i umożliwia użytkownikowi określenie modelu w BNGL. SRSim pozwala użytkownikom określić geometrię cząstek w symulacji, a także miejsca interakcji. Dlatego jest szczególnie dobry w symulowaniu składania i struktury złożonych kompleksów biomolekularnych, o czym świadczy niedawny model wewnętrznego kinetochoru .
MCell umożliwia śledzenie pojedynczych cząsteczek w dowolnie złożonych środowiskach geometrycznych, które są definiowane przez użytkownika. Pozwala to na symulacje biomolekuł w realistycznych rekonstrukcjach żywych komórek, w tym komórek o złożonej geometrii, takich jak neurony . Komora reakcyjna jest rekonstrukcją kolca dendrytycznego.
MCell wykorzystuje formalizm ad-hoc w samym MCell, aby określić model wielostanowy: w MCell możliwe jest przypisanie „szczelin” do dowolnego gatunku molekularnego . Każda szczelina oznacza określoną modyfikację, a do cząsteczki można przypisać dowolną liczbę szczelin. Każde miejsce może być zajęte przez określony stan. Stany niekoniecznie są binarne. Na przykład szczelina opisująca wiązanie określonego ligandu z białkiem będącym przedmiotem zainteresowania może przyjmować stany „niezwiązany”, „częściowo związany” i „całkowicie związany”.
Składnia slot-and-state w MCell może być również wykorzystana do modelowania białek multimerycznych lub kompleksów makrocząsteczkowych. Kiedy jest używany w ten sposób, szczelina jest symbolem zastępczym dla podjednostki lub składnika molekularnego kompleksu , a stan szczeliny będzie wskazywał, czy określony składnik białkowy jest nieobecny, czy obecny w kompleksie. Sposób myślenia o tym polega na tym, że makrocząsteczki MCell mogą mieć kilka wymiarów : „wymiar stanu” i jeden lub więcej „wymiarów przestrzennych”. „Wymiar stanu” jest używany do opisania wielu możliwych stanów składających się na białko wielostanowe, podczas gdy wymiar(y) przestrzenny(e) opisują topologiczne między sąsiednimi podjednostkami lub członkami kompleksu makrocząsteczkowego. Jedną wadą tej metody przedstawiania kompleksów białkowych, w porównaniu z Meredys, jest to, że MCell nie pozwala na dyfuzję kompleksów , a tym samym cząsteczek wielostanowych. W niektórych przypadkach można to obejść, dostosowując stałe dyfuzji ligandów, które oddziałują z kompleksem, stosując funkcje punktów kontrolnych lub łącząc symulacje na różnych poziomach.
Przykłady modeli wielostanowych w biologii
(bynajmniej nie wyczerpujący) wybór modeli układów biologicznych obejmujących cząsteczki wielostanowe i wykorzystujących niektóre z omówionych tu narzędzi przedstawiono w poniższej tabeli.
| Układ biologiczny | Specyfikacja | Obliczenie | Odniesienie | |
|---|---|---|---|---|
| Bakteryjny szlak sygnałowy chemotaksji | StochSim | StochSim | ||
| Regulacja CaMKII | StochSim | StochSim | ||
| Sygnalizacja receptora ERBB | BioNet Gen | NFSim | ||
| Eukariotyczne obwody genów syntetycznych | BioNetGen, PROMOT | KOPASI | ||
| Sygnalizacja RNA | Kappa | KaSim | ||
| Kooperatywność białek allosterycznych | Kompilator sieci allosterycznej (ANC) | MATLAB | ||
| Chemosensing w Dictyostelium | Szymon | Szymon | ||
| Aktywacja receptora komórek T | SSC | SSC | ||
| Ludzki kinetochor mitotyczny | BioNet Gen | SRSim | ||
| Cykl komórkowy drożdży rozszczepialnych | Zasady ML | JAKUB II |
Zobacz też
- Biomolekuły
- Sygnalizacja komórkowa
- Wiązanie chemiczne
- Kinetyka enzymów
- Artykuły recenzowane zewnętrznie
- Białka
- Symulacja stochastyczna
- Artykuły Wikipedii opublikowane w PLOS Computational Biology
- Artykuły z Wikipedii opublikowane w recenzowanej literaturze
- Artykuły z Wikipedii opublikowane w recenzowanej literaturze (J2W)