Niezbędny gen
Niezbędne geny to geny niezbędne organizmom do wzrostu i reprodukcji potomstwa w określonym środowisku. Jednak bycie niezbędnym w dużym stopniu zależy od warunków, w jakich żyje organizm. Na przykład gen wymagany do trawienia skrobi jest niezbędny tylko wtedy, gdy skrobia jest jedynym źródłem energii. Ostatnio podjęto systematyczne próby zidentyfikowania tych genów, które są absolutnie niezbędne do utrzymania życia, pod warunkiem dostępności wszystkich składników odżywczych. Takie eksperymenty doprowadziły do wniosku, że bezwzględnie wymagana liczba genów dla bakterii jest rzędu około 250-300. Niezbędne geny organizmów jednokomórkowych kodują białka dla trzech podstawowych funkcji, w tym przetwarzania informacji genetycznej, otoczek komórkowych i produkcji energii. Te funkcje genów są wykorzystywane do utrzymania centralnego metabolizmu , replikacji DNA , translacji genów na białka , utrzymania podstawowej struktury komórkowej i pośredniczenia w procesach transportu do iz komórki. W porównaniu z organizmami jednokomórkowymi, organizmy wielokomórkowe mają bardziej istotne geny związane z komunikacją i rozwojem. Większość podstawowych genów wirusów jest związana z przetwarzaniem i utrzymywaniem informacji genetycznej. W przeciwieństwie do większości organizmów jednokomórkowych wirusy nie mają wielu genów niezbędnych do metabolizmu, co zmusza je do przejęcia kontroli nad metabolizmem gospodarza. Większość genów nie jest niezbędna, ale zapewnia selektywne korzyści i zwiększoną sprawność . Dlatego zdecydowana większość genów nie jest niezbędna, a wiele z nich można usunąć bez konsekwencji, przynajmniej w większości przypadków.
Bakterie: badania całego genomu
Zastosowano dwie główne strategie identyfikacji istotnych genów na podstawie całego genomu: ukierunkowana delecja genów i losowa mutageneza przy użyciu transpozonów . W pierwszym przypadku poszczególne geny z adnotacjami (lub ORF ) są całkowicie usuwane z genomu w systematyczny sposób. W mutagenezie za pośrednictwem transpozonów transpozony są losowo wstawiane w jak największej liczbie pozycji w genomie, mając na celu zakłócenie funkcji docelowych genów (patrz rysunek poniżej). Mutanty insercyjne, które wciąż są zdolne do przeżycia lub wzrostu, sugerują transpozon wstawiony do genu, który nie jest niezbędny do przeżycia. Położenie insercji transpozonu można określić poprzez hybrydyzację z mikromacierzami lub poprzez sekwencjonowanie transpozonów . Wraz z rozwojem CRISPR określono również istotność genów poprzez hamowanie ekspresji genów poprzez interferencję CRISPR . Podsumowanie takich ekranów przedstawiono w tabeli.
| Organizm | Mutageneza | metoda | Odczytać | ORF | nie-ess. | Niezbędny | % Es. | Notatki | Ref. |
|---|---|---|---|---|---|---|---|---|---|
| Mycoplasma genitalium/pneumoniae | Losowy | Populacja | Sekwencjonowanie | 482 | 130 | 265–350 | 55–73% | --- | |
| Mycoplasma genitalium | Losowy | Klony | Sekwencjonowanie | 482 | 100 | 382 | 79% | pne | |
| Staphylococcus aureus WCUH29 | Losowy | Klony | Sekwencjonowanie | 2600 | nie dotyczy | 168 | nie dotyczy | pne | |
| Staphylococcus aureus RN4220 | Losowy | Klony | Sekwencjonowanie | 2892 | nie dotyczy | 658 | 23% | --- | |
| Haemophilus influenzae Rd | Losowy | Populacja | Footprint-PCR | 1657 | 602 | 670 | 40% | --- | |
| Streptococcus pneumoniae Rx-1 | Ukierunkowane | Klony | Tworzenie kolonii | 2043 | 234 | 113 | nie dotyczy | C | |
| Streptococcus pneumoniae D39 | Ukierunkowane | Klony | Tworzenie kolonii | 2043 | 560 | 133 | nie dotyczy | C | |
| Streptococcus pyogenes 5448 | Losowy | transpozon | Tn-nast | 1865 | ? | 227 | 12% | --- | |
| Streptococcus pyogenes NZ131 | Losowy | transpozon | Tn-nast | 1700 | ? | 241 | 14% | --- | |
| Streptococcus sanguinis SK36 | Ukierunkowane | Klony | Tworzenie kolonii | 2270 | 2052 | 218 | 10% | a, j | |
| Mycobacterium tuberculosis H37Rv | Losowy | Populacja | Mikromacierz | 3989 | 2567 | 614 | 15% | --- | |
| Prątek gruźlicy | Losowy | transpozon | ? | 3989 | ? | 401 | 10% | --- | |
| Mycobacterium tuberculosis H37Rv | Losowy | transpozon | Sekwencjonowanie NG | 3989 | ? | 774 | 19% | --- | |
| Mycobacterium tuberculosis H37Rv | Losowy | transpozon | Sekwencjonowanie NG | 3989 | 3364 | 625 | 16% | Cześć | |
| Prątek gruźlicy | --- | Obliczeniowy | Obliczeniowy | 3989 | ? | 283 | 7% | --- | |
| Mycobacterium tuberculosis H37Rv | Ukierunkowane | CRISPRi | Sekwencjonowanie NG | 4052 | 33,15 | 737 | 18% | --- | |
| Bacillus subtilis 168 | Ukierunkowane | Klony | Tworzenie kolonii | 4105 | 3830 | 261 | 7% | a,d,g | |
| Escherichia coli K-12 MG1655 | Losowy | Populacja | Footprint-PCR | 4308 | 3126 | 620 | 14% | --- | |
| Escherichia coli K-12 MG1655 | Ukierunkowane | Klony | Tworzenie kolonii | 4308 | 2001 | nie dotyczy | nie dotyczy | a, e | |
| Escherichia coli K-12 BW25113 | Ukierunkowane | Klony | Tworzenie kolonii | 4390 | 3985 | 303 | 7% | A | |
| Pseudomonas aeruginosa PAO1 | Losowy | Klony | Sekwencjonowanie | 5570 | 4783 | 678 | 12% | A | |
| Porphyromonas gingivalis | Losowy | transpozon | Sekwencjonowanie | 1990 | 1527 | 463 | 23% | --- | |
| Pseudomonas aeruginosa PA14 | Losowy | Klony | Sekwencjonowanie | 5688 | 4469 | 335 | 6% | za,f | |
| Salmonella typhimurium | Losowy | Klony | Sekwencjonowanie | 4425 | nie dotyczy | 257 | ~11% | pne | |
| Helicobacter pylori G27 | Losowy | Populacja | Mikromacierz | 1576 | 1178 | 344 | 22% | --- | |
| Corynebacterium glutamicum | Losowy | Populacja | ? | 3002 | 2352 | 650 | 22% | --- | |
| Francisella novicida | Losowy | transpozon | ? | 1719 | 1327 | 392 | 23% | --- | |
| Vibrio cholerae N16961 | Losowy | transpozon | ? | 3890 | ? | 779 | 20% | --- | |
| Salmonella Typhi | Losowy | transpozon | ? | 4646 | ? | 353 | 8% | --- | |
| Staphylococcus aureus | Losowy | transpozon | ? | ~ 2600 | ? | 351 | 14% | --- | |
| Caulobacter crescentus | Losowy | transpozon | Tn-Sekw | 3876 | 3240 | 480 | 12,2% | --- | |
| Neisseria meningitidis | Losowy | transpozon | ? | 2158 | ? | 585 | 27% | --- | |
| Desulfovibrio alaskensis | Losowy | transpozon | Sekwencjonowanie | 3258 | 2871 | 387 | 12% | --- |
Tabela 1. Niezbędne geny u bakterii . Mutageneza : docelowymi mutantami są delecje genów; losowe mutanty to insercje transpozonu . Metody : Klony wskazują na delecje pojedynczego genu, populacja wskazuje na mutagenezę całej populacji, np. przy użyciu transpozonów. Niezbędne geny z ekranów populacji obejmują geny niezbędne dla sprawności (patrz tekst). ORF : liczba wszystkich otwartych ramek odczytu w tym genomie. Uwagi : (a) dostępna kolekcja mutantów; (b) metoda bezpośredniego badania przesiewowego pod kątem istotności (np. za pomocą antysensownego RNA), która nie dostarcza informacji o genach nieistotnych. (c) Dostępny jest tylko częściowy zestaw danych. (d) Obejmuje przewidywaną niezbędność genów i kompilację danych z opublikowanych badań nad niezbędnością pojedynczego genu. (e) Projekt w toku. ( f ) Wydedukowane przez porównanie dwóch zestawów danych dotyczących niezbędności genów uzyskanych niezależnie w P. aeruginosa PA14 i PAO1. (g) Pierwotny wynik 271 niezbędnych genów został poprawiony do 261, przy czym 31 genów, które uważano za niezbędne, w rzeczywistości nie są niezbędne, podczas gdy od tego czasu opisano 20 nowych niezbędnych genów. (h) Zliczanie genów z domenami niezbędnymi i tymi, które prowadzą do defektów wzrostu, gdy są zakłócone, jako niezbędne, oraz tych, które prowadzą do przewagi wzrostu, gdy są zakłócone, jako nieistotne. (i) Zaangażowany w pełni nasyconą bibliotekę mutantów składającą się z 14 powtórzeń, z 84, 3% możliwych miejsc insercji z co najmniej jedną insercją transpozonu. (j) Każdy niezbędny gen został niezależnie potwierdzony co najmniej pięć razy.

Na podstawie badań eksperymentalnych całego genomu i analizy biologii systemów, Kong i in. opracowali niezbędną bazę danych genów. (2019) do przewidywania > 4000 gatunków bakterii.
eukarionty
W Saccharomyces cerevisiae (pączkujące drożdże) 15-20% wszystkich genów jest niezbędnych. W Schizosaccharomyces pombe (drożdże rozszczepialne) skonstruowano 4836 heterozygotycznych delecji obejmujących 98,4% z 4914 otwartych ramek odczytu kodujących białka. 1260 z tych delecji okazało się niezbędnych.
Podobne badania przesiewowe są trudniejsze do przeprowadzenia u innych organizmów wielokomórkowych, w tym ssaków (jako model dla człowieka), ze względów technicznych, a ich wyniki są mniej jednoznaczne. Jednak opracowano różne metody dla nicienia C. elegans , muszki owocówki i danio pręgowanego (patrz tabela). Niedawne badanie 900 genów myszy wykazało, że 42% z nich było niezbędnych, chociaż wybrane geny nie były reprezentatywne.
Eksperymenty z nokautem genów nie są możliwe, a przynajmniej nieetyczne u ludzi. Jednak naturalne mutacje doprowadziły do identyfikacji mutacji, które prowadzą do wczesnej śmierci zarodka lub późniejszej śmierci. Należy zauważyć, że wiele genów u ludzi nie jest absolutnie niezbędnych do przeżycia, ale zmutowane mogą powodować poważne choroby. Takie mutacje są skatalogowane w Online Mendelian Inheritance in Man (OMIM). W analizie obliczeniowej zmienności genetycznej i mutacji u 2472 ludzkich ortologów znanych podstawowych genów u myszy, Georgi i in. stwierdzili silną, oczyszczającą selekcję i stosunkowo zmniejszone poziomy zmienności sekwencji, co wskazuje, że te ludzkie geny są również niezbędne.
Chociaż udowodnienie, że gen jest niezbędny u ludzi, może być trudne, można wykazać, że gen nie jest niezbędny lub nawet nie powoduje choroby. Na przykład sekwencjonowanie genomów 2636 obywateli Islandii i genotypowanie 101 584 dodatkowych osób wykazało, że 8041 osób miało całkowicie wyłączony 1 gen (tj. osoby te były homozygotami pod względem niefunkcjonalnego genu). Spośród 8041 osobników z całkowitym nokautem, 6885 oszacowano jako homozygoty , a 1249 jako złożone heterozygoty (tj. oba allele genu zostały wyeliminowane, ale oba allele miały różne mutacje). U tych osobników łącznie 1171 z 19135 genów ludzkich ( RefSeq ) (6,1%) zostało całkowicie znokautowanych. Stwierdzono, że te 1171 genów jest nieistotnych dla ludzi — przynajmniej nie zgłoszono żadnych powiązanych chorób. Podobnie, sekwencje egzomu 3222 dorosłych brytyjskiego pochodzenia pakistańskiego o wysokim pokrewieństwie rodzicielskim ujawniły 1111 rzadkich wariantów homozygotycznych genotypów z przewidywaną utratą funkcji genu (LOF = nokaut) w 781 genach. To badanie wykazało średnio 140 przewidywanych genotypów LOF (na osobę), w tym 16 rzadkich ( częstość mniejszych alleli <1%) heterozygot, 0,34 rzadkich homozygot, 83,2 pospolitych heterozygot i 40,6 pospolitych homozygot. Prawie wszystkie rzadkie homozygotyczne genotypy LOF znaleziono w autozygotycznych (94,9%). Chociaż większość z tych osób nie miała oczywistych problemów zdrowotnych wynikających z ich wadliwych genów, możliwe jest, że po bardziej szczegółowym badaniu można wykryć drobne problemy zdrowotne.
Podsumowanie ekranów niezbędności przedstawiono w poniższej tabeli (głównie w oparciu o bazę danych niezbędnych genów.
| Organizm | metoda | Niezbędne geny | Ref. |
| Arabidopsis thaliana | Wstawienie T-DNA | 777 | |
| Caenorhabditis elegans (robak) | interferencja RNA | 294 | |
| Danio rerio (danio pręgowany) | Mutageneza insercyjna | 288 | |
| Drosophila melanogaster (muszka owocowa) | Mutageneza wstawiania elementu P | 339 | |
| Homo sapiens (człowiek) | Wyszukiwanie literatury | 118 | |
| Homo sapiens (człowiek) | Ekran oparty na CRISPR/Cas9 | 1878 | |
| Homo sapiens (człowiek) | Ekran haploidalnej pułapki genowej | ~ 2000 | |
| Homo sapiens (człowiek) | ortologi myszy | 2472 | |
| Mus musculus (mysz) | Wyszukiwanie literatury | 2114 | |
| Saccharomyces cerevisiae (drożdże) | Delecje pojedynczych genów | 878 | |
| Saccharomyces cerevisiae (drożdże) | Delecje pojedynczych genów | 1105 | |
| Schizosaccharomyces pombe (drożdże) | Delecje pojedynczych genów | 1260 |
Wirusy
Wirusom brakuje wielu genów niezbędnych do metabolizmu, co zmusza je do przejęcia kontroli nad metabolizmem żywiciela. W przypadku kilku wirusów przeprowadzono badania przesiewowe pod kątem niezbędnych genów. Na przykład stwierdzono , że ludzki wirus cytomegalii (CMV) ma 41 niezbędnych, 88 nieistotnych i 27 wzmacniających ORF (łącznie 150 ORF). Większość genów niezbędnych i wzmacniających znajduje się w regionie centralnym, a geny nieistotne generalnie skupiają się w pobliżu końców genomu wirusa.
Tscharke i Dobson (2015) opracowali kompleksowe badanie podstawowych genów wirusa krowianki i przypisali role każdej z 223 ORF szczepu Western Reserve (WR) i 207 ORF szczepu Copenhagen, oceniając ich rolę w replikacji w hodowli komórkowej. Zgodnie z ich definicją, gen jest uważany za niezbędny (tj. odgrywa rolę w hodowli komórkowej), jeśli jego delecja powoduje ponad 10-krotny spadek miana wirusa na krzywej wzrostu jedno- lub wieloetapowego. Wszystkie geny biorące udział w produkcji zawiniętego wirionu, tworzeniu ogona aktyny i pozakomórkowym uwalnianiu wirionu również uznano za niezbędne. Geny, które wpływają na rozmiar płytki, ale nie na replikację, zostały określone jako nieistotne. Zgodnie z tą definicją 93 geny są wymagane do replikacji wirusa krowianki w hodowli komórkowej, podczas gdy 108 i 94 ORF, odpowiednio z WR i Kopenhagi, nie są niezbędne. Wirusy krowianki z delecjami na obu końcach genomu zachowywały się zgodnie z oczekiwaniami, wykazując jedynie łagodne defekty lub defekty w zakresie gospodarza. W przeciwieństwie do tego połączenie delecji na obu końcach genomu szczepu VACV WR spowodowało niszczący defekt wzrostu we wszystkich testowanych liniach komórkowych. Pokazuje to, że delecje pojedynczego genu nie są wystarczające do oceny istotności genów i że więcej genów jest niezbędnych w wirusie krowianki, niż pierwotnie sądzono.
Jednym z bakteriofagów przeszukiwanych pod kątem niezbędnych genów jest mykobakteriofag Giles. Co najmniej 35 z 78 przewidywanych genów Gilesa (45%) jest nieistotnych dla wzrostu litycznego. Stwierdzono, że 20 genów jest niezbędnych. Głównym problemem związanym z genami fagowymi jest to, że większość ich genów pozostaje funkcjonalnie nieznana, stąd ich rola jest trudna do oceny. Badanie przesiewowe Salmonella enterica SPN3US ujawniło 13 niezbędnych genów, chociaż pozostaje niejasne, ile genów naprawdę przetestowano.
Ilościowa analiza niezbędności genów
Teoretycznie istotne geny są jakościowe. Jednak w zależności od otaczającego środowiska niektóre podstawowe mutanty genów mogą wykazywać częściowe funkcje, które można określić ilościowo w niektórych badaniach. Na przykład delecja określonego genu może zmniejszyć tempo wzrostu (lub współczynnik płodności lub inne cechy) do 90% typu dzikiego. Jeśli istnieją izozymy lub alternatywne szlaki dla niezbędnych genów, można je całkowicie usunąć. Za pomocą interferencji CRISPR można modulować lub „dostrajać” ekspresję podstawowych genów, co prowadzi do ilościowych (lub ciągłych) zależności między poziomem ekspresji genów a wielkością kosztów sprawności wykazywanych przez danego mutanta.
Śmiertelność syntetyczna
Dwa geny są syntetycznie śmiertelne, jeśli żaden z nich nie jest niezbędny, ale gdy oba są zmutowane, podwójny mutant jest śmiertelny. Niektóre badania oszacowały, że liczba syntetycznych genów śmiercionośnych może być rzędu 45% wszystkich genów.
Warunkowo niezbędne geny

Wiele genów jest niezbędnych tylko w określonych okolicznościach. Na przykład, jeśli aminokwas lizyna jest dostarczany do komórki, jakikolwiek gen wymagany do wytworzenia lizyny nie jest niezbędny. Jednak gdy lizyna nie jest dostarczana, niezbędne stają się geny kodujące enzymy do biosyntezy lizyny, ponieważ bez lizyny nie jest możliwa synteza białek.
Streptococcus pneumoniae wymaga 147 genów do wzrostu i przeżycia w ślinie , więcej niż 113-133, które znaleziono w poprzednich badaniach.
Delecja genu może spowodować śmierć lub blokadę podziału komórki . Chociaż ten ostatni przypadek może implikować „przeżycie” przez pewien czas, bez podziału komórki komórka może ostatecznie umrzeć. Podobnie, zamiast zablokowanego podziału komórki, komórka może mieć zmniejszony wzrost lub metabolizm w zakresie od prawie niewykrywalnego do prawie normalnego. Tak więc istnieje gradient od „niezbędnego” do całkowicie nieistotnego, ponownie w zależności od stanu. W ten sposób niektórzy autorzy rozróżniają geny „ niezbędne do przeżycia ” i „ niezbędne do sprawności ”.
Rola podłoża genetycznego . Podobnie jak w przypadku warunków środowiskowych, podłoże genetyczne może determinować niezbędność genu: gen może być niezbędny u jednej osoby, ale nie u innej, biorąc pod uwagę jej podłoże genetyczne. Duplikacje genów są jednym z możliwych wyjaśnień (patrz poniżej).
Uzależnienie metaboliczne . Geny zaangażowane w pewne szlaki biosyntezy, takie jak synteza aminokwasów , mogą stać się nieistotne, jeśli jeden lub więcej aminokwasów jest dostarczanych przez pożywkę hodowlaną lub inny organizm. Jest to główny powód, dla którego wiele pasożytów (np. Cryptosporidium hominis ) lub bakterii endosymbiontycznych utraciło wiele genów (np. Chlamydia ). Takie geny mogą być niezbędne, ale obecne tylko w organizmie gospodarza. Na przykład Chlamydia trachomatis nie może syntetyzować de novo nukleotydów purynowych i pirymidynowych , więc te bakterie są zależne od genów biosyntezy nukleotydów gospodarza.
Inny rodzaj zależności metabolicznej, niezwiązany z interakcjami międzygatunkowymi, można znaleźć, gdy bakterie hoduje się w określonych warunkach odżywczych . Na przykład ponad 100 genów staje się niezbędnych, gdy Escherichia coli jest hodowana na podłożach o ograniczonej zawartości składników odżywczych. Konkretnie, dehydrogenaza izocytrynianowa (icd) i syntaza cytrynianowa (gltA) to dwa enzymy, które są częścią cyklu kwasu trikarboksylowego (TCA) . Oba geny są niezbędne w pożywce minimalnej M9 (która dostarcza tylko najbardziej podstawowych składników odżywczych). Jednakże, gdy pożywki są uzupełniane 2-oksoglutaranem lub glutaminianem , te geny nie są już niezbędne.
Duplikacje genów i alternatywne szlaki metaboliczne
Wiele genów jest zduplikowanych w genomie, a wiele organizmów ma różne szlaki metaboliczne (alternatywny szlak metaboliczny) do syntezy tych samych produktów. Takie duplikacje ( paralogi ) i alternatywne szlaki metaboliczne często sprawiają, że istotne geny stają się nieistotne, ponieważ duplikat może zastąpić oryginalną kopię. Na przykład gen kodujący enzym kinazę aspartokinazy jest niezbędny w E. coli . Natomiast Bacillus subtilis zawiera trzy kopie tego genu, z których żadna sama w sobie nie jest niezbędna. Jednak potrójna delecja wszystkich trzech genów jest śmiertelna. W takich przypadkach istotność genu lub grupy paralogów można często przewidzieć na podstawie istotności podstawowego pojedynczego genu u innego gatunku. W drożdżach kilka niezbędnych genów jest zduplikowanych w genomie: 8,5% genów nieistotnych, ale tylko 1% genów niezbędnych ma homolog w genomie drożdży.
U robaka C. elegans geny nieistotne są wysoce nadreprezentowane wśród duplikatów, prawdopodobnie dlatego, że duplikacja genów istotnych powoduje nadekspresję tych genów. Woods i in. odkryli, że nieistotne geny są częściej z powodzeniem duplikowane (naprawiane) i tracone w porównaniu z genami niezbędnymi. W przeciwieństwie do tego, podstawowe geny są rzadziej powielane, ale po udanej duplikacji są utrzymywane przez dłuższe okresy.
Ochrona
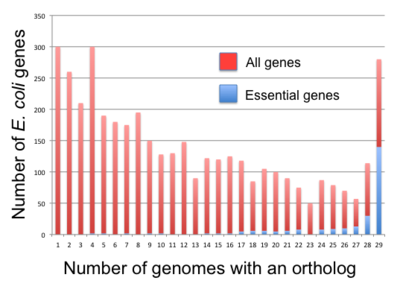
U bakterii geny niezbędne wydają się być bardziej konserwatywne niż geny nieistotne, ale korelacja nie jest bardzo silna. Na przykład tylko 34% B. subtilis ma wiarygodne ortologi we wszystkich Bacillota , a 61% niezbędnych genów E. coli ma wiarygodne ortologi we wszystkich proteobakteriach Gamma . Fang i in. (2005) zdefiniowali trwałe geny jako geny obecne w ponad 85% genomów kladu. Znaleźli 475 i 611 takich genów odpowiednio dla B. subtilis i E. coli . Ponadto sklasyfikowali geny na pięć klas według trwałości i istotności: geny trwałe, geny niezbędne, geny trwałe nieistotne (PNE) (276 w B. subtilis , 409 w E. coli ), geny niezbędne nietrwałe (ENP) (73 w B. subtilis , 33 w E. coli ) i nietrwałe geny nieistotne (NPNE) (3558 w B. subtilis , 3525 w E. coli ). Fang i in. znaleźli 257 trwałych genów, które istnieją zarówno w B. subtilis (dla Bacillota), jak i E. coli (dla Gamma-proteobacteria). Spośród nich 144 (odpowiednio 139) zostało wcześniej zidentyfikowanych jako istotne w B. subtilis (odpowiednio E. coli ), a 25 (odpowiednio 18) z 257 genów nie występuje w 475 przetrwałych B. subtilis (odpowiednio 611 E. coli). geny. Wszyscy pozostali członkowie puli to geny PNE.
U eukariotów 83% ortologów jeden do jednego między Schizosaccharomyces pombe i Saccharomyces cerevisiae ma zachowaną istotność, to znaczy, że są one nieistotne u obu gatunków lub niezbędne u obu gatunków. Pozostałe 17% genów jest nieistotnych u jednego gatunku, a niezbędnych u drugiego. Jest to dość niezwykłe, biorąc pod uwagę, że S. pombe dzieli od S. cerevisiae około 400 milionów lat ewolucji.
Ogólnie rzecz biorąc, wysoce konserwatywne, a zatem starsze geny (tj. geny o wcześniejszym pochodzeniu filogenetycznym) są bardziej prawdopodobne niż geny młodsze - nawet jeśli zostały zduplikowane.
Badanie
Eksperymentalne badanie podstawowych genów jest ograniczone przez fakt, że z definicji inaktywacja podstawowego genu jest śmiertelna dla organizmu. Dlatego nie można ich po prostu usunąć lub zmutować w celu analizy powstałych fenotypów (powszechna technika w genetyce ).
Istnieją jednak pewne okoliczności, w których można manipulować istotnymi genami. W diploidalnych może być potrzebna tylko pojedyncza funkcjonalna kopia niektórych niezbędnych genów ( haplosufficiency ), przy czym heterozygota wykazuje pouczający fenotyp. Niektóre podstawowe geny mogą tolerować mutacje, które są szkodliwe, ale nie całkowicie śmiertelne, ponieważ nie eliminują one całkowicie funkcji genu.
Analiza obliczeniowa może ujawnić wiele właściwości białek bez przeprowadzania ich eksperymentalnej analizy, np. patrząc na homologiczne białka , funkcje, strukturę itp. (patrz również poniżej, Przewidywanie niezbędnych genów ). Produkty niezbędnych genów można również badać, gdy ulegają ekspresji w innych organizmach lub gdy są oczyszczone i badane in vitro .
Warunkowo istotne geny są łatwiejsze do zbadania. Zidentyfikowano wrażliwe na temperaturę warianty niezbędnych genów, które kodują produkty, które tracą funkcję w wysokich temperaturach, a zatem wykazują fenotyp tylko w podwyższonej temperaturze.
Powtarzalność
Jeśli badania przesiewowe pod kątem niezbędnych genów są powtarzane w niezależnych laboratoriach, często skutkują różnymi listami genów. Na przykład badania przesiewowe w E. coli dały od ~300 do ~600 niezbędnych genów (patrz Tabela 1 ). Takie różnice są jeszcze bardziej wyraźne, gdy stosuje się różne szczepy bakteryjne (patrz Figura 2 ). Powszechnym wyjaśnieniem jest to, że warunki doświadczalne są różne lub że natura mutacji może być inna (np. całkowita delecja genu vs. mutant transpozonowy). W szczególności ekrany transpozonowe są trudne do odtworzenia, biorąc pod uwagę, że transpozon może zostać wstawiony w wielu pozycjach w obrębie genu. Insercje w kierunku końca 3' podstawowego genu mogą nie mieć śmiertelnego fenotypu (lub w ogóle nie mieć fenotypu), a zatem mogą nie być rozpoznawane jako takie. Może to prowadzić do błędnych adnotacji (tutaj: fałszywych negatywów).
Porównanie ekranów CRISPR /cas9 i RNAi . Badania przesiewowe mające na celu identyfikację podstawowych genów w linii komórkowej K562 ludzkiej przewlekłej białaczki szpikowej za pomocą tych dwóch metod wykazały jedynie ograniczone nakładanie się. Przy 10% wskaźniku wyników fałszywie dodatnich na ekranie Cas9 zidentyfikowano ~ 4500 genów w porównaniu z ~ 3100 na ekranie shRNA , przy czym tylko ~ 1200 genów zidentyfikowano w obu.
Różne niezbędne geny w różnych organizmach
Różne organizmy mogą mieć różne podstawowe geny. Na przykład Bacillus subtilis ma 271 niezbędnych genów. Około połowa (150) genów ortologicznych w E. coli jest również niezbędna. Kolejnych 67 genów, które są niezbędne w E. coli, nie jest niezbędnych w B. subtilis , podczas gdy 86 niezbędnych genów E. coli nie ma ortologów B. subtilis . W Mycoplasma genitalium niezbędnych jest co najmniej 18 genów, które nie są niezbędne w M. bovis. Wiele z tych różnych niezbędnych genów jest spowodowanych przez paralogi lub alternatywne szlaki metaboliczne.
Takie różne niezbędne geny w bakteriach można wykorzystać do opracowania ukierunkowanych terapii przeciwbakteryjnych przeciwko niektórym określonym patogenom w celu zmniejszenia oporności na antybiotyki w erze mikrobiomu. Stone i wsp. (2015) wykorzystali różnice w podstawowych genach bakterii do opracowania selektywnych leków przeciwko patogenowi jamy ustnej Porphyromonas gingivalis , zamiast pożytecznych bakterii Streptococcus sanguis .
Prognoza
Niezbędne geny można przewidzieć obliczeniowo. Jednak większość metod w pewnym stopniu wykorzystuje dane eksperymentalne („zbiory treningowe”). Chen i in. określili cztery kryteria wyboru zestawów treningowych do takich prognoz: (1) istotne geny w wybranym zestawie treningowym powinny być wiarygodne; (2) warunki wzrostu, w których definiowane są podstawowe geny, powinny być spójne w zestawach uczących i prognozujących; (3) gatunki wykorzystywane jako zbiór uczący powinny być blisko spokrewnione z organizmem docelowym; oraz (4) organizmy wykorzystywane jako zestawy treningowe i prognostyczne powinny wykazywać podobne fenotypy lub style życia. Odkryli również, że rozmiar zestawu uczącego powinien wynosić co najmniej 10% wszystkich genów, aby uzyskać dokładne przewidywania. Niektóre podejścia do przewidywania niezbędnych genów to:
Genomika porównawcza . Wkrótce po udostępnieniu pierwszych genomów ( Haemophilus influenzae i Mycoplasma genitalium ) Mushegian i in. próbowali przewidzieć liczbę niezbędnych genów na podstawie wspólnych genów u tych dwóch gatunków. Przypuszczano, że tylko niezbędne geny powinny być konserwowane na długim dystansie ewolucyjnym, który dzielił te dwie bakterie. W badaniu tym zidentyfikowano około 250 kandydujących genów niezbędnych. W miarę jak dostępnych było coraz więcej genomów, liczba przewidywanych niezbędnych genów stale się zmniejszała, ponieważ coraz więcej genomów dzieliło coraz mniej genów. W konsekwencji stwierdzono, że uniwersalny konserwowany rdzeń składa się z mniej niż 40 genów. Jednak ten zestaw konserwatywnych genów nie jest identyczny z zestawem niezbędnych genów, ponieważ różne gatunki polegają na różnych niezbędnych genach.
Podobne podejście zastosowano do wywnioskowania niezbędnych genów z całego genomu gatunków Brucella . Wykorzystano 42 kompletne Brucelli i łącznie 132 143 genów kodujących białka, aby przewidzieć 1252 potencjalnie niezbędnych genów, uzyskanych z genomu rdzenia przez porównanie z prokariotyczną bazą danych niezbędnych genów.
Analiza sieci . Po opublikowaniu pierwszych sieci interakcji białek drożdży odkryto, że białka silnie połączone (np. poprzez interakcje białko-białko ) są bardziej prawdopodobne. Jednak wysoce połączone białka mogą być artefaktami eksperymentalnymi, a wysoka łączność może raczej reprezentować plejotropię zamiast niezbędności. Niemniej jednak metody sieciowe zostały ulepszone poprzez dodanie innych kryteriów i dlatego mają pewną wartość w przewidywaniu niezbędnych genów.
Uczenie maszynowe . Hua i in. wykorzystali uczenie maszynowe do przewidywania istotnych genów u 25 gatunków bakterii .
Indeks Hursta . Liu i in. (2015) wykorzystali wykładnik Hursta , charakterystyczny parametr do opisania korelacji dalekiego zasięgu w DNA w celu przewidywania istotnych genów. W 31 z 33 genomów bakteryjnych poziomy istotności wykładników Hursta niezbędnych genów były znacznie wyższe niż dla odpowiedniego pełnego zestawu genów, podczas gdy poziomy istotności wykładników Hursta genów nieistotnych pozostały niezmienione lub wzrosły tylko nieznacznie.
Minimalne genomy . Uważano również, że niezbędne geny można wywnioskować z minimalnych genomów , które rzekomo zawierają tylko niezbędne geny. Problem polega na tym, że najmniejsze genomy należą do gatunków pasożytniczych (lub symbiontycznych), które mogą przetrwać ze zredukowanym zestawem genów, ponieważ uzyskują wiele składników odżywczych od swoich żywicieli. Na przykład jednym z najmniejszych genomów jest Hodgkinia cicadicola , symbiont cykad, zawierający tylko 144 kb DNA kodującego tylko 188 genów. Podobnie jak inne symbionty, Hodgkinia otrzymuje wiele składników odżywczych od gospodarza, więc jego geny nie muszą być niezbędne.
Modelowanie metaboliczne . Niezbędne geny można również przewidzieć w całkowicie zsekwencjonowanych genomach poprzez rekonstrukcję metaboliczną , to znaczy poprzez rekonstrukcję pełnego metabolizmu z zawartości genów, a następnie identyfikację tych genów i szlaków, które okazały się istotne u innych gatunków. Jednak ta metoda może być zagrożona przez białka o nieznanej funkcji. Ponadto wiele organizmów ma ścieżki zapasowe lub alternatywne, które należy wziąć pod uwagę (patrz ryc. 1). Modelowanie metaboliczne zostało również wykorzystane przez Baslera (2015) do opracowania metody przewidywania istotnych genów metabolicznych. Analiza bilansu przepływu , metoda modelowania metabolicznego, została ostatnio wykorzystana do przewidywania genów niezbędnych w metabolizmie jasnokomórkowego raka nerkowokomórkowego.
Geny o nieznanej funkcji . Co zaskakujące, znaczna liczba niezbędnych genów nie ma żadnej znanej funkcji. Na przykład wśród 385 niezbędnych kandydatów u M. genitalium żadnej funkcji nie można było przypisać 95 genom, mimo że do 2011 r. liczba ta została zmniejszona do 75. Większość nieznanych funkcjonalnie niezbędnych genów ma potencjalne funkcje biologiczne związane z jednym z trzech podstawowych genów Funkcje.
ZUPLE . Piosenka i in. przedstawił nową metodę przewidywania istotnych genów, która wykorzystuje tylko krzywą Z i inne cechy oparte na sekwencji. Takie cechy można łatwo obliczyć z sekwencji DNA/aminokwasów. Jednak niezawodność tej metody pozostaje nieco niejasna.
Podstawowe serwery przewidywania genów . Guo i in. (2015) opracowali trzy usługi online do przewidywania niezbędnych genów w genomach bakteryjnych. Te swobodnie dostępne narzędzia mają zastosowanie do pojedynczych sekwencji genów bez przypisanych funkcji, pojedynczych genów o określonych nazwach i pełnych genomów szczepów bakteryjnych. Kong i in. (2019) opracowali ePath , której można użyć do wyszukiwania > 4000 gatunków bakterii w celu przewidywania niezbędnych genów.
Niezbędne domeny białkowe
Chociaż większość niezbędnych genów koduje białka, wiele niezbędnych białek składa się z jednej domeny. Fakt ten został wykorzystany do zidentyfikowania niezbędnych domen białkowych. Goodacre i in. zidentyfikowali setki podstawowych domen o nieznanej funkcji (eDUF). Lu i in. przedstawili podobne podejście i zidentyfikowali 3450 domen, które są niezbędne w co najmniej jednym gatunku drobnoustrojów.
Zobacz też
Dalsza lektura
- Gao F, Luo H, Zhang CT, Zhang R (2015). „Analiza istotności genów oparta na DEG 10, zaktualizowanej bazie danych niezbędnych genów”. Niezbędność genów . Metody w biologii molekularnej . Tom. 1279. s. 219–33. doi : 10.1007/978-1-4939-2398-4_14 . ISBN 978-1-4939-2397-7 . PMID 25636622 .
- Długi JL, wyd. (2015). Niezbędność genów - metody i protokoły Springera . Metody w biologii molekularnej . Tom. 1279. Humana Press. P. 248. doi : 10.1007/978-1-4939-2398-4 . ISBN 978-1-4939-2397-7 . S2CID 27547825 .
- Zhang R., wyd. (2022). Niezbędne geny i genomy . Metody w biologii molekularnej . Tom. 2377. Humana Press. P. 434. doi : 10.1007/978-1-0716-1720-5 . ISBN 978-1-0716-1719-9 . S2CID 240006552 .
Linki zewnętrzne
- Baza danych niezbędnych genów
- OGEE: Internetowa baza danych Essentiality
- Baza danych EGGS (Essential Genes on Genome Scale).
- Baza danych ePath (niezbędne geny w ścieżce).
- Niezbędne geny w E. coli (EcoliWiki )
- Niezbędne geny w E. coli (Ecogene)
- Podstawowe geny Benjamina Lewina (podręcznik), Pearson/Prentice-Hall .