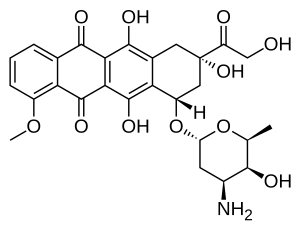
Biosynteza doksorubicyny
Doksorubicyna (DXR) jest 14- hydroksylowaną wersją daunorubicyny , bezpośredniego prekursora DXR w jej szlaku biosyntezy . Daunorubicyna występuje częściej jako produkt naturalny , ponieważ jest wytwarzana przez wiele różnych dzikich szczepów streptomyces . W przeciwieństwie do tego, początkowo stwierdzono, że tylko jeden znany gatunek inny niż dziki , streptomyces peucetius podgatunek caesius ATCC 27952, jest zdolny do wytwarzania szerzej stosowanej doksorubicyny. Szczep ten został stworzony przez Arcamone et al. w 1969 roku poprzez mutację szczepu produkującego daunorubicynę, ale nie DXR , przynajmniej w wykrywalnych ilościach. Następnie grupa Hutchinsona wykazała, że w specjalnych warunkach środowiskowych lub poprzez wprowadzenie modyfikacji genetycznych inne szczepy streptomyces mogą wytwarzać doksorubicynę. Jego grupa sklonowała również wiele genów wymaganych do produkcji DXR, chociaż nie wszystkie z nich zostały w pełni scharakteryzowane. W 1996 roku grupa Strohla odkryła, wyizolowała i scharakteryzowała dox A, gen kodujący enzym , który przekształca daunorubicynę w DXR. Do 1999 roku wyprodukowali rekombinowaną Dox A, oksydazę cytochromu P450 i odkryli, że katalizuje ona wiele etapów biosyntezy DXR , w tym etapy prowadzące do daunorubicyny. Było to istotne, ponieważ stało się jasne, że wszystkie szczepy produkujące daunorubicynę mają geny niezbędne do wytwarzania DXR, o wiele ważniejszego terapeutycznie z tych dwóch. Grupa Hutchinsona opracowała metody poprawy wydajności DXR, z fermentacji stosowanego w jego komercyjnej produkcji, nie tylko poprzez wprowadzenie plazmidów kodujących Dox A , ale także poprzez wprowadzenie mutacji w celu dezaktywacji enzymów , które przesuwają prekursory DXR do mniej użytecznych produktów, na przykład glikozydy podobne do baumycyny . Niektóre potrójne mutanty, które również nadeksprymowały Dox A, były w stanie podwoić wydajność DXR. Jest to coś więcej niż zainteresowanie akademickie, ponieważ w tamtym czasie DXR kosztował około 1,37 miliona dolarów za kg, a obecna produkcja w 1999 roku wynosiła 225 kg rocznie. Bardziej wydajne techniki produkcji obniżyły cenę nieliposomalnego do 1,1 miliona dolarów za kg . Chociaż DXR można wytwarzać półsyntetycznie z daunorubicyny, proces ten obejmuje bromowanie elektrofilowe i wiele etapów, a wydajność jest niska. Ponieważ daunorubicyna jest wytwarzana w procesie fermentacji , byłoby idealnie, gdyby bakterie mogły skuteczniej zakończyć syntezę DXR.
Przegląd
Szkielet antracyklinowy doksorubicyny (DXR) jest wytwarzany przez syntazę poliketydową typu II (PKS) w streptomyces peucetius . Najpierw syntetyzowany jest 21-węglowy łańcuch dekektydowy (ryc. 1. (1)) z pojedynczej 3-węglowej grupy propionylowej z propionylo-CoA i 9 jednostek 2-węglowych pochodzących z 9 kolejnych ( iteracyjnych ) dekarboksylacyjnych kondensacji malonylo -CoA . Każda malonylo-CoA wnosi 2-węglową jednostkę ketidową do rosnącego łańcucha poliketydowego. Każda addycja jest katalizowana przez „minimalną PKS” składającą się z acylowego białka nośnikowego (ACP), heterodimeru ketosyntazy (KS)/czynnika długości łańcucha (CLF) i acylotransferazy malonylo-Coa:ACP (MAT). (patrz górna część rysunku 10.
Proces ten jest bardzo podobny do syntezy kwasów tłuszczowych przez syntazy kwasów tłuszczowych i do syntezy poliketydów typu I. Ale w przeciwieństwie do syntezy kwasów tłuszczowych , grupy ketonowe rosnącego łańcucha poliketydowego nie są modyfikowane podczas wydłużania łańcucha i zwykle nie są w pełni redukowane . W przeciwieństwie do systemów PKS typu I, syntetyczne enzymy (KS, CLF, ACP i AT) nie są połączone ze sobą kowalencyjnie i mogą nawet nie pozostawać połączone podczas każdego etapu syntezy łańcucha poliketydowego.
Po zakończeniu 21-węglowego łańcucha dekektydowego DXR dokonuje się kolejnych modyfikacji, aby ostatecznie wytworzyć tetracykliczny aglikon antracyklinowy (bez przyłączonego glikozydu ). Aminocukier daunosaminy , aktywowany przez dodanie difosforanu tiaminy (TDP) , powstaje w kolejnej serii reakcji. Łączy się z antracyklinowym i przeprowadza się dalsze modyfikacje w celu wytworzenia najpierw daunorubicyny, a następnie DXR. Istnieją co najmniej 3 klastry genów ważne dla biosyntezy DXR : geny dps , które określają enzymy wymagane do syntezy liniowego łańcucha poliketydowego i jego pierwszych cyklizacji, klaster dnr odpowiedzialny za pozostałe modyfikacje struktury antracyklin oraz geny dnm zaangażowane w aminocukier , daunosamina , synteza. Dodatkowo istnieje zestaw genów „samoodporności” w celu zmniejszenia toksycznego wpływu antracykliny na organizm produkujący . Jednym z mechanizmów jest membranowa , która powoduje wypływ DXR z komórki (drr loci ). Ponieważ te złożone cząsteczki są korzystne tylko w określonych warunkach i wymagają dużo energii do wytworzenia, ich synteza jest ściśle regulowana.
Synteza łańcucha poliketydowego
Doksorubicyna jest syntetyzowana przez wyspecjalizowaną syntazę poliketydową .
Początkowym wydarzeniem w syntezie DXR jest wybór jednostki startowej propionylo-CoA i jej dekarboksylacyjne dodanie do dwuwęglowej jednostki ketidowej, pochodzącej z malonylo-CoA, w celu wytworzenia pięciowęglowego B-ketowalerylu ACP. Pięciowęglowy diketyd jest dostarczany przez ACP do grupy sulfhydrylowej cysteiny w miejscu aktywnym KS, poprzez wymianę tioestru , a ACP jest uwalniany z łańcucha. Wolny ACP odbiera kolejną grupę malonianową z malonylo-CoA , również przez wymianę tioestrową , z uwolnieniem CoA .
ACP przenosi nowy malonian do miejsca aktywnego KS, gdzie jest on dekarboksylowany , prawdopodobnie za pomocą podjednostki CLF i łączony, tworząc triketyd o 7 atomach węgla, teraz zakotwiczony w ACP (patrz górna część ryciny 1). Ponownie ACP przekazuje łańcuch podjednostce KS i proces jest powtarzany iteracyjnie, aż do zakończenia dekektydu.
W większości układów typu II zdarzeniem inicjującym jest dostarczenie przez ACP jednostki octanowej , pochodzącej z acetylo-CoA , do miejsca aktywnego podjednostki ketosyntazy (KS) heterodimeru KS/CLF. Domyślnym trybem dla systemów PKS typu II jest włączenie octanu jako jednostki podkładu i dotyczy to „minimalnego PKS” DXR. Innymi słowy, działanie KS/CLF/ACP (Dps A, B i G) z tego układu nie spowoduje wytworzenia 21- węglowych dekektydów, ale zamiast nich 20-węglowych dekekitydów, ponieważ octan jest „preferowanym” starterem. Proces określania propionianu nie jest w pełni zrozumiały, ale jasne jest, że zależy on od dodatkowego białka, Dps C, które może działać jako ketosyntaza lub acylotransferaza selektywna dla propionylo-CoA, i prawdopodobnie Dps D ma swój udział.
Stwierdzono, że dedykowana MAT jest zbędna do produkcji poliketydu w warunkach in vitro. PKS może „pożyczyć” MAT od własnej syntazy kwasów tłuszczowych i może to być główny sposób, w jaki ACP otrzymuje swoją grupę malonianową w biosyntezie DXR. Ponadto istnieją doskonałe dowody na to, że „samomalonylacja” jest nieodłączną cechą AKP typu II. Podsumowując, dany PKS Typu II może zapewnić własny MAT (y), może pożyczyć go od FAS lub jego ACP może „samo-malonylować”.
Nie wiadomo, czy ten sam trójskładnikowy kompleks KS/CLF/ACP jest opiekunem wzrostu pełnej długości łańcucha poliketydowego przez cały cykl katalityczny, czy też ACP dysocjuje po każdej reakcji kondensacji. Struktura aktynorodyny KS/CLF o rozdzielczości 2,0 Å , która jest bardzo podobna do dps KS/CLF, pokazuje wydłużenie poliketydów wewnątrz amfipatycznego tunelu utworzonego na styku podjednostek KS i CLF. Tunel ma długość około 17 Å , a po jednej stronie znajduje się wiele naładowanych reszt aminokwasowych , które wydają się stabilizować grupy karbonylowe łańcucha, podczas gdy druga strona jest hydrofobowa . Ta struktura wyjaśnia, dlaczego obie podjednostki są niezbędne do wydłużenia łańcucha i jak reaktywny rosnący łańcuch jest chroniony przed przypadkowymi reakcjami spontanicznymi , dopóki nie zostanie odpowiednio ustawiony do uporządkowanej cyklizacji. Struktura sugeruje również mechanizm regulacji długości łańcucha. aminokwasów rozciągają się do tunelu i działają jak „bramy”. Kilka szczególnie dużych reszt może być nieprzepuszczalnych dla łańcucha, powodując terminację. Modyfikacje pozostałości tuneli oparte na tej strukturze były w stanie zmienić długość łańcucha produktu końcowego. Końcowa kondensacja powoduje, że łańcuch poliketydowy „zapina się”, umożliwiając wewnątrzcząsteczkowy atak karboanionu metylenu C-12 , generowanego przez katalizowane przez enzym usuwanie protonów i stabilizowane przez oddziaływania elektrostatyczne w tunelu, na karbonyl C-7 (patrz 3 na ryc. 1 ). Ta wewnątrzcząsteczkowa kondensacja aldolowa wspomagana przez tunel zapewnia pierwszą cyklizację, gdy łańcuch nadal znajduje się w tunelu. Ten sam atak C-7/C-12 występuje w biosyntezie DXR , w podobny sposób.
Konwersja do kwasu 12-dezoksyalkalonowego
21-węglowy deketyd jest przekształcany w kwas 12-dezoksyalkalonowy (5), pierwszy wolny, łatwy do wyizolowania związek pośredni w biosyntezie DXR, w 3 etapach. Te etapy są katalizowane przez ostatnie 3 enzymy w klastrze genów dps i są uważane za część syntazy poliketydowej .
Podczas gdy dekektyd jest nadal związany z heterodimerem KS/CLF, grupa 9- karbonylowa jest redukowana przez Dps E, 9-ketoreduktazę, przy użyciu NADPH jako środka redukującego / donora wodorków . Dps F, „ cyklaza pierwszego pierścienia ” /aromataza, jest bardzo specyficzna i należy do rodziny cyklaz C-7/C-12, które wymagają uprzedniej redukcji keto-C-9. Uważa się, że te dwie reakcje zachodzą, gdy łańcuch poliketydowy jest nadal częściowo w tunelu KS / CLF i nie wiadomo, co ostatecznie rozszczepia łańcuch od jego wiązania kowalencyjnego do KS lub ACP. cyklaza Dps F jest inaktywowana przez mutacje lub delecje genów , łańcuch będzie ulegał spontanicznej cyklizacji w losowy sposób. Tak więc uważa się, że Dps F „opiekuje się” lub pomaga fałdować poliketyd, aby zapewnić nielosową cyklizację, reakcję, która jest energetycznie korzystna i prowadzi do późniejszego odwodnienia i wynikającej z tego aromatyzacji .
Następnie Dps Y regioselektywnie promuje tworzenie kolejnych dwóch wiązań węgiel-węgiel , a następnie katalizuje odwodnienie prowadzące do aromatyzacji jednego z pierścieni dając (5).
Konwersja do ε-rodomycynonu
Kolejne reakcje są katalizowane przez enzymy pochodzące z klastra genów dnr . Dnr G, oksygenaza C-12 (patrz numeracja (5)) wprowadza grupę ketonową przy użyciu tlenu cząsteczkowego . Jest to „ oksygenaza typu antronowego ”, zwana także monooksygenazą tworzącą chinony , z których wiele jest ważnymi „ enzymami dopasowującymi ” w biosyntezie kilku rodzajów aromatycznych antybiotyków poliketydowych . Nie mają kofaktorów : flawin , metali ani źródeł energii. Ich mechanizm jest słabo poznany, ale może obejmować „ rodnik białkowy ”.
Produktem jest kwas alkaliczny (6), chinon . Dnr C, O- metylotransferaza kwasu alkalonowego metyluje koniec kwasu karboksylowego cząsteczki , tworząc ester , wykorzystując S-adenozylometioninę (SAM) jako kofaktor / donor grupy metylowej . Produktem jest ester metylowy kwasu alkalicznego (7). Grupa metylowa jest usuwana później, ale służy do aktywacji sąsiedniego mostka metylenowego, ułatwiając jego atak na końcową grupę karbonylową , reakcję katalizowaną przez DnrD.
Dnr D, cyklaza czwartego pierścienia (cyklaza AAME), katalizuje wewnątrzcząsteczkową reakcję addycji aldolowej . Nie są wymagane żadne kofaktory i nie dochodzi do aromatyzacji ani odwodnienia. Zaproponowano prosty mechanizm katalizowany zasadą . Produktem jest aklaviketone (8).
reduktaza aklawiketonu , stereospecyficznie redukuje grupę 17-keto nowego czwartego pierścienia do grupy 17-OH, dając aklawinon (9). Wprowadza to nowe chiralne , a NADPH jest kofaktorem.
Dnr F, aklavinone-11- hydroksylaza , jest monooksygenazą FAD , która wykorzystuje NADPH do aktywacji tlenu cząsteczkowego w celu późniejszej hydroksylacji . Produktem jest ε-rodomycynon (10).
Konwersja do doksorubicyny
Dnr S, glikozylotransferaza daunosaminy katalizuje dodanie glikozydu aktywowanego TDP , L- daunosaminy - TDP do ε-rodomycynonu, dając rodomycynę D (ryc. 2). Uwolnienie TDP napędza reakcję do przodu. Enzym wykazuje podobieństwo sekwencji do glikozylotransferaz innych „niezwykłych cukrów” dodawanych do produktów aromatycznych typu II PKS .
Dnr P, metyloesteraza rodomycyny D , usuwa grupę metylową dodaną wcześniej przez DnrC. Początkowo służył do aktywacji sąsiedniego mostka metylenowego, a następnie zapobiegał opuszczeniu przez jego grupę karboksylową węgla C-10 (patrz ryc. 2). Gdyby grupa karboksylowa nie została CO2 zestryfikowana przed cyklizacją czwartego pierścienia, jej odejściu jako sprzyjałoby utworzenie bicyklicznego układu aromatycznego . Po redukcji C-7 i glikozylacji mostek metylenowy C-8 nie jest już aktywowany do deprotonowania , co zmniejsza prawdopodobieństwo aromatyzacji. Zauważ, że nieizolowalny związek pośredni , z numeracją, jest trzecią cząsteczką na rycinie 2. System numeracji jest bardzo dziwny i stanowi pozostałość po wczesnym nazewnictwie. Dekarboksylacja związku pośredniego zachodzi spontanicznie lub pod wpływem Dnr P, dając 13-deoksykarminomycynę .
Struktura krystaliczna , z produktami związanymi, metyloesterazy aklacinomycyny , [enzymu] o 53% homologii sekwencji do Dnr P, ze Streptomyces purpurascens , została rozwiązana. Jest w stanie katalizować tę samą reakcję i wykorzystuje klasyczną triadę katalityczną Ser - His - Asp z seryną pełniącą rolę nukleofila i gly - met zapewniającą stabilizację stanu przejściowego poprzez tworzenie „ dziury oksyanionowej ”. Aminokwasy miejsca aktywnego są prawie całkowicie takie same jak Dnr P, a mechanizm jest prawie na pewno identyczny. Chociaż Dox A jest pokazany jako następny na schemacie biosyntetycznym (ryc. 2), Dnr K, 4-O- metylotransferaza karminomycyny jest zdolna do O- metylacji grupy 4- hydroksylowej dowolnego z glikozydów na ryc. 2. Struktura krystaliczna o rozdzielczości 2,35 Å enzymu z produktami związanymi został niedawno rozwiązany . Orientacja produktów jest zgodna z mechanizmem przenoszenia metylu SN2 . Ukierunkowana mutageneza potencjalnych reszt kwasowo-zasadowych w miejscu aktywnym nie wpłynęła na katalizę , co doprowadziło do wniosku, że Dnr K najprawdopodobniej działa jako enzym entropiczny , ponieważ zwiększenie szybkości wynika głównie z efektów orientacji i bliskości. Kontrastuje to z większością innych O-metylotransferaz, w których wykazano, że kataliza kwasowo-zasadowa jest istotnym wkładem w zwiększenie szybkości. Dox A katalizuje trzy kolejne utleniania w streptomyces peucetius . Niedobór produkcji DXR nie wynika głównie z niskiego poziomu lub nieprawidłowego działania Dox A, ale z faktu, że istnieje wiele produktów odwróconych od szlaku pokazanego na rycinie 2. Każdy z glikozydów jest potencjalnym celem enzymów bocznikowych, których nie pokazano, z których niektóre są produktami klastra genów dnr . Mutacje tych enzymów znacznie zwiększają produkcję DXR. Ponadto Dox A ma bardzo niską kcat/Km dla utleniania C-14 (130/M) w porównaniu z utlenianiem C-13 (do 22 000/M dla niektórych substratów). Manipulacja genetyczna mająca na celu nadekspresję Dox A również zwiększyła wydajność, szczególnie jeśli geny enzymów bocznikowych są jednocześnie inaktywowane. Dox A jest monooksygenazą cytochromu P-450 , która ma szeroką specyficzność substratową , katalizującą hydroksylację antracykliny na C-13 i C-14 (ryc. 2). Enzym ma bezwzględne zapotrzebowanie na tlen cząsteczkowy i NADPH . Początkowo przeprowadza się dwa kolejne utleniania przy C-13, a następnie pojedyncze utlenianie C-14, które przekształca daunorubicynę w doksorubicynę.
- ^ a b c Lomovskaya N, Otten SL, Doi-Katayama Y i in. (1999). „Nadprodukcja doksorubicyny w Streptomyces peucetius: klonowanie i charakterystyka genów dnrU ketoreduktazy i dnrV oraz genu hydroksylazy cytochromu P-450 doxA” . J. Bacteriol . 181 (1): 305–18. doi : 10.1128/JB.181.1.305-318.1999 . PMC 103563 . PMID 9864344 .
- ^ Arcamone F, Cassinelli G, Fantini G i in. (1969). „Adriamycyna, 14-hydroksydaunomycyna, nowy antybiotyk przeciwnowotworowy z S. peucetius var. Caesius”. Biotechnologia. Bioeng . 11 (6): 1101–10. doi : 10.1002/bit.260110607 . PMID 5365804 .
- ^ Grimm A, Madduri K, Ali A, Hutchinson CR (1994). „Charakterystyka genów Streptomyces peucetius ATCC 29050 kodujących syntazę poliketydową doksorubicyny”. gen . 151 (1–2): 1–10. doi : 10.1016/0378-1119(94)90625-4 . PMID 7828855 .
- ^ Dickens ML, Strohl WR (1996). „Izolacja i charakterystyka genu ze Streptomyces sp. szczep C5, który nadaje zdolność przekształcania daunomycyny w doksorubicynę na Streptomyces lividans TK24” . J. Bacteriol . 178 (11): 3389–95. doi : 10.1128/jb.178.11.3389-3395.1996 . PMC 178102 . PMID 8655530 .
- ^ a b Walczak RJ, Dickens ML, Priestley ND, Strohl WR (1999). „Oczyszczanie, właściwości i charakterystyka rekombinowanych Streptomyces sp. Szczep C5 DoxA, cytochrom P-450 katalizujący wiele etapów biosyntezy doksorubicyny” . J. Bacteriol . 181 (1): 298–304. doi : 10.1128/JB.181.1.298-304.1999 . PMC 103562 . PMID 9864343 .
- ^ Hutchinson CR, Colombo AL (1999). „Inżynieria genetyczna produkcji doksorubicyny w Streptomyces peucetius: przegląd”. J. Ind. Microbiol. Biotechnologia . 23 (1): 647–52. doi : 10.1038/sj.jim.2900673 . PMID 10455495 .
- ^ Lown JW (1993). „Antracykliny i antrachinonowe środki przeciwnowotworowe: aktualny stan i ostatnie osiągnięcia”. Farmakol. Tam . 60 (2): 185–214. doi : 10.1016/0163-7258(93)90006-Y . PMID 8022857 .
- ^ Hutchinson CR (1997). „Badania biosyntetyczne daunorubicyny i tetracenomycyny C”. Recenzje chemiczne . 97 (7): 2525–2536. doi : 10.1021/cr960022x . PMID 11851469 .
- ^ Otten SL, Gallo MA, Madduri K, Liu X, Hutchinson CR (1997). „Klonowanie i charakterystyka genów Streptomyces peucetius dnmZUV kodujących trzy enzymy wymagane do biosyntezy prekursora daunorubicyny tymidynodifosfo-L-daunosaminy” . J. Bacteriol . 179 (13): 4446–50. PMC 179277 . PMID 9209071 .
- ^ Dickens ML, Priestley ND, Strohl WR (1997). „Biokonwersja in vivo i in vitro glikozydu epsilon-rodomycynonu do doksorubicyny: funkcje DauP, DauK i DoxA” . J. Bacteriol . 179 (8): 2641–50. PMC 179014 . PMID 9098063 .
- ^ Gandlur SM, Wei L, Levine J, Russell J, Kaur P (2004). „Topologia błony białka DrrB transportera doksorubicyny Streptomyces peucetius” . J. Biol. chemia . 279 (26): 27799–806. doi : 10.1074/jbc.M402898200 . PMID 15090538 .
- ^ Jiang H, Hutchinson CR (2006). „Regulacja sprzężenia zwrotnego biosyntezy doksorubicyny u Streptomyces peucetius”. Rez. Mikrobiol . 157 (7): 666–74. doi : 10.1016/j.resmic.2006.02.004 . PMID 16545946 .
- ^ Bao W, Sheldon PJ, Hutchinson CR (1999). „Oczyszczanie i właściwości Streptomyces peucetius DpsC beta-ketoacylo: acylowa syntaza białka nośnikowego III, która określa jednostkę propionian-starter dla biosyntezy poliketydów typu II”. Biochemia . 38 (30): 9752-7. doi : 10.1021/bi990751h . PMID 10423255 .
- ^ Bao W, Sheldon PJ, Wendt-Pienkowski E, Hutchinson CR (1999). „Gen Streptomyces peucetius dpsC determinuje wybór jednostki startowej w biosyntezie poliketydu daunorubicyny” . J. Bacteriol . 181 (15): 4690-5. PMC 103607 . PMID 10419974 .
- ^ Matharu AL, Cox RJ, Crosby J, Byrom KJ, Simpson TJ (1998). „MCAT nie jest wymagane do syntezy poliketydów in vitro w minimalnej syntazie poliketydowej aktynorodyny ze Streptomyces coelicolor” . chemia Biol . 5 (12): 699–711. doi : 10.1016/S1074-5521(98)90663-9 . PMID 9862793 .
- ^ Arthur CJ, Szafrańska A, Evans SE i in. (2005). „Samomalonylacja jest nieodłączną właściwością chemicznie syntetyzowanego białka nośnikowego syntazy poliketydowej typu II”. Biochemia . 44 (46): 15414–21. doi : 10.1021/bi051499i . PMID 16285746 .
- ^ Dreier J, Khosla C (2000). „Analiza mechanistyczna syntazy poliketydowej typu II. Rola konserwowanych reszt w heterodimerze czynnika długości łańcucha syntazy beta-ketoacylowej”. Biochemia . 39 (8): 2088–95. doi : 10.1021/bi992121l . PMID 10684659 .
- ^ Keatinge-Clay AT, Maltby DA, Medzihradszky KF, Khosla C, Stroud RM (2004). „Fabryka antybiotyków złapana w akcji” . Nat. Struktura. Mol. Biol . 11 (9): 888–93. doi : 10.1038/nsmb808 . PMID 15286722 .
- ^ Tang Y, Tsai SC, Khosla C (2003). „Kontrola długości łańcucha poliketydowego przez współczynnik długości łańcucha”. J. Am. chemia soc . 125 (42): 12708–9. doi : 10.1021/ja0378759 . PMID 14558809 .
- ^ Meurer G, Gerlitz M, Wendt-Pienkowski E, Vining LC, Rohr J, Hutchinson CR (1997). „Iteracyjne syntazy poliketydowe typu II, cyklazy i ketoreduktazy wykazują zachowanie zależne od kontekstu w biosyntezie liniowych i kątowych dekapoliketydów” . chemia Biol . 4 (6): 433–43. doi : 10.1016/S1074-5521(97)90195-2 . PMID 9224566 .
- ^ Wohlert SE, Wendt-Pienkowski E, Bao W, Hutchinson CR (2001). „Produkcja aromatycznych minimalnych poliketydów przez geny syntazy poliketydowej daunorubicyny ujawnia niezgodność heterologicznych cyklaz DpsY i JadI”. J. Nat. prod . 64 (8): 1077–80. doi : 10.1021/np010067f . PMID 11520231 .
- Bibliografia _ „Oksygenazy bez wymogu kofaktorów lub jonów metali”. Aplikacja Mikrobiol. Biotechnologia . 60 (3): 243–57. doi : 10.1007/s00253-002-1123-4 . PMID 12436305 .
- ^ a b Kendrew SG, Katayama K, Deutsch E, Madduri K, Hutchinson CR (1999). „Cyklaza DnrD zaangażowana w biosyntezę doksorubicyny: oczyszczanie i charakterystyka rekombinowanego enzymu”. Biochemia . 38 (15): 4794–9. doi : 10.1021/bi9827924 . PMID 10200167 .
- ^ Niemi J, Wang Y, Airas K, Ylihonko K, Hakala J, Mäntsälä P (1999). „Charakterystyka 11-hydroksylazy aklawinonu ze Streptomyces purpurascens”. Biochim. Biofiza. Akta . 1430 (1): 57–64. doi : 10.1016/S0167-4838(98)00265-9 . PMID 10082933 .
- ^ Otten SL, Liu X, Ferguson J, Hutchinson CR (1995). „Klonowanie i charakterystyka genów Streptomyces peucetius dnrQS kodujących enzym biosyntezy daunosaminy i transferazę glikozylową zaangażowaną w biosyntezę daunorubicyny” . J. Bacteriol . 177 (22): 6688–92. PMC 177529 . PMID 7592454 .
- ^ Jansson A, Niemi J, Mäntsälä P, Schneider G (2003). „Struktura krystaliczna metyloesterazy aklacinomycyny ze związanymi analogami produktu: implikacje dla rozpoznawania i mechanizmu antracyklin” . J. Biol. chemia . 278 (40): 39006-13. doi : 10.1074/jbc.M304008200 . PMID 12878604 .
- ^ Jansson A, Koskiniemi H, Mäntsälä P, Niemi J, Schneider G (2004). „Struktura krystaliczna trójskładnikowego kompleksu DnrK, metylotransferazy w biosyntezie daunorubicyny, z produktami związanymi” . J. Biol. chemia . 279 (39): 41149–56. doi : 10.1074/jbc.M407081200 . PMID 15273252 .