Słowniczek genetyki (0–L)
Ten słowniczek genetyki to lista definicji terminów i pojęć powszechnie używanych w badaniach genetyki i pokrewnych dziedzin biologii , w tym biologii molekularnej , biologii komórki i biologii ewolucyjnej . Jest on podzielony na dwa artykuły:
- Ta strona, Glosariusz genetyki (0–L) zawiera listę terminów zaczynających się od cyfr i zaczynających się od liter od A do L.
- Glosariusz genetyki (M – Z) wymienia terminy zaczynające się na litery od M do Z.
Glosariusz jest przeznaczony jako materiał wprowadzający dla nowicjuszy; bardziej szczegółowe i techniczne szczegóły można znaleźć w artykule odpowiadającym każdemu terminowi. Aby zapoznać się z terminami pokrewnymi, zobacz Słowniczek biologii ewolucyjnej i Słowniczek chemii .
0–9
- Region nieulegający translacji 3' (3'-UTR)
- koniec 3'
- Jeden z dwóch końców pojedynczej liniowej nici DNA lub RNA , w szczególności koniec, na którym łańcuch nukleotydów kończy się na trzecim atomie węgla w pierścieniu furanozy dezoksyrybozy lub rybozy (tj. koniec, na którym węgiel 3' nie jest przyłączony z innym nukleotydem poprzez wiązanie fosfodiestrowe ; in vivo węgiel 3' jest często nadal związany z grupą hydroksylową Grupa). Zgodnie z konwencją, sekwencje i struktury znajdujące się bliżej 3'-końca w stosunku do innych są określane jako downstream . Kontrast 5'-koniec .
- czapka 5'
- Specjalnie zmieniony nukleotyd przyłączony do 5'-końca niektórych pierwotnych transkryptów RNA jako część zestawu modyfikacji potranskrypcyjnych , które przekształcają surowe transkrypty w dojrzałe produkty RNA. Dokładna struktura czapeczki 5' różni się znacznie w zależności od organizmu; u eukariontów najbardziej podstawowa czapka składa się z metylowanego nukleozydu guaniny związanego z grupą trifosforanową, która kończy 5'-koniec sekwencji RNA. Wśród innych funkcji capping pomaga regulować eksport dojrzałych RNA z jądra , zapobiegają ich degradacji przez egzonukleazy i promują translację w cytoplazmie. Dojrzałe mRNA można również zdekapować .
- Nieulegający translacji region 5' (5'-UTR)
- koniec 5'
- Jeden z dwóch końców pojedynczej liniowej nici DNA lub RNA , w szczególności koniec, na którym łańcuch nukleotydów kończy się na piątym atomie węgla w pierścieniu furanozy dezoksyrybozy lub rybozy (tj. koniec, na którym węgiel 5' nie jest przyłączony z innym nukleotydem poprzez wiązanie fosfodiestrowe ; in vivo węgiel 5' jest często nadal związany z fosforanem Grupa). Zgodnie z konwencją, sekwencje i struktury znajdujące się bliżej końca 5' w stosunku do innych są określane jako upstream . Kontrast 3'-koniec .
- 5-bromodeoksyurydyna
- Patrz bromodeoksyurydyna .
- 5-Metyluracyl
- Patrz tymina .
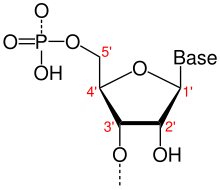
rybozy z atomami węgla ponumerowanymi od 1' do 5' zgodnie z konwencją chemiczną . Mówi się, że węgiel 5' znajduje się powyżej ; mówi się, że ' znajduje się węgiel 3 poniżej . Pokazano również wiązania z ogólną zasadą i grupą fosforanową .
A
- A-DNA
- Jedna z trzech głównych biologicznie aktywnych strukturalnych konformacji podwójnej helisy DNA , wraz z B-DNA i Z- DNA . Helisa w kształcie litery A ma prawoskrętny skręt z 11 parami zasad na pełny obrót, tylko nieco bardziej zwarta niż B-DNA, ale jej podstawy są ostro nachylone względem osi helisy. Często jest preferowany w warunkach odwodnionych iw obrębie sekwencji kolejnych purynowych (np. GAAGGGGA ); jest to również podstawowa konformacja przyjęta przez dwuniciowe hybrydy RNA i RNA-DNA .
- acentryczny (z
- chromosomu liniowego lub fragmentu chromosomu) Nie ma centromeru .
- akrocentryczny
- (liniowego chromosomu lub fragmentu chromosomu) Posiadający centromer umieszczony bardzo blisko jednego końca chromosomu, w przeciwieństwie do końca lub w środku .
- aktywacja
- Patrz regulacja w górę .
- aktywator
- Rodzaj czynnika transkrypcyjnego który zwiększa transkrypcję genu lub zestawu genów. Większość aktywatorów działa poprzez wiązanie się ze specyficzną sekwencją zlokalizowaną w obrębie lub w pobliżu wzmacniacza lub promotora i ułatwianie wiązania polimerazy RNA i innych maszyn transkrypcyjnych w tym samym regionie. Zobacz także koaktywator ; represor kontrastu .
- adenina ( A )
- Nukleozasada purynowa stosowana jako jedna z czterech standardowych zasad nukleotydowych w obu Cząsteczki DNA i RNA . Adenina tworzy parę zasad z tyminą w DNA i z uracylem w RNA.
- adenozyna ( A )
- Jeden z czterech standardowych nukleozydów stosowanych w cząsteczkach RNA , składający się z zasady adeninowej z azotem N9 związanym z atomem węgla C1 cukru rybozy . Adenina związana z dezoksyrybozą to tzw deoksyadenozyna , która jest wersją stosowaną w DNA .
- dotknięta para względna
- Każda para organizmów, które są spokrewnione genetycznie i oba dotknięte tą samą cechą . Na przykład dwóch kuzynów, którzy obaj mają niebieskie oczy, jest dotkniętą parą względną, ponieważ obaj są dotknięci allelem kodującym niebieskie oczy.
- allel
- Jedna z wielu alternatywnych wersji pojedynczego genu , z których każda jest żywotną sekwencją DNA zajmującą określoną pozycję lub locus na chromosomie . Na przykład u ludzi jeden allel genu koloru oczu daje niebieskie oczy, a inny allel genu koloru oczu - brązowe oczy.
- częstość alleli
- Względna częstość występowania określonego allelu danego genu (w przeciwieństwie do innych alleli tego samego genu) w określonym locus u członków populacji; dokładniej, jest to odsetek wszystkich chromosomów w populacji, które niosą określony allel, wyrażony jako ułamek lub procent. Częstotliwość alleli różni się od częstotliwości genotypów , chociaż są one ze sobą powiązane.
- alosom
- Dowolny chromosom , który różni się od zwykłego autosomu rozmiarem, formą lub zachowaniem i który jest odpowiedzialny za określenie płci organizmu. U ludzi chromosom X i chromosom Y to chromosomy płciowe.
- alfa helisa (α-helisa)
- Powszechny motyw strukturalny w drugorzędowych strukturach białek składający się z prawoskrętnej konformacji helisy wynikającej z wiązań wodorowych między aminokwasami pozostałości, które nie sąsiadują bezpośrednio ze sobą.
- splicing alternatywny
- Regulowane zjawisko ekspresji genów eukariotycznych , w którym określone eksony lub części eksonów z tego samego pierwotnego transkryptu są zmiennie włączane lub usuwane z końcowego, dojrzałego transkryptu informacyjnego RNA . Klasa modyfikacji potranskrypcyjnych , alternatywny splicing, umożliwia pojedynczemu genowi kodowanie wielu izoform białek i znacznie zwiększa różnorodność białek, które mogą być wytwarzane przez pojedynczy genom . Zobacz także splicing RNA .
- bursztynowy
- Jeden z trzech kodonów stop używanych w standardowym kodzie genetycznym ; w RNA jest określony przez tryplet nukleotydowy UAG . Pozostałe dwa kodony stop to ochra i opal .
- aminokwas
- Dowolny z klasy związków organicznych, których podstawowy wzór strukturalny obejmuje centralny atom węgla związany z aminowymi i karboksylowymi grupami funkcyjnymi oraz ze zmiennym łańcuchem bocznym . Spośród prawie 500 znanych aminokwasów zestaw 20 jest kodowanych przez standardowy kod genetyczny i włączanych do długich łańcuchów polimerowych jako budulec peptydów , a tym samym polipeptydów i białek . Konkretne sekwencje aminokwasów w łańcuchach polipeptydowych, które tworzą białko, są ostatecznie odpowiedzialne za określenie struktury i funkcji białka.
- koniec aminowy
- Patrz koniec N.
- aminoacylo-tRNA (aa-tRNA)
- Przenoszący RNA , z którym związany jest chemicznie pokrewny aminokwas ; tj. produkt reakcji transestryfikacji katalizowanej przez syntetazę aminoacylo-tRNA . Aminoacylo-tRNA wiążą się z miejscem aminoacylowym rybosomu podczas translacji .
- syntetaza aminoacylo-tRNA
- Dowolny zestaw enzymów, które katalizują reakcję transestryfikacji , która prowadzi do przyłączenia określonego aminokwasu (lub prekursora) do jednej z jego pokrewnych cząsteczek przenoszących RNA , tworząc aminoacylo-tRNA . Każdy z 20 różnych aminokwasów używanych w kodzie genetycznym jest rozpoznawany i przyłączany przez własny specyficzny enzym syntetazy, a większość syntetaz jest spokrewniona z kilkoma różnymi tRNA zgodnie z ich specyficznymi antykodonami .
- amplikon
- Dowolna sekwencja lub fragment DNA lub RNA będący źródłem i/lub produktem reakcji amplifikacji . Termin ten jest najczęściej używany do opisania licznych skopiowanych fragmentów, które są produktami reakcji łańcuchowej polimerazy lub reakcji łańcuchowej ligazy , chociaż może również odnosić się do sekwencji, które są naturalnie amplifikowane w genomie, np. przez duplikację genu .
- amplifikacja
- Replikacja biocząsteczki, w szczególności wytwarzanie jednej lub więcej kopii sekwencji kwasu nukleinowego , znanej jako amplikon , naturalnie (np. przez spontaniczne duplikacje ) lub sztucznie (np. przez PCR ), aw szczególności implikuje wiele powtarzających się zdarzeń replikacyjnych skutkujących tysiącami, milionami lub miliardami kopii docelowej sekwencji, o której mówi się, że jest amplifikowana .
- anafaza
- Etap mitozy i mejozy , który występuje po metafazie i przed telofazą , kiedy replikowane chromosomy są segregowane i każda z siostrzanych chromatyd są przenoszone na przeciwne strony komórki .
- opóźnienie anafazowe
- Niepowodzenie prawidłowej migracji jednej lub więcej par siostrzanych chromatyd lub homologicznych chromosomów do przeciwnych stron komórki podczas anafazy mitozy lub mejozy z powodu wadliwego aparatu wrzeciona . W konsekwencji obie komórki potomne są aneuploidalne : jednej brakuje jednego lub więcej chromosomów (tworząc monosomię ), podczas gdy drugi ma jedną lub więcej dodatkowych kopii tego samego chromosomu (tworząc polisomię ).
- aneucentryczny
- ( chromosomu liniowego lub fragmentu chromosomu) mający nieprawidłową liczbę centromerów , tj. więcej niż jeden.
- aneuploidia
- Stan komórki lub organizmu mający nieprawidłową liczbę jednego lub więcej określonych chromosomów (ale z wyłączeniem nieprawidłowej liczby kompletnych zestawów chromosomów, co zamiast tego jest znane jako euploidia ).
- wyżarzanie
- Hybrydyzacja dwóch _ jednoniciowe cząsteczki kwasu nukleinowego zawierające komplementarne sekwencje, tworzące dwuniciową cząsteczkę ze sparowanymi zasadami nukleinowymi . Termin ten jest używany w szczególności do opisania etapów niektórych technik laboratoryjnych, takich jak reakcja łańcuchowa polimerazy , w której dwuniciowe cząsteczki DNA są wielokrotnie denaturowane do pojedynczych nici przez ogrzewanie, a następnie wystawiane na działanie niższych temperatur, powodując ponowne połączenie nici ze sobą lub z podkładami uzupełniającymi . Dokładny Temperatura, w której następuje wygrzewanie, jest silnie uzależniona od długości i określonej kolejności poszczególnych pasm.
- antycypacja
- Zjawisko, w wyniku którego objawy zaburzenia genetycznego stają się widoczne (i często bardziej nasilone) w młodszym wieku u osób dotkniętych tą chorobą z każdym pokoleniem, które dziedziczy to zaburzenie.
- antykodon
- Seria trzech kolejnych nukleotydów w transferowym RNA , które uzupełniają trzy nukleotydy kodonu w transkrypcie mRNA . Podczas translacji , każdy tRNA rekrutowany do rybosomu zawiera pojedynczy tryplet antykodonu, który łączy się w parę z jednym lub kilkoma komplementarnymi kodonami z sekwencji mRNA, umożliwiając każdemu kodonowi określenie konkretnego aminokwasu , który ma zostać dodany do rosnącego łańcucha peptydowego. Antykodony zawierające inozynę na pierwszej pozycji są zdolne do łączenia się w pary z więcej niż jednym kodonem dzięki zjawisku znanemu jako parowanie wobble base .
- antykogen
- A gen który pomaga regulować wzrost komórek i hamować guzy, gdy działa prawidłowo, tak że jego brak lub nieprawidłowe działanie może skutkować niekontrolowanym wzrostem komórek i prawdopodobnie rakiem. Porównaj onkogen .
- antyrównoległość
- Orientacja dwóch nici dwuniciowego kwasu nukleinowego (i bardziej ogólnie dowolnej pary biopolimerów ), które są równoległe do siebie, ale mają przeciwny kierunek . Na przykład dwie komplementarne nici cząsteczki DNA biegną obok siebie, ale w przeciwnych kierunkach, przy czym jedna nić jest zorientowana 5' -do -3' i pozostałe 3'-do-5'.
- antysensowny
- Zobacz nić wzorcową .
- antysensowny RNA (asRNA)
- Jednoniciowa cząsteczka niekodującego RNA zawierająca sekwencję antysensowną komplementarną do nici sensownej, takiej jak matrycowy RNA , z którą łatwo hybrydyzuje , hamując w ten sposób dalszą aktywność nici sensownej (np. translację do białka). Wiele różnych klas naturalnie występujących RNA, takich jak siRNA , działa zgodnie z tą zasadą, czyniąc je silnymi tłumikami genów w różnych regulacjach genów mechanizmy. Syntetyczny antysensowny RNA znalazł również szerokie zastosowanie w nad nokautem genów oraz w praktycznych zastosowaniach, takich jak terapia antysensowna .
- sztuczna synteza genów
- Zestaw metod laboratoryjnych stosowanych w syntezie de novo genu (lub dowolnej innej sekwencji kwasu nukleinowego ) z wolnych nukleotydów , tj. bez polegania na istniejącej nici matrycowej .
- asocjacyjna przewaga
- Zjawisko, przez które powiązanie neutralnego locus do selektywnie utrzymywanego polimorfizmu powoduje wzrost heterozygotyczności neutralnego locus.
- asynapsis
- Nieprawidłowe parowanie homologicznych chromosomów podczas mejozy . Porównaj synapsy i desynapsy .
- załączony X
- Pojedynczy chromosom monocentryczny zawierający dwie lub więcej fizycznie połączonych kopii normalnego chromosomu X w wyniku naturalnej duplikacji wewnętrznej lub dowolnej z różnych metod inżynierii genetycznej . Powstały złożony chromosom skutecznie przenosi dwie lub więcej dawek wszystkich genów i sekwencji zawartych w X, ale pod wszystkimi innymi względami funkcjonuje jako pojedynczy chromosom, co oznacza, że haploidalne gamety „XX” (zamiast zwykłych gamet „X”) będą wytwarzane przez mejozę i dziedziczone przez potomstwo. W mechanizmach takich jak równowaga genów , w których płeć organizmu jest określona przez całkowitą dawkę genów sprzężonych z chromosomem X, nieprawidłowa zygota „XXY” , zapłodniona przez jedną gametę XX i jedną gametę Y, rozwinie się w kobietę.
- autosom
- Każdy chromosom , który nie jest allosomem , a zatem nie bierze udziału w określaniu płci organizmu . W przeciwieństwie do chromosomów płciowych, autosomy w diploidach komórki istnieją w parach, przy czym członkowie każdej pary mają tę samą strukturę, morfologię i loci genetyczne .
- autozygota
- Komórka lub organizm homozygotyczny pod względem locus , w którym dwa allele homologiczne są identyczne pod względem pochodzenia, oba pochodzą z jednego genu wspólnego przodka. Porównaj allozygotę .
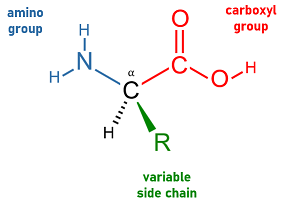
Każdy aminokwas ma ten sam podstawowy wzór strukturalny, centralny węgiel „alfa” (α) związany z trzema głównymi podstawnikami: jedną grupą aminową (niebieska), jedną grupą karboksylową (czerwoną) i jednym zmiennym łańcuchem bocznym (zielony). Łańcuch boczny nadaje każdemu aminokwasowi jego unikalną tożsamość i może wahać się od prostej grupy metylowej ( alanina ) do bardziej złożonych grup funkcyjnych, takich jak indol z podwójnym pierścieniem ( tryptofan ). Podczas tłumaczenia , aminokwasy są łączone w liniowy łańcuch poprzez reakcje kondensacji, które tworzą wiązania peptydowe między grupą karboksylową jednego aminokwasu a grupą aminową sąsiedniego aminokwasu. Mówi się, że pierwszy i ostatni aminokwas w łańcuchu są odpowiednio N-końcowe i C-końcowe , w odniesieniu do niezwiązanej grupy aminowej pierwszego aminokwasu i niezwiązanej grupy karboksylowej ostatniego.
B
- Chromosom B
- Dowolna nadliczbowa cząsteczka jądrowego DNA , która nie jest duplikatem ani homologiem żadnego ze standardowego dopełnienia normalnych chromosomów „A” składających się na genom. Zazwyczaj bardzo małe i pozbawione genów strukturalnych chromosomy B z definicji nie są niezbędne do życia. Chociaż występują naturalnie u wielu gatunków eukariotycznych, nie są stabilnie dziedziczone i dlatego różnią się znacznie liczbą kopii, nawet między blisko spokrewnionymi osobnikami.
- B-DNA
- „Standardowa” lub klasyczna konformacja strukturalna podwójna helisa DNA in vivo reprezentuje średnią z różnych różnych konformacji przyjmowanych przez bardzo długie cząsteczki DNA w warunkach fizjologicznych. Podwójna helisa typu B ma prawoskrętny skręt o średnicy 23,7 angstremów i skoku 35,7 angstremów lub około 10,5 par zasad na pełny obrót, tak że każda para nukleotydów jest obrócona o 36° wokół osi helisy w stosunku do swojej sąsiednie pary. Zobacz także A-DNA i Z-DNA .
- mutacja Mutacja , która odwraca efekt poprzedniej mutacji, która inaktywowała gen, przywracając w ten sposób funkcję typu dzikiego . Zobacz także mutację odwrotną .
- wsteczna
- krzyżowanie wsteczne
- Hodowla organizmu hybrydowego z jednym z jego rodziców lub osobnikiem genetycznie podobnym do jednego z rodziców, często celowo jako rodzaj selektywnej hodowli , w celu wytworzenia potomstwa o tożsamości genetycznej bliższej rodzicowi. Zdarzenie reprodukcyjne i wynikające z niego potomstwo są określane jako krzyżówka wsteczna , często skracana w genetyce skrótem symbolu BC .
- podstawa
- sztucznego chromosomu bakteryjnego (BAC)
- Skrót od zasady azotowej i zasady nukleinowej .
- para zasad (bp)
- Para dwóch zasad nukleinowych na komplementarnych niciach DNA lub RNA , które są połączone ze sobą wiązaniami wodorowymi . Zdolność kolejnych par zasad do układania się jedna na drugiej przyczynia się do długołańcuchowych podwójnej helisy obserwowanych zarówno w cząsteczkach dwuniciowego DNA , jak i dwuniciowego RNA .
- linia bazowa
- Miara poziomu ekspresji genu lub geny przed zaburzeniem w eksperymencie, jak w kontroli negatywnej . Ekspresja linii podstawowej może również odnosić się do oczekiwanej lub historycznej miary ekspresji genu.
- podstawowe narzędzie do wyszukiwania lokalnego dopasowania (BLAST)
- Algorytm komputerowy szeroko stosowany w bioinformatyce do dopasowywania i porównywania informacji o podstawowych sekwencjach biologicznych, takich jak sekwencje nukleotydowe DNA lub RNA lub sekwencje aminokwasowe białek. Programy BLAST umożliwiają naukowcom szybkie sprawdzenie homologii między dwiema lub więcej sekwencjami poprzez bezpośrednie porównanie nukleotydów lub aminokwasów obecnych w każdej pozycji w każdej sekwencji; powszechnym zastosowaniem jest wyszukiwanie dopasowań między określoną sekwencją zapytania a cyfrową bazą danych sekwencji, taką jak biblioteka genomu , przy czym program zwraca listę sekwencji z bazy danych, które przypominają sekwencję zapytania powyżej określonego progu podobieństwa. Takie porównania mogą pozwolić na identyfikację organizmu z nieznanej próbki lub wnioskowanie o ewolucyjnych związkach między genami, białkami lub gatunkami.
- replikacja dwukierunkowa
- Powszechny mechanizm replikacji DNA , w którym dwa widełki replikacyjne poruszają się w przeciwnych kierunkach od tego samego początku ; skutkuje to obszarem przypominającym bąbelek , w którym cząsteczka dupleksu jest lokalnie rozdzielona na dwie pojedyncze nici .
- blot
- biwalentny
- Dowolna z różnych metod biologii molekularnej, za pomocą których elektroforetycznie lub chromatograficznie rozdziela się DNA , RNA lub białko są przenoszone z nośnika, takiego jak żel poliakryloamidowy, na nośnik, taki jak unieruchamiający papier lub matryca membranowa. Niektóre metody polegają na przenoszeniu cząsteczek przez działanie kapilarne (np. Southern i Northern blotting ), podczas gdy inne opierają się na transporcie naładowanych cząsteczek przez elektroforezę (np. western blotting ). Przeniesione cząsteczki są następnie wizualizowane przez barwienie barwnikiem, autoradiografię lub selektywne znakowanie cząsteczek zawierających określone sekwencje lub epitopy z sondy hybrydyzacyjne lub przeciwciała związane z reporterem chemiluminescencyjnym.
- tępy koniec
- bromodeoksyurydyny (BUDR, BrdU)
C
- C-koniec
- Koniec liniowego łańcucha aminokwasów (np. peptyd ), który jest zakończony wolną grupą karboksylową ( -COOH ) ostatniego aminokwasu, który ma zostać dodany do łańcucha podczas translacji . Mówi się, że ten aminokwas jest C-końcowy . Zgodnie z konwencją, sekwencje, domeny, miejsca aktywne lub dowolna inna struktura znajdująca się bliżej C-końca polipeptydu lub jego postaci sfałdowanego białka w stosunku do innych są opisywane jako znajdujące się poniżej . Kontrast N-koniec .
- Wartość C
- Całkowita ilość DNA zawarta w haploidalnym jądrze (np. gamecie ) określonego organizmu lub gatunku, wyrażona w liczbie par zasad lub w jednostkach masy (zwykle pikogramach ); lub, równoważnie, połowę ilości w diploidalnej komórce somatycznej . W przypadku prostych diploidalnych eukariontów termin ten jest często używany zamiennie z wielkością genomu , ale w niektórych przypadkach, np. w hybrydowych poliploidalnych pochodzą od rodziców różnych gatunków, wartość C może faktycznie reprezentować dwa lub więcej różnych genomów zawartych w tym samym jądrze. Wartości C odnoszą się tylko do DNA genomowego iw szczególności wykluczają DNA pozajądrowe .
- Zagadka wartości C
- Termin używany do opisania różnorodnych pytań dotyczących ogromnej zmienności wartości jądrowej C lub wielkości genomu wśród gatunków eukariotycznych, w szczególności obserwacji, że wielkość genomu nie koreluje z postrzeganą złożonością organizmów ani niekoniecznie z liczbą genów posiadają; na przykład wiele jednokomórkowych protistów ma genomy zawierające tysiące razy więcej DNA niż genom ludzki . Uznano to za paradoks aż do odkrycia, że genomy eukariotyczne składają się głównie z niekodującego DNA , który jest całkowicie pozbawiony genów. Od tego czasu zagadka skupiła się na zrozumieniu, dlaczego i w jaki sposób genomy zostały wypełnione tak dużą ilością niekodującego DNA oraz dlaczego niektóre genomy mają wyższą zawartość genów niż inne.
- gen Gen regulatorowy , który ogranicza ekspresję innych genów do określonych tkanek lub części ciała w organizmie, zwykle poprzez wytwarzanie produktów genowych , które w różny sposób hamują lub umożliwiają transkrypcję innych genów w różnych typach komórek. Termin ten jest najczęściej używany w genetyce roślin .
- katastralny
- kanalizacja
- Zdolność populacji do konsekwentnego wytwarzania tego samego fenotypu niezależnie od zmienności jej środowiska lub zmienności genetycznej w jej genomie . Pojęcie to jest najczęściej używane w biologii rozwoju do interpretacji obserwacji, że ścieżki rozwojowe są często kształtowane przez dobór naturalny takie, że rozwijające się linie komórkowe są „kierowane” lub „kanalizowane” w kierunku pojedynczego, określonego losu, stając się stopniowo coraz bardziej odporne na wszelkie drobne zakłócenia, które mogą przekierować rozwój komórek z dala od ich początkowego przebiegu.
- kandydujący
- gen Gen , którego lokalizacja na chromosomie jest powiązana z określonym fenotypem (często fenotypem związanym z chorobą), w związku z czym podejrzewa się, że powoduje fenotyp lub przyczynia się do jego powstania. Geny kandydujące są często wybierane do badań na podstawie a priori wiedza lub spekulacje na temat ich funkcjonalnego znaczenia dla badanej cechy lub choroby.
- sekwencja kanoniczna
- Zobacz sekwencję konsensusową .
- koniec karboksylowy
- Zobacz C-koniec .
- nosiciel
- Osoba, która odziedziczyła recesywny allel dla cechy genetycznej lub mutacji, ale u której ta cecha zwykle nie jest wyrażona ani obserwowalna w fenotypie . Nosiciele są zwykle heterozygotami dla allelu recesywnego i dlatego nadal jest w stanie przekazać allel swojemu potomstwu, gdzie powiązany fenotyp może pojawić się ponownie, jeśli potomstwo odziedziczy inną kopię allelu. Termin ten jest powszechnie używany w genetyce medycznej w kontekście recesywnego allelu powodującego chorobę.
- Pudełko CCAAT
- Wysoce konserwatywna regulatorowa sekwencja DNA zlokalizowana około 75 par zasad w górę (tj. -75) od miejsca startu transkrypcji wielu genów eukariotycznych.
- cDNA
- Patrz komplementarny DNA .
- DNA wolne od komórek (cfDNA)
- Każda cząsteczka DNA istniejąca poza komórką lub jądrem , swobodnie unosząca się w płynie pozakomórkowym, takim jak osocze krwi .
- przeprogramowanie komórkowe
- Konwersja a komórki z jednego specyficznego dla tkanki typu komórek do innego. Obejmuje to odróżnicowanie do stanu pluripotentnego ; przykładem jest konwersja mysich komórek somatycznych do niezróżnicowanego stanu embrionalnego , która opiera się na czynnikach transkrypcyjnych Oct4 , Sox2 , Myc i Klf4 .
- centymorgan (cM)
- Jednostka miary powiązań genetycznych zdefiniowana jako odległość między loci chromosomalnymi , dla której oczekiwana średnia liczba pośrednich krzyżowań chromosomów w jednym pokoleniu wynosi 0,01. Chociaż nie jest to rzeczywista miara odległości fizycznej, jest używana do wnioskowania o rzeczywistej odległości między dwoma loci na podstawie widocznego prawdopodobieństwa wystąpienia między nimi skrzyżowania.
- centralny dogmat biologii molekularnej
- Uogólnione ramy zrozumienia przepływu informacji genetycznej między makrocząsteczkami w systemach biologicznych. Centralny dogmat nakreśla fundamentalną zasadę, że informacja o sekwencji zakodowana w trzech głównych klasach biopolimerów — DNA , RNA i białka — może być przenoszona między tymi trzema klasami tylko w określony sposób, a w inny nie. kwasy nukleinowe iz kwasu nukleinowego na białko jest możliwe, ale przeniesienie z białka na białko lub z białka z powrotem do dowolnego typu kwasu nukleinowego jest niemożliwe i nie zachodzi naturalnie.
- centriola
- Cylindryczne organelle złożone z mikrotubul, obecne tylko u niektórych eukariotów. Para centrioli migruje i definiuje dwa przeciwne bieguny dzielącej się komórki , gdzie jako część centrosomu inicjuje wzrost aparatu wrzeciona .
- centromer
- Wyspecjalizowana sekwencja DNA w obrębie chromosomu , która łączy parę siostrzanych chromatyd . Podstawową funkcją centromeru jest działanie jako miejsce składania kinetochorów , kompleksów białkowych, które kierują przyłączaniem włókna wrzeciona do centromeru i ułatwiają segregację chromatyd podczas mitozy lub mejozy .
- indeks centromerowy
- Proporcja całkowitej długości chromosomu objętego jego krótkim ramieniem , zwykle wyrażana w procentach; np. chromosom o indeksie centromerowym 15 jest akrocentryczny , z krótkim ramieniem stanowiącym tylko 15% jego całkowitej długości.
- cfDNA
- Patrz DNA wolne od komórek .
- Reguły Chargaffa
- Zestaw aksjomatów, które stwierdzają, że w DNA dowolnego chromosomu, gatunku lub organizmu całkowita liczba reszt adeniny ( A ) będzie w przybliżeniu równa całkowitej liczbie reszt tyminy ( T ) i liczbie reszt guaniny ( G ) reszty będą równe liczbie reszt cytozyny ( C ); odpowiednio, całkowita liczba puryn ( A + G ) będzie równa całkowitej liczbie pirymidyny ( T + C ). Obserwacje te ilustrują wysoce specyficzną naturę komplementarnych par zasad , które występują we wszystkich cząsteczkach dupleksu DNA: chociaż niestandardowe parowanie jest technicznie możliwe, są one wyjątkowo rzadkie, ponieważ standardowe są silnie faworyzowane w większości warunków. Mimo to równoważność 1: 1 rzadko jest dokładna, ponieważ w dowolnym momencie stosunki nukleozasad są nieuchronnie zniekształcone w pewnym niewielkim stopniu przez nienaprawione niedopasowania , brakujące zasady i zasady niekanoniczne. Obecność jednoniciowe polimery DNA również zmieniają proporcje, ponieważ sekwencja pojedynczej nici może zawierać dowolną liczbę dowolnych zasad.
- naładowany tRNA
- Przenoszący RNA, do którego przyłączono aminokwas ; tj. aminoacylowany tRNA . Nienaładowane tRNA nie mają aminokwasów.
- chDNA
- Patrz DNA chloroplastów .
- chiazma
- Połączenie w kształcie krzyża, które tworzy fizyczny punkt styku między dwiema niesiostrzanymi chromatydami należącymi do chromosomów homologicznych podczas synapsy . Oprócz zapewnienia właściwej segregacji chromosomów, połączenia te są również punktami przerwania , w których może wystąpić krzyżowanie chromosomów podczas mitozy lub mejozy , co skutkuje wzajemną wymianą DNA między synapsowanymi chromatydami.
- chimeryzm
- Obecność dwóch lub więcej populacji komórek o różnych cechach genotypy w pojedynczym organizmie, znanym jako chimera , który rozwinął się z fuzji komórek pochodzących z oddzielnych zygot ; każda populacja komórek zachowuje swój własny genom, tak że organizm jako całość jest mieszanką genetycznie nieidentycznych tkanek. Chimeryzm genetyczny może być dziedziczony (np. przez fuzję wielu zarodków podczas ciąży) lub nabyty po urodzeniu (np. przez allogeniczne przeszczepienie komórek, tkanek lub narządów od genetycznie nieidentycznego dawcy); u roślin może być wynikiem szczepienia lub błędy w podziale komórki. Jest podobny do mozaicyzmu , ale różni się od niego .
- chloroplastowy DNA (cpDNA, chDNA, ctDNA)
- Zestaw cząsteczek DNA zawartych w chloroplastach , rodzaju fotosyntetycznych plastydowych organelli zlokalizowanych w komórkach niektórych organizmów eukariotycznych, takich jak rośliny i glony, reprezentujący częściowo autonomiczny genom odrębny od genomu znajdującego się w jądrze komórkowym . Podobnie jak inne rodzaje plastydowego DNA, cpDNA zwykle występuje w postaci małych okrągłych plazmidów .
- chondrom
- Kompletny zestaw mitochondriów lub mitochondrialnego DNA w komórce, tkance, organizmie lub gatunku.
- chromatyda
- Jedna kopia nowo skopiowanego chromosomu , która jest połączona z oryginalnym chromosomem przez centromer .
- chromatyna
- Kompleks DNA , RNA i białka występujący w komórkach eukariotycznych , będący pierwotną substancją zawierającą chromosomy . Chromatyna działa jako środek do pakowania bardzo długich cząsteczek DNA w wysoce zorganizowane i gęsto upakowane kształty, co zapobiega splątaniu nici, wzmacnia DNA podczas podziału komórki, pomaga zapobiegać uszkodzeniom DNA oraz odgrywa ważną rolę w regulacji ekspresji genów i replikacji DNA .
- chromocentrum
- immunoprecypitacji chromatyny (ChIP)
- Centralna bezpostaciowa masa chromosomów polietylenowych występująca w jądrach komórek gruczołów ślinowych larw Drosophila i powstała w wyniku połączenia heterochromatycznych regionów otaczających centromery somatycznie sparowanych chromosomów, z dystalnymi euchromatycznymi ramionami promieniującymi na zewnątrz.
- chromomer
- Region chromosomu , który został lokalnie zagęszczony lub zwinięty w chromatynę , widoczny pod mikroskopem jako „kulka”, węzeł lub zabarwiony na ciemno prążek, zwłaszcza w porównaniu z pobliskimi nieskompaktowanymi łańcuchami DNA.
- krzyżowanie chromosomów
- duplikacja chromosomu
- Duplikacja całego chromosomu , w przeciwieństwie do fragmentu chromosomu lub pojedynczego genu .
- chromosom
- Cząsteczka DNA zawierająca część lub całość materiału genetycznego organizmu. Chromosomy można uznać za rodzaj molekularnego „pakietu” do przenoszenia DNA w jądrze komórkowym i u większości eukariontów składają się z długich nici DNA zwiniętych z białkami pakującymi , które wiążą się i kondensują pasemka, aby nie stały się niemożliwą do opanowania plątaniną. Chromosomy najłatwiej rozróżnić i zbadać w ich całkowicie skondensowanej formie, która występuje tylko podczas podziału komórki . Niektóre proste organizmy mają tylko jeden chromosom zbudowany z kolistego DNA, podczas gdy większość eukariontów ma wiele chromosomów zbudowanych z liniowego DNA.
- kondensacja chromosomów
- Proces, w wyniku którego chromosomy eukariotyczne stają się krótsze, grubsze, gęstsze i bardziej widoczne pod mikroskopem podczas profazy z powodu systemowego zwijania i superskręcania chromatycznych nici DNA przygotowujące do podziału komórki .
- segregacja chromosomów
- Proces, w którym siostrzane chromatydy lub sparowane chromosomy homologiczne oddzielają się od siebie i migrują do przeciwnych stron dzielącej się komórki podczas mitozy lub mejozy .
- chodzenie po chromosomie
- Zobacz chodzenie elementarza .
- krążące DNA nowotworu (ctDNA)
- Wszelkie pozakomórkowe fragmenty DNA pochodzące z komórek nowotworowych, które swobodnie krążą w krwioobiegu.
- cis
- Po tej samej stronie; sąsiadujący z; działające z tej samej cząsteczki. Kontrast trans .
- działanie cis
- Oddziaływanie na gen lub sekwencję na tej samej cząsteczce kwasu nukleinowego. Mówi się, że locus lub sekwencja w określonej cząsteczce DNA, takiej jak chromosom , działa w układzie cis , jeśli wpływa lub oddziałuje na inne sekwencje znajdujące się w niewielkiej odległości (tj. fizycznie w pobliżu, zwykle, ale niekoniecznie poniżej ) na tej samej cząsteczce lub chromosomie; lub, w najszerszym znaczeniu, jeśli wpływa lub działa na inne sekwencje znajdujące się w dowolnym miejscu (niekoniecznie w niewielkiej odległości) na tym samym chromosomie homologicznej pary . Czynniki działające w układzie cis są często zaangażowane w regulację ekspresji genów , hamując lub ułatwiając transkrypcję . Kontrastowe aktorstwo .
- cis -dominująca mutacja
- Mutacja występująca w elemencie cis -regulatorowym (taki jak operator ), który zmienia działanie pobliskiego genu lub genów na tym samym chromosomie . Mutacje dominujące cis wpływają na ekspresję genów, ponieważ występują w miejscach kontrolujących transkrypcję, a nie w samych genach.
- cis - element regulacyjny (CRE)
- Dowolna sekwencja lub region niekodującego DNA , który reguluje transkrypcję pobliskich genów (np. promotor , operator , wyciszacz lub wzmacniacz ), zwykle służąc jako miejsce wiązania dla jednego lub większej liczby czynników transkrypcyjnych . Kontrast transregulacyjny .
- cisgenesis
- cistron
- genetyka klasyczna
- Dział genetyki opiera się wyłącznie na obserwacji widocznych rezultatów aktów reprodukcyjnych, w przeciwieństwie do tego, który jest możliwy dzięki nowoczesnym technikom i metodologiom biologii molekularnej . Porównaj genetykę molekularną .
- klonowanie
- Proces wytwarzania, w sposób naturalny lub sztuczny, pojedynczych organizmów lub komórek, które są genetycznie identyczne. Klony są wynikiem wszelkich form rozmnażania bezpłciowego i komórek, które przechodzą mitozę produkują komórki potomne, które są klonami komórki macierzystej i siebie nawzajem. Klonowanie może również odnosić się do metod biotechnologicznych, które sztucznie tworzą kopie organizmów lub komórek lub, w przypadku klonowania molekularnego , kopie fragmentów DNA lub innych cząsteczek.
- zamknięta chromatyna
- Patrz heterochromatyna .
- koaktywator
- Typ koregulatora , który zwiększa ekspresję jednego lub więcej genów poprzez wiązanie się z aktywatorem .
- nić kodująca
- Nić dwuniciowej cząsteczki DNA, której sekwencja nukleotydów odpowiada bezpośrednio sekwencji transkryptu RNA powstałego podczas transkrypcji (z wyjątkiem tego, że zasady tyminy są zastąpione zasadami uracylowymi w cząsteczce RNA). Chociaż sama nie podlega transkrypcji, nić kodująca jest zgodnie z konwencją nicią używaną podczas prezentacji sekwencji DNA ze względu na bezpośrednią analogię między jej sekwencją a kodonami produktu RNA. Kontrastowe pasmo szablonu ; zobacz także sens .
- kodon
- kodominacyjny
- Seria trzech kolejnych nukleotydów w regionie kodującym sekwencji kwasu nukleinowego . Każda z tych trójek koduje określony aminokwas lub sygnał stop podczas syntezy białek . Cząsteczki DNA i RNA są zapisane w języku przy użyciu czterech „liter” (cztery różne zasady nukleinowe ), ale język używany do konstruowania białek obejmuje 20 „liter” (20 różnych aminokwasów). Kodony zapewniają klucz, który umożliwia tłumaczenie tych dwóch języków w siebie. Ogólnie rzecz biorąc, każdy kodon odpowiada pojedynczemu aminokwasowi (lub sygnałowi stop). Pełny zestaw kodonów nazywany jest kodem genetycznym .
- błąd użycia kodonów
- Preferencyjne użycie określonego kodonu do kodowania określonego aminokwasu zamiast alternatywnych kodonów, które są synonimami tego samego aminokwasu, o czym świadczą różnice między organizmami w częstotliwości występowania synonimicznych kodonów w ich DNA kodującym. Ponieważ kod genetyczny jest zdegenerowany , większość aminokwasów można określić za pomocą wielu kodonów. Niemniej jednak niektóre kodony są zwykle nadreprezentowane (a inne niedoreprezentowane) u różnych gatunków.
- kofaktor
- Każdy niebiałkowy związek organiczny związany z enzymem . Do zainicjowania katalizy wymagane są kofaktory .
- porównawcza hybrydyzacja genomowa (CGH)
- komplementarność
- Właściwość biopolimerów kwasu nukleinowego polegająca na tym, że dwa łańcuchy polimerowe lub „ nici ” ustawione antyrównolegle względem siebie będą miały tendencję do tworzenia się pary zasad składające się z wiązań wodorowych między poszczególnymi zasadami nukleotydowymi składającymi się na każdy łańcuch, przy czym każdy rodzaj zasad nukleinowych tworzy pary prawie wyłącznie z jednym innym rodzajem zasad nukleinowych; np. w cząsteczkach dwuniciowego DNA A łączy się w pary tylko z T , a pary C tylko z G. Mówi się, że nici sparowane w taki sposób i same zasady są komplementarne . Stopień komplementarności między dwoma pasmami silnie wpływa na stabilność cząsteczka dupleksowa ; niektóre sekwencje mogą być również wewnętrznie komplementarne, co może prowadzić do wiązania się pojedynczej nici ze sobą . Komplementarność ma fundamentalne znaczenie dla mechanizmów rządzących replikacją DNA , transkrypcją i naprawą DNA .
- komplementarny DNA (cDNA)
- DNA , które jest syntetyzowane z matrycy jednoniciowego RNA (zazwyczaj mRNA lub miRNA ) w reakcji katalizowanej przez enzym odwrotnej transkryptazy . cDNA jest wytwarzany zarówno naturalnie przez retrowirusy , jak i sztucznie za pomocą pewnych technik laboratoryjnych, zwłaszcza klonowania molekularnego . W bioinformatyce termin ten może być również używany w odniesieniu do sekwencji transkryptu mRNA wyrażanego jako odpowiednik nici kodującej DNA (tj. uracyl ).
- cecha kompleksu
- komplementarnego
- Patrz cecha ilościowa .
- związek X
- Patrz załącznik X.
- ekspresja warunkowa
- Kontrolowana, indukowana ekspresja transgenu , in vitro lub in vivo .
- kongres
- Ruch chromosomów do równika wrzeciona podczas prometafazowych i metafazowych stadiów mitozy .
- spokrewniony
- (dwóch lub więcej osobników) blisko spokrewniony genetycznie; dzieląc niedawnego wspólnego przodka (zwykle nie dalej niż trzy lub cztery pokolenia). Efektem kojarzenia spokrewnionego, znanego również jako chów wsobny , jest zwiększenie prawdopodobieństwa, że potomstwo będzie homozygotyczne w dowolnej parze loci genetycznych .
- Sekwencja konsensusu
- Obliczona kolejność najczęstszych reszt ( nukleotydów lub aminokwasów ) znalezionych w każdej pozycji we wspólnym dopasowaniu sekwencji i uzyskana przez porównanie wielu blisko spokrewnionych dopasowań sekwencji.
- genetyka konserwatorska
- Interdyscyplinarna gałąź genetyki populacyjnej , która stosuje metody i koncepcje genetyczne w celu zrozumienia dynamiki genów w populacjach, a jej głównym celem jest zapobieganie wymieraniu oraz zachowanie i przywracanie różnorodności biologicznej .
- konserwatywna replikacja
- Hipotetyczny tryb replikacji DNA , w którym dwie nici rodzicielskie oryginalnej dwuniciowej cząsteczki DNA ostatecznie pozostają do siebie wzajemnie uzupełnione na końcu procesu replikacji, a dwie nici potomne ostatecznie tworzą własną, oddzielną cząsteczkę; stąd jedna cząsteczka składa się z obu nici wyjściowych, podczas gdy druga składa się wyłącznie z nowo zsyntetyzowanych nici. Kontrastuje to z replikacją semikonserwatywną , w której każda cząsteczka jest hybrydą jednej starej i jednej nowej nici. Zobacz także replikację dyspersyjną .
- konserwowana sekwencja Sekwencja
- kwasu nukleinowego lub białka , która jest bardzo podobna lub identyczna u wielu gatunków lub w obrębie genomu , co wskazuje, że pozostała względnie niezmieniona przez długi okres ewolucji.
- conspecific
- Należący do tego samego gatunku.
- ekspresja konstytutywna
- 1. Ciągła transkrypcja genu , w przeciwieństwie do ekspresji fakultatywnej , w której gen jest transkrybowany tylko w razie potrzeby. Gen, który podlega ciągłej transkrypcji, nazywa się a gen konstytutywny .
- 2. Gen, którego ekspresja zależy tylko od skuteczności jego promotora w wiązaniu polimerazy RNA , a nie od jakichkolwiek czynników transkrypcyjnych lub innych elementów regulatorowych , które mogłyby promować lub hamować jego transkrypcję.
- contig
- Ciągła sekwencja genomowego DNA generowana przez składanie sklonowanych fragmentów za pomocą ich nakładających się sekwencji.
- kopia DNA (cDNA)
- Patrz komplementarny DNA .
- błąd kopiowania
- Mutacja będąca wynikiem błędu popełnionego podczas replikacji DNA .
- zmienność liczby kopii (CNV)
- Zjawisko, w którym sekcje genomu są powtarzane, a liczba powtórzeń jest różna u poszczególnych osobników w populacji, zwykle w wyniku zdarzeń duplikacji lub delecji , które wpływają na całe geny lub sekcje chromosomów. Zmienność liczby kopii odgrywa ważną rolę w generowaniu zmienności genetycznej w populacji.
- koregulator
- Białko współpracujące z jednym lub więcej czynniki transkrypcyjne regulujące ekspresję genów .
- korepresor
- Rodzaj koregulatora , który zmniejsza (represjonuje) ekspresję jednego lub większej liczby genów poprzez wiązanie się z represorem i jego aktywację .
- cosmid
- cpDNA
- Patrz DNA chloroplastów .
- witryna CpG
- Sekwencja DNA, w której bezpośrednio po nukleotydzie cytozyny następuje nukleotyd guaniny na tej samej nici w kierunku od 5' do 3' ; „p” w CpG odnosi się po prostu do pośredniej fosforanowej łączącej dwa kolejne nukleotydy.
- Wyspa CpG
- Region genomu , w którym miejsca CpG występują powtarzalnie lub z dużą częstotliwością.
- Krzyżowanie
- Hodowla czystorasowych rodziców należących do dwóch różnych ras , odmian lub populacji, często celowo jako rodzaj hodowli selektywnej , w celu wytworzenia potomstwa, które ma cechy wspólne z obydwoma liniami rodzicielskimi lub które wykazuje heterozję . W hodowli zwierząt potomstwo z krzyżówki ras tego samego gatunku nazywa się mieszańcem , natomiast potomstwo z krzyżówki różnych gatunków nazywa się mieszańcem .
- crossing over
- Patrz krzyżowanie chromosomów .
- usieciowanie
- Nieprawidłowe wiązanie chemiczne między dwiema lub większą liczbą zasad nukleinowych na przeciwległych niciach dwuniciowej cząsteczki DNA ( interstrand ) lub między zasadami na tej samej nici ( intrastrand ), w szczególności poprzez tworzenie wiązań kowalencyjnych , które są silniejsze niż wiązania wodorowe zasady parowanie . Wiązania poprzeczne mogą być generowane przez różne czynniki egzogenne i endogenne i mają tendencję do zakłócania normalnych procesów komórkowych, takich jak i transkrypcja DNA . Są częstym celem naprawy DNA .
- Edycja genów CRISPR
- ctDNA
- 1. Skrót od circulating nowotworowego DNA .
- 2. Skrót od chloroplastowego DNA .
- kryptyczny niestabilny transkrypt (CUT)
- cytydyna ( C , Cyd) Jeden
- z czterech standardowych nukleozydów stosowanych w cząsteczkach RNA , składający się z zasady cytozyny z azotem N9 związanym z C1 węgiel cukru rybozowego . Cytozyna związana z dezoksyrybozą jest znana jako deoksycytydyna , która jest wersją stosowaną w DNA .
- cytogenetyka
- Dział genetyki , który bada, w jaki sposób chromosomy wpływają na zachowanie i funkcje komórek , szczególnie podczas mitozy i mejozy .
- cytokineza
- Końcowy etap podziału komórki zarówno w mitozie , jak i mejozie , podczas którego zamknięcie pierścienia kurczliwego powoduje, że cytoplazma komórki macierzystej zostaje w przybliżeniu równomiernie podzielona między dwie komórki potomne. Porównaj kariokinezę .
- cytozyna ( C )
- Pirymidynowa zasada nukleinowa stosowana jako jedna z czterech standardowych zasad nukleinowych zarówno w cząsteczkach DNA , jak i RNA . Cytozyna tworzy parę zasad z guaniną .

Możliwe typy przekazywania informacji według centralnego dogmatu biologii molekularnej . Trzy ogólne transfery, zaznaczone na czerwono, występują rutynowo we wszystkich żywych komórkach: DNA-do-DNA ( replikacja DNA ), DNA-do-RNA ( transkrypcja ) i RNA-do-białka ( translacja ). Wiadomo, że trzy specjalne transfery, zaznaczone na niebiesko, występują tylko w wirusach lub w laboratorium: RNA-na-RNA ( replikacja RNA ), RNA-na-DNA ( odwrotna transkrypcja ) i DNA na białko (translacja bezpośrednia bez pośredniego mRNA). Uważa się, że dodatkowe trzy transfery w ogóle nie są możliwe: z białka na białko, z białka na RNA i z białka na DNA - chociaż argumentowano, że istnieją wyjątki, w których wszystkie trzy mogą wystąpić.
D
- mutacja de novo
- Spontaniczna mutacja w genomie pojedynczego organizmu, która jest nowa w linii tego organizmu, która po raz pierwszy pojawiła się w komórce rozrodczej jednego z rodziców organizmu lub w zapłodnionej komórce jajowej, z której rozwija się organizm; tj. mutacja, która nie była obecna w genomie żadnego z rodziców.
- synteza de novo
- Złożenie syntetycznej sekwencji kwasu nukleinowego z wolnych nukleotydów bez polegania na istniejącej nici matrycowej , tj. de novo , za pomocą dowolnej z różnych metod laboratoryjnych. Synteza de novo teoretycznie umożliwia konstruowanie całkowicie sztucznych cząsteczek bez naturalnie występujących odpowiedników i bez ograniczeń co do wielkości lub sekwencji. Jest wykonywany rutynowo w komercyjnej produkcji dostosowanych, wykonywanych na zamówienie oligonukleotydowych, takich jak startery .
- degeneracja
- Redundancja kodu genetycznego przejawiająca się mnogością różnych kodonów określających ten sam aminokwas . Na przykład w standardowym kodzie genetycznym aminokwas seryna jest określony przez sześć unikalnych kodonów ( UCA , UCG , UCC , UCU , AGU i AGC ). Degeneracja kodonów odpowiada za istnienie mutacji synonimicznych .
- usunięcie
- Rodzaj mutacji , w której jeden lub więcej nukleotydów jest usuwanych z sekwencji kwasu nukleinowego .
- denaturacja
- Proces, w wyniku którego kwasy nukleinowe lub białka tracą swoją czwartorzędową , trzeciorzędową i/lub drugorzędową strukturę , odwracalnie lub nieodwracalnie, poprzez zastosowanie zewnętrznego obciążenia chemicznego lub mechanicznego, np. przez ogrzewanie, mieszanie lub wystawienie na działanie silnego kwasu lub zasady, z których wszystkie mogą zakłócać siły międzycząsteczkowe, takie jak wiązania wodorowe a tym samym zmienić lub zniszczyć aktywność chemiczną. Zdenaturowane białka mogą być zarówno przyczyną, jak i skutkiem śmierci komórki. Denaturacja może być również normalnym procesem; denaturacja dwuniciowych cząsteczek DNA, która rozrywa wiązania wodorowe między parami zasad i powoduje rozdzielenie cząsteczki dupleksu na dwie pojedyncze nici , jest niezbędnym etapem replikacji i transkrypcji DNA , a zatem jest rutynowo przeprowadzana przez enzymy, takie jak jako helikazy . Ten sam mechanizm jest również fundamentalny dla metod laboratoryjnych, takich jak PCR .
- dezoksyadenozyna
- Jeden z czterech standardowych dezoksyrybonukleozydów stosowanych w cząsteczkach DNA , składający się z zasady adeninowej z azotem N9 związanym z atomem węgla C1 cukru dezoksyrybozy . Adenina związana z rybozą tworzy alternatywny związek znany po prostu jako adenozyna , który jest używany w RNA .
- dezoksycytydyna
- Jeden z czterech standardowych dezoksyrybonukleozydów stosowanych w cząsteczkach DNA , składający się z zasady cytozyny z azotem N9 związanym z atomem węgla C1 cukru dezoksyrybozy . Cytozyna związana z rybozą tworzy alternatywny związek znany po prostu jako cytydyna , który jest używany w RNA .
- deoksyguanozyna
- Jeden z czterech standardowych dezoksyrybonukleozydów stosowanych w cząsteczkach DNA , składający się z zasady guaniny z azotem N9 związanym z atomem węgla C1 cukru dezoksyrybozy . Guanina związana z rybozą tworzy alternatywny związek znany po prostu jako guanozyna , który jest używany w RNA .
- dezoksytymidyna
- Patrz tymidyna .
- dezoksyrybonukleaza (DNaza)
- Dowolna z klasy nukleazy , które katalizują hydrolityczne rozszczepianie wiązań fosfodiestrowych w cząsteczkach DNA , odcinając w ten sposób nici dezoksyrybonukleotydów i powodując degradację polimerów DNA na mniejsze składniki. Porównaj rybonukleazę .
- kwas dezoksyrybonukleinowy (DNA)
- Polimeryczna cząsteczka kwasu nukleinowego złożona z szeregu dezoksyrybonukleotydów , z których każdy zawiera jedną z czterech kanonicznych zasad nukleinowych : adenina ( A ), guanina ( G ), cytozyna ( C ) i tymina ( T ). DNA najczęściej występuje w dwuniciowej , która składa się z dwóch komplementarnych antyrównoległych łańcuchów nukleotydowych, w których każda z zasad nukleinowych na każdej pojedynczej nici jest sparowana poprzez wiązanie wodorowe z zasadą na przeciwległej nici; struktura ta zwykle występuje w kształcie podwójnej helisy . DNA może również istnieć w jednoniciowej .
- dezoksyrybonukleotyd
- Nukleotyd zawierający dezoksyrybozę jako składnik pentozowy oraz monomeryczną podjednostkę cząsteczek kwasu dezoksyrybonukleinowego ( DNA). Dezoksyrybonukleotydy kanonicznie zawierają dowolną z czterech zasad azotowych : adeninę ( A ), guaninę ( G ), cytozynę ( C ) i tyminę ( T ). Porównywać rybonukleotyd .
- dezoksyryboza
- Cukier monosacharydowy otrzymywany z rybozy przez utratę pojedynczego atomu tlenu. D-dezoksyryboza, w postaci pierścienia pentozowego, jest jedną z trzech podstawowych grup funkcyjnych dezoksyrybonukleotydów , a tym samym cząsteczek kwasu dezoksyrybonukleinowego (DNA).
- desynapsis
- Niepowodzenie homologicznych chromosomów , które normalnie tworzyły synapsy podczas pachynemy , aby pozostały sparowane podczas diplonemy . Desynapsis jest zwykle spowodowane nieprawidłowym tworzeniem chiasmata . Kontrastowe asynapsy .
- diakineza
- W mejozie piąty i ostatni podetap profazy I , następujący po diplonema i poprzedzający metafazę I. Podczas diakinezy chromosomy ulegają dalszej kondensacji, obydwa centrosomy docierają do przeciwległych biegunów komórki, a aparat wrzeciona zaczyna rozciągać się od biegunów do równika.
- dicentryczny (
- chromosomu liniowego lub fragmentu chromosomu) Posiadający dwa centromery zamiast jednego normalnego.
- dinukleotyd
- Dowolne dwa nukleotydy bezpośrednio przylegające do siebie na tej samej nici cząsteczki kwasu nukleinowego .
- diploidalny
- (komórki lub organizmu) Posiadanie dwóch homologicznych kopii każdego chromosomu . Porównaj haploidalne i poliploidalne .
- dyplomowany
- W mejozie czwarty z pięciu podetapów profazy I , następujący po pachynemie i poprzedzający diakinezę . Podczas diplonemy kompleks synaptonemalny rozkłada się, a sparowane homologiczne chromosomy zaczynają się od siebie oddzielać, chociaż pozostają ściśle związane w skrzyżowaniu , w którym nastąpiło skrzyżowanie .
- kierunkowość
- Orientacja chemiczna od końca do końca pojedynczej liniowej nici lub sekwencji a kwasu nukleinowego lub polipeptyd . Nomenklatura używana do wskazania kierunkowości kwasów nukleinowych opiera się na chemicznej konwencji identyfikacji poszczególnych atomów węgla w cukrach rybozy lub dezoksyrybozy nukleotydów , w szczególności na węglu 5' i węglu 3' pierścienia pentozowego. Sekwencja nukleotydów w łańcuchu polimerowym może być odczytywana lub interpretowana w kierunku od 5' do 3' – tj. zaczynając od końcowego nukleotydu, w którym węgiel 5' nie jest połączony z innym nukleotydem i przechodząc do drugiego końcowego nukleotydu , w którym węgiel 3' nie jest połączony z innym nukleotydem – lub w kierunku przeciwnym od 3' do 5'. Większość rodzajów syntezy kwasów nukleinowych, w tym i transkrypcja DNA , działają wyłącznie w kierunku od 5' do 3', ponieważ zaangażowane enzymy mogą katalizować jedynie dodanie wolnych nukleotydów do otwartego końca 3' poprzedniego nukleotydu w łańcuchu. Z tego powodu konwencja podczas zapisywania dowolnej sekwencji kwasu nukleinowego polega na przedstawianiu jej w kierunku od 5' do 3' od lewej do prawej. W dwuniciowych kwasach nukleinowych dwie sparowane nici muszą być zorientowane w przeciwnych kierunkach, aby ze sobą sparować zasady . Kierunkowość polipeptydu jest podobnie oparta na znakowaniu grup funkcyjnych w aminokwasach , w szczególności grupę aminową, która tworzy N-koniec , i grupę karboksylową, która tworzy C-koniec ; sekwencje aminokwasowe są składane w kierunku N-do-C podczas translacji i prawie zawsze są zapisywane w tym samym kierunku.
- powtórzenie bezpośrednie
- Jakiekolwiek dwa lub więcej powtórzeń określonej sekwencji nukleotydów występujących w tej samej orientacji (tj. dokładnie w tej samej kolejności i nieodwróconej ) i na tej samej nici , albo oddzielone interweniującymi nukleotydami, albo nie. Przykładem jest sekwencja TACCG nnnnnn TACCG , w której TACCG występuje dwukrotnie, chociaż jest oddzielona sześcioma nukleotydami, które nie są częścią powtarzającej się sekwencji. Bezpośrednie powtórzenie, w którym powtórzenia bezpośrednio przylegają do siebie, jest znane jako powtórzenie tandemowe .
- dyspersyjna
- miara odległości replikacji
- Każda wielkość używana do pomiaru odmienności między poziomami ekspresji różnych genów .
- DNA
- Zobacz kwas dezoksyrybonukleinowy .
- DNA barcoding
- Metoda identyfikacji taksonomicznej, w której krótkie sekwencje DNA z jednego lub większej liczby określonych genów są izolowane z niezidentyfikowanych próbek, a następnie porównywane z sekwencjami z biblioteki referencyjnej w celu jednoznacznej identyfikacji gatunku lub innego taksonu, z którego pochodzą próbki. Sekwencje użyte do porównania są wybierane ostrożnie z genów, które są zarówno szeroko konserwowane , jak i wykazują większe zróżnicowanie między gatunkami niż w obrębie gatunku, np. oksydaza cytochromu c gen dla eukariontów lub niektóre geny rybosomalnego RNA dla prokariotów. Geny te są obecne w prawie wszystkich żywych organizmach, ale mają tendencję do ewoluowania różnych mutacji u różnych gatunków, tak że unikalny wariant sekwencji można powiązać z jednym konkretnym gatunkiem, skutecznie tworząc unikalny identyfikator podobny do detalicznego kodu kreskowego . Kody kreskowe DNA umożliwiają identyfikację nieznanych okazów z niewyraźnych tkanek lub części ciała, gdzie identyfikacja na podstawie morfologii byłaby trudna lub niemożliwa, a biblioteka kodów kreskowych organizmów jest obecnie na tyle obszerna, że nawet organizmy wcześniej nieznane nauce mogą być często filogenetycznie sklasyfikowane z pewnością. Jednoczesna identyfikacja wielu różnych gatunków z mieszanej próbki jest znana jako metabarkodowanie .
- Kondensacja DNA
- Proces zagęszczania bardzo długich cząsteczek DNA w gęsto upakowane, uporządkowane konfiguracje, takie jak chromosomy , in vivo lub in vitro .
- odcisków palców DNA
- Mikromacierz DNA
- Wysokowydajna technologia używana do pomiaru poziomu ekspresji mRNA Pobieranie transkryptów lub do wykrywania pewnych zmian w sekwencji nukleotydów . Składa się z szeregu tysięcy mikroskopijnych plamek oligonukleotydów DNA , zwanych cechami , z których każda zawiera pikomole o określonej sekwencji DNA. Może to być krótki odcinek genu lub dowolnego innego elementu DNA i jest używany jako sonda do hybrydyzacji próbki cDNA , cRNA lub genomowego DNA (nazywanej celem ) w warunkach o wysokiej ostrości . Hybrydyzacja sondy z celem jest zwykle wykrywany i oznaczany ilościowo przez wykrywanie oparte na fluorescencji celów znakowanych fluoroforem .
- Polimeraza DNA
- Dowolna klasa enzymów syntetyzujących cząsteczki DNA z poszczególnych dezoksyrybonukleotydów . Polimerazy DNA są niezbędne do replikacji DNA i zwykle działają w parach, tworząc identyczne kopie dwóch nici oryginalnej dwuniciowej cząsteczki. Budują długie łańcuchy DNA, dodając po jednym nukleotydzie do 3'-końca nici DNA, zwykle opierając się na matryca dostarczona przez komplementarną nić, aby wiernie skopiować sekwencję nukleotydów.
- Naprawa DNA
- Zestaw procesów, za pomocą których komórka identyfikuje i koryguje uszkodzenia strukturalne lub mutacje w cząsteczkach DNA kodujących jej genom . Zdolność komórki do naprawy swojego DNA ma zasadnicze znaczenie dla integralności genomu i normalnej funkcjonalności organizmu.
- Replikacja DNA
- Proces, w którym cząsteczka DNA kopiuje się, tworząc dwie identyczne kopie jednej oryginalnej cząsteczki DNA.
- Sekwencjonowanie DNA Proces określania, za
- pomocą różnych metod i technologii, kolejności zasad w długim łańcuchu nukleotydów, który tworzy sekwencję DNA .
- Obrót DNA
- Dowolny mechanizm, za pomocą którego sekwencje DNA są wymieniane w sposób nieodwrotny (np. poprzez konwersję genów , transpozycję lub nierówne crossing-over ), który powoduje ciągłe fluktuacje liczby kopii motywów DNA w ciągu życia organizmu. Takie mechanizmy są często głównymi czynnikami napędzającymi specjację między populacjami.
- Domena wiążąca DNA (DBD)
- Białko wiążące DNA (DBP)
- Dowolny polipeptyd lub białko zawierające jedną lub więcej domen zdolnych do chemicznego oddziaływania z jedną lub większą liczbą części cząsteczki DNA , a w konsekwencji wykazujące specyficzne lub ogólne powinowactwo do pojedynczych i/lub lub dwuniciowego DNA . Aktywność wiązania DNA często zależy od obecności i fizycznej dostępności określonej sekwencji zasad nukleinowych i występuje głównie w głównym rowku , ponieważ eksponuje więcej grup funkcyjnych, które jednoznacznie identyfikują zasady. Na wiązanie wpływa również konformacja przestrzenna łańcucha DNA i zajęcie innych białek w pobliżu miejsca wiązania; wiele białek nie może wiązać się z DNA bez uprzedniego przejścia zmian strukturalnych wywołanych interakcjami z innymi cząsteczkami.
- DNaza
- Patrz dezoksyrybonukleaza .
- dominacja
- Związek między allelami a gen , w którym jeden allel wywiera wpływ na fenotyp , który przewyższa lub „maskuje” wkład innego allelu w tym samym locus ; mówi się , że pierwszy allel i związana z nim cecha fenotypowa są dominujące , a drugi allel i związana z nim cecha są recesywne . Często dominujący allel koduje funkcjonalne białko, podczas gdy jego recesywny odpowiednik nie. Dominacja nie jest nieodłączną właściwością żadnego allelu lub fenotypu, ale po prostu opisuje jego związek z jednym lub większą liczbą innych alleli lub fenotypów; możliwe jest, aby jeden allel był jednocześnie dominujący nad drugim allelem, recesywny wobec trzeciego i kodominujący wobec czwartego. W skrócie genetyki dominujące allele są często reprezentowane przez pojedynczą wielką literę (np. „A”, w przeciwieństwie do recesywnego „a”).
- kompensacja dawki
- Każdy mechanizm, za pomocą którego organizmy neutralizują dużą różnicę w dawkowanie genu spowodowane obecnością różnej liczby chromosomów płciowych u różnych płci, wyrównując w ten sposób ekspresję genów sprzężonych z płcią, tak aby przedstawiciele każdej płci otrzymywali takie same lub podobne ilości produktów takich genów. Przykładem jest inaktywacja X u samic ssaków.
- podwójna helisa
- Kształt najczęściej przyjmowany przez dwuniciowe cząsteczki kwasu nukleinowego , przypominający drabinę skręconą wokół swojej długiej osi, której szczeble składają się z parzystych zasad nukleinowych . Ta drugorzędowa struktura jest najbardziej stabilną energetycznie konformacją dwuniciowych form zarówno DNA , jak i RNA w większości naturalnie występujących warunków, powstającą w wyniku pierwotnej struktury szkieletu fosfodiestrowego i ułożenia związanych z nim nukleotydów . W B-DNA , najczęstszy wariant DNA występujący w naturze, podwójna helisa ma prawoskrętny skręt z około 10 parami zasad na pełny obrót, a geometria molekularna skutkuje naprzemiennym wzorem „rowków” o różnych szerokościach (główny rowek i mniejszy rowek ) między równoległymi grzbietami.
- pęknięcie dwuniciowe (DSB)
- Utrata ciągłości szkieletu fosforanowo-cukrowego w obu niciach dwuniciowej cząsteczki DNA, w szczególności gdy dwa pęknięcia występują w miejscach położonych bezpośrednio naprzeciwko siebie lub bardzo blisko siebie na nici uzupełniające. Kontrastowa przerwa jednoniciowa .
- dwuniciowy
- Złożony z dwóch antyrównoległych , komplementarnych cząsteczek lub nici kwasów nukleinowych ( DNA-DNA , RNA-RNA lub hybryda DNA-RNA ), które są utrzymywane razem przez wiązania wodorowe między komplementarnymi zasadami nukleotydowymi każdej nici, znanymi jako parowanie zasad . Porównaj jednoniciowe .
- dwuniciowy DNA (dsDNA)
- Dowolna cząsteczka DNA złożona z dwóch antyrównoległych , komplementarnych polimerów dezoksyrybonukleotydowych , znanych jako nici , które są połączone ze sobą wiązaniami wodorowymi między komplementarnymi zasadami nukleotydowymi . Chociaż możliwe jest, aby DNA istniało jako pojedyncza nić , generalnie jest bardziej stabilna i bardziej powszechna w postaci dwuniciowej. W większości przypadków komplementarne parowanie zasad powoduje, że bliźniacze nici zwijają się wokół siebie w kształcie podwójnej helisy .
- dwuniciowy RNA (dsRNA)
- Dowolna cząsteczka RNA , która składa się z dwóch antyrównoległych , komplementarnych polimerów rybonukleotydowych , znanych jako nici , które są połączone ze sobą wiązaniami wodorowymi między komplementarnymi zasadami nukleotydowymi . Chociaż RNA zwykle występuje w postaci jednoniciowej , jest również zdolne do tworzenia dupleksów w taki sam sposób jak DNA ; przykładem jest mRNA z antysensowną wersją tego samego transkryptu, co skutecznie wycisza gen, z którego transkrybowano mRNA, zapobiegając translacji. Podobnie jak w przypadku dsDNA, parowanie zasad w dsRNA zwykle powoduje, że bliźniacze nici zwijają się wokół siebie w kształcie podwójnej helisy .
- regulacja w dół
- Każdy proces, naturalny lub sztuczny, który obniża poziom ekspresji określonego genu . Mówi się, że gen, którego ekspresja jest obserwowana na stosunkowo niskim poziomie (na przykład poprzez wykrycie niższych poziomów jego mRNA ) w jednej próbce w porównaniu z inną próbką, jest regulowany w dół . Zwiększenie kontrastu .
- poniżej
- W kierunku lub bliżej końca 3' łańcucha nukleotydów lub C-końca łańcucha peptydowego . Kontrast w górę .
- dsDNA
- Patrz dwuniciowy DNA .
- dsRNA
- Patrz dwuniciowy RNA .
- dupleks
- Zobacz dwuniciowy .
- duplikacja
- Wytwarzanie drugiej kopii części lub całości sekwencji nukleotydowej lub sekwencji aminokwasowej , naturalnie lub sztucznie, oraz zachowanie obu kopii; zwłaszcza gdy zarówno kopia, jak i oryginalna sekwencja są zachowane in situ w tej samej cząsteczce, często, ale niekoniecznie, przylegając do siebie. Zobacz też duplikacja genów , duplikacja chromosomów i powtórzenia .
- diada
- Patrz siostrzane chromatydy .

Dezoksyryboza różni się od rybozy tylko atomem węgla 2', gdzie ryboza ma atom tlenu, którego brakuje dezoksyrybozie (stąd jej nazwa).
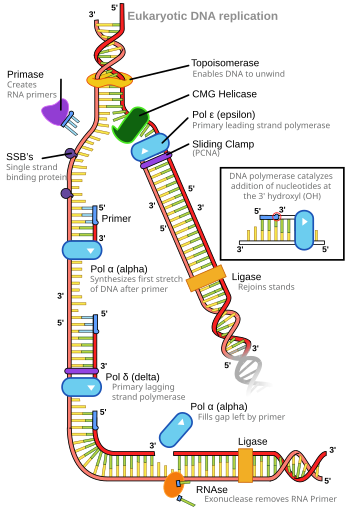
Diagram przedstawiający wiele składników replikacji DNA

Dwuniciowy DNA najczęściej występuje w kształcie podwójnej helisy .
mi
- genetyka ekologiczna
- Nauka genetyki w zakresie ekologii i przydatności naturalnych populacji organizmów żywych.
- elektroporacja
- Technika biologii molekularnej, w której do żywych komórek przykłada się silne pole elektryczne w celu tymczasowego zwiększenia przepuszczalności ich błon komórkowych, umożliwiając egzogennym kwasom nukleinowym, białkom lub związkom chemicznym łatwe przechodzenie przez błonę, a tym samym przedostawanie się do komórek. Jest to powszechna metoda przeprowadzania transformacji i transfekcji .
- wydłużenie
- Liniowy wzrost polimeru kwasu nukleinowego przez sekwencyjne dodawanie pojedynczych monomerów nukleotydowych do powstającej nici , np. podczas transkrypcji lub replikacji , zwłaszcza gdy zachodzi przez komplementarne parowanie z nicią matrycową . Termin ten jest często używany do opisania etapów niektórych technik laboratoryjnych, takich jak reakcja łańcuchowa polimerazy .
- emergenesis
- Jakość cech genetycznych wynika to ze specyficznej konfiguracji oddziałujących ze sobą genów , a nie tylko z ich kombinacji.
- endonukleaza
- Każdy enzym , którego aktywność polega na rozszczepianiu wiązań fosfodiestrowych w łańcuchu nukleotydów , włączając te, które rozszczepiają stosunkowo nieswoiście (bez względu na sekwencję ) i te, które rozszczepiają tylko w bardzo specyficznych sekwencjach (tak zwane endonukleazy restrykcyjne ). Gdy wymagane jest rozpoznanie określonej sekwencji, endonukleazy dokonują nacięć w środku sekwencji. Kontrast egzonukleaza .
- wzmacniacz
- Region DNA w pobliżu genu , który może być związany przez aktywator w celu zwiększenia ekspresji genu lub przez represor w celu zmniejszenia ekspresji.
- wzmacniacz RNA (eRNA)
- Podklasa długich niekodujących RNA transkrybowanych z regionów DNA zawierających wzmacniacz sekwencje. Ekspresja danego eRNA generalnie koreluje z aktywnością odpowiedniego wzmacniacza we wzmacnianiu transkrypcji jego genów docelowych, co sugeruje, że eRNA odgrywają aktywną rolę w regulacji trans genów w cis lub .
- epigenetyka
- epigenom
- episom
- 1. Inna nazwa plazmidu , zwłaszcza takiego, który jest zdolny do integracji z chromosomem .
- 2. U eukariontów każdy niezintegrowany kolisty DNA pozachromosomalny cząsteczka, która jest stabilnie utrzymywana i replikowana w jądrze jednocześnie z resztą komórki gospodarza. Takie cząsteczki mogą obejmować genomy wirusowe, plazmidy bakteryjne i nieprawidłowe fragmenty chromosomów.
- epistaza
- Zbiorowe działanie wielu genów oddziałujących na siebie podczas ekspresji genów . Epistaza, będąca formą działania genów, może być addytywna lub multiplikatywna pod względem wpływu na określone cechy fenotypowe .
- ergosom
- Zobacz polisom .
- euchromatyna
- Stosunkowo otwarta, lekko zwarta forma chromatyny , w której DNA jest tylko sporadycznie związane w nukleosomach , a zatem jest szeroko dostępna do wiązania i manipulacji przez białka i inne cząsteczki. Euchromatyczne regiony genomu są często wzbogacone w geny i aktywnie przechodzą transkrypcję , w przeciwieństwie do heterochromatyny , która jest stosunkowo uboga w geny, bogata w nukleosomy i mniej dostępna dla maszynerii transkrypcyjnej.
- euploidia
- Stan komórki lub organizmu z nieprawidłową liczbą kompletnych zestawów chromosomów , prawdopodobnie z wyłączeniem chromosomów płciowych . Euploidia różni się od aneuploidii , w której komórka lub organizm ma nieprawidłową liczbę jednego lub więcej określonych pojedynczych chromosomów.
- ewolucja
- Zmiana dziedzicznych cech populacji biologicznych w kolejnych pokoleniach. W najbardziej tradycyjnym sensie następuje to poprzez zmiany częstości alleli w puli genowej populacji .
- wycięcie
- Enzymatyczne usunięcie sekwencji polinukleotydowej z jednej lub większej liczby nici kwasu nukleinowego lub sekwencji polipeptydowej z białka , zwykle obejmujące zarówno rozerwanie cząsteczki polimeru w dwóch miejscach , jak i późniejsze ponowne połączenie dwóch punktów przerwania po sekwencji między zostały one usunięte. Termin ten może być używany do opisania szerokiej gamy procesów wykonywanych przez różne enzymy, w tym większość szlaków splicingu i naprawy DNA .
- egzon
- Cały zestaw eksonów w obrębie określonego genomu , w tym nieulegające translacji regiony dojrzałych mRNA, jak również regiony kodujące .
- egzon
- Dowolna część genu , która koduje część ostatecznego dojrzałego informacyjnego RNA wytwarzanego przez ten gen po usunięciu intronów przez alternatywne składanie . Termin ten odnosi się zarówno do sekwencji istniejącej w cząsteczce DNA, jak i do odpowiedniej sekwencji w transkryptach RNA.
- egzonukleaza
- z pominięciem eksonu
- Dowolny enzym , którego aktywność polega na rozszczepianiu wiązań fosfodiestrowych w łańcuchu nukleotydów , w tym takich, które rozszczepiają się dopiero po rozpoznaniu określonej sekwencji (tzw. egzonukleazy restrykcyjne ). Egzonukleazy dokonują cięć na 3' lub 5' sekwencji (a nie w środku, jak w przypadku endonukleaz ).
- kompleks egzosomów
- Wewnątrzkomórkowy kompleks wielobiałkowy pełniący funkcję degradacji różnych typów cząsteczek RNA .
- wektor ekspresji
- Typ wektora , zwykle plazmidowy lub wirusowy , zaprojektowany specjalnie do ekspresji wstawki transgenu w komórce docelowej, a nie do innych celów, takich jak klonowanie .
- ekspresyjność
- Dla danego genotypu powiązanego ze zmiennym fenotypem niebinarnym , odsetek osobników z tym genotypem, które wykazują lub wyrażają fenotyp w określonym stopniu, zwykle podawany w procentach. Ze względu na wiele złożonych interakcji, które regulują ekspresję genów , ten sam allel może wytwarzać szeroką gamę możliwych fenotypów o różnych cechach lub stopniach u różnych osobników; w takich przypadkach można powiedzieć, że zarówno fenotyp, jak i genotyp wykazują zmienną ekspresję . Ekspresyjność próbuje ilościowo określić zakres możliwych poziomów zmienności fenotypowej w populacji osobników wyrażających fenotyp będący przedmiotem zainteresowania. Porównaj penetrację .
- eksteina
- Każda część sekwencji aminokwasowej , która jest zachowana w polipeptydzie prekursorowym , tj. nie jest wycięta przez potranslacyjny splicing białka , a zatem jest obecna w dojrzałym białku , analogicznie do eksonów transkryptów RNA. kontrast . _
- DNA pozachromosomalne
- Każdy DNA , który nie występuje w chromosomach lub w jądrze komórki, a zatem nie jest DNA genomowym . Może to obejmować DNA zawarte w plazmidach lub organellach , takich jak mitochondria lub chloroplasty , lub, w najszerszym znaczeniu, DNA wprowadzone przez infekcję wirusową . DNA pozachromosomalne zwykle wykazuje znaczące różnice strukturalne z DNA jądrowym w tym samym organizmie.

Mapa plazmidu wektora ekspresyjnego o długości 3756 pz , użytego do ekspresji transgenu wytwarzającego białko zielonej fluorescencji (GFP). Wektor zawiera również gen dla represora lac (lacI) i gen nadający oporność na antybiotyk kanamycynę (KanR), jak również różne promotory kierujące ekspresją tych genów.
F
- ekspresja fakultatywna
- Transkrypcja genu tylko w razie potrzeby, w przeciwieństwie do ekspresji konstytutywnej , w której gen jest transkrybowany w sposób ciągły . Gen, który jest transkrybowany w razie potrzeby, nazywany jest genem fakultatywnym .
- fluorescencyjna hybrydyzacja in situ (FISH)
- czapeczka z 5 prime
- Zobacz czapeczka 5' .
- koniec z pięcioma pierwszymi
- Zobacz koniec 5' .
- nieulegający translacji region pięciu liczb pierwszych
- Zobacz region nieulegający translacji 5' .
- fiksacja
- Proces, w którym pojedynczy allel dla określonego genu z wieloma różnymi allelami zwiększa częstotliwość w danej populacji, tak że zostaje trwale ustalony ze 100% częstotliwością - to znaczy jedynym allelem w tym locus w puli genowej populacji . W przypadku braku mutacji i przewagi heterozygoty , każdy dany allel ostatecznie ma zostać trwale utrwalony w stosunku do wszystkich innych wariantów lub całkowicie utracony z populacji, chociaż czas, jaki to zajmie, zależy od presje selekcyjne i przypadkowe fluktuacje częstości alleli.
- forwardgenetics
- mutacja w
- przód mutacja z przesunięciem ramki odczytu
- Typ mutacji w sekwencji kwasu nukleinowego spowodowany insercją lub delecją pewnej liczby nukleotydów , która nie jest podzielna przez trzy. Ze względu na trypletową naturę, dzięki której nukleotydy kodują aminokwasy, mutacja tego rodzaju powoduje przesunięcie w ramce odczytu sekwencji nukleotydów, co skutkuje sekwencją kodony poniżej miejsca mutacji są całkowicie różne od oryginału.
- Funkcjonalne Towarzystwo Danych Genomiki (FGED).
- Organizacja, która współpracuje z innymi „w celu opracowania standardów jakości, adnotacji i wymiany danych z badań biologicznych”, a także narzędzi programowych ułatwiających ich wykorzystanie.
G
- Paski G
- Technika stosowana w cytogenetyce do uzyskiwania widocznego kariotypu poprzez barwienie skondensowanych chromosomów barwnikiem Giemsy . Barwienie daje spójne i możliwe do zidentyfikowania wzory ciemnych i jasnych „prążków” w regionach chromatyny , co pozwala na łatwe rozróżnienie określonych chromosomów.
- gameta
- Komórka haploidalna będąca produktem mejotycznym progenitorowej komórki zarodkowej i końcowym produktem linii zarodkowej w rozmnażające się płciowo organizmy wielokomórkowe. Gamety to środki, za pomocą których organizm przekazuje informacje genetyczne swojemu potomstwu; podczas zapłodnienia dwie gamety (po jednej od każdego rodzica) łączą się w jedną diploidalną zygotę .
- gDNA
- Zobacz genomowe DNA .
- gen
- Dowolny segment lub zestaw segmentów cząsteczki kwasu nukleinowego , który zawiera informacje niezbędne do wytworzenia funkcjonalnego transkryptu RNA w kontrolowany sposób. W żywych organizmach geny są często uważane za podstawowe jednostki dziedziczności i są zazwyczaj zakodowane w DNA . Konkretny gen może mieć wiele różnych wersji lub alleli , a pojedynczy gen może skutkować produktem genu , który wpływa na wiele różnych fenotypów .
- dawkowanie genu
- Liczba kopii określonego genu obecnego w genomie . Dawka genu bezpośrednio wpływa na ilość produktu genu, który komórka jest w stanie wyrazić, chociaż wyewoluowało wiele kontroli, które ściśle regulują ekspresję genów . Zmiany w dawkowaniu genów spowodowane mutacjami obejmują zmiany liczby kopii .
- duplikacja
- genu napędowego
- Typ mutacji definiowany jako dowolna duplikacja regionu DNA zawierającego gen . Porównaj duplikację chromosomów .
- ekspresja genu
- Zestaw procesów, w których informacja zakodowana w genie jest wykorzystywana do syntezy produktu genu , takiego jak białko lub niekodujący RNA , lub w inny sposób udostępniana w celu wpływania na jeden lub więcej fenotypów . Kanonicznie, pierwszym krokiem jest transkrypcja , która daje informacyjnego RNA komplementarna do cząsteczki DNA , w której kodowany jest gen; w przypadku genów kodujących białka, drugim etapem jest translacja , w której informacyjny RNA jest odczytywany przez rybosom w celu wytworzenia białka . Informacje zawarte w sekwencji DNA nie muszą jednak koniecznie podlegać transkrypcji i translacji, aby wywrzeć wpływ na zdarzenia molekularne; szersze definicje obejmują ogromną różnorodność innych sposobów wyrażania informacji genetycznej.
- Gene Expression Omnibus (GEO)
- Baza danych o dużej przepustowości genomika funkcjonalna i dane dotyczące ekspresji genów pochodzące z eksperymentalnych chipów i sekwencjonowania nowej generacji i zarządzane przez National Center for Biotechnology Information .
- fuzja genów
- Połączenie dwóch lub więcej wcześniej niezależnych genów, które kodują różne produkty genowe, w wyniku naturalnej mutacji lub rekombinacji technik laboratoryjnych, w taki sposób, że stają się one przedmiotem kontroli tych samych systemów regulacyjnych . Otrzymana sekwencja hybrydowa jest znana jako gen fuzyjny .
- mapowanie genów
- Dowolna z różnorodnych metod używanych do dokładnego określenia lokalizacji określonego genu w cząsteczce DNA (takiej jak chromosom) i/lub odległości fizycznych lub odległości między nim a innymi genami.
- gen Gen badany w ramach eksperymentu naukowego, zwłaszcza takiego, który jest przedmiotem zainteresowania techniki inżynierii genetycznej , takiej jak klonowanie .
- będący przedmiotem zainteresowania (GOI)
- pula genów
- Suma wszystkich różnych alleli wspólnych dla członków jednej populacji.
- produkt genu
- Dowolny materiał biochemiczny powstały w wyniku ekspresji genu , najczęściej interpretowany jako funkcjonalny transkrypt mRNA wytwarzany w wyniku transkrypcji genu lub w pełni skonstruowane białko wytwarzane w wyniku translacji transkryptu, chociaż niekodujące cząsteczki RNA, takie jak transfer RNA można również uznać za produkty genów. Pomiar ilości danego produktu genu, który jest wykrywalny w komórce lub tkance, jest czasami używany do wywnioskowania, jak aktywny jest odpowiedni gen.
- regulacja genów
- Szeroka gama mechanizmów wykorzystywanych przez komórki do kontrolowania aktywności ich genów, w szczególności do zezwalania, blokowania, zwiększania lub zmniejszania produkcji lub ekspresji określonych produktów genów , takich jak RNA lub białka . Regulacja genów zwiększa wszechstronność i zdolności adaptacyjne organizmu, umożliwiając jego komórkom ekspresję różnych produktów genów, gdy wymagają tego zmiany w środowisku. W organizmach wielokomórkowych regulacja ekspresji genów napędza również różnicowanie komórek i morfogenezę w zarodku , umożliwiając tworzenie różnorodnych typów komórek z tego samego genomu .
- wyciszanie genów
- Dowolny mechanizm regulacji genów , który drastycznie zmniejsza lub całkowicie uniemożliwia ekspresję określonego genu. Wyciszanie genów może zachodzić naturalnie podczas transkrypcji lub translacji . Techniki laboratoryjne często wykorzystują naturalne mechanizmy wyciszania w celu uzyskania wyciszenia genów .
- terapia genowa
- Wstawienie funkcjonalnego lub typu dzikiego genu lub części genu do organizmu (zwłaszcza pacjenta) w celu skorygowania defektu genetycznego , albo przez bezpośrednie zastąpienie wadliwego genu, albo przez uzupełnienie drugim, funkcjonalnym wersja.
- pułapkowanie genów
- Wysoka przepustowość technologia stosowana do jednoczesnej inaktywacji, identyfikacji i zgłaszania ekspresji docelowego genu w genomie ssaka poprzez wprowadzenie mutacji insercyjnej składającej się z bezpromotorowego genu reporterowego i/lub selekcyjnego markera genetycznego otoczonego przez miejsce splicingowe w górę i w dół poliadenylowaną sekwencję terminacyjną .
- pokolenie
- 1. W danym organizmie pojedynczy cykl reprodukcyjny lub fazę między dwoma następującymi po sobie zdarzeniami reprodukcyjnymi, tj. między reprodukcją pojedynczego organizmu a reprodukcją potomstwa tej reprodukcji; lub rzeczywisty lub średni czas potrzebny do zakończenia pojedynczego cyklu reprodukcyjnego, albo dla określonej linii , albo dla populacji lub gatunku jako całości.
- 2. W danej populacji osobniki (często, ale niekoniecznie żyjące w tym samym czasie), które są w równym stopniu oddalone od danego wspólnego przodka z powodu tej samej liczby zdarzeń reprodukcyjnych, które wystąpiły między nimi a przodkiem.
- stowarzyszenie genetyczne
- Współwystępowanie w populacji jednego lub więcej alleli lub genotypów z określoną cechą fenotypową częściej, niż można by się spodziewać na podstawie samego przypadku; taka korelacja statystyczna może być wykorzystana do wywnioskowania, że allele lub genotypy są odpowiedzialne za wytworzenie danego fenotypu.
- tło genetyczne
- kod genetyczny
- Zestaw reguł, według których informacje zakodowane w kwasach nukleinowych są tłumaczone na białka przez żywe komórki . Zasady te określają, w jaki sposób sekwencje nukleotydów zwane kodonami określają, który aminokwas zostanie dodany jako następny podczas syntezy białka . Zdecydowana większość żywych organizmów używa tego samego kodu genetycznego (czasami określanego jako „standardowy” kod genetyczny ), ale istnieją warianty kodów .
- poradnictwo genetyczne
- Proces udzielania porad osobom lub rodzinom dotkniętym lub zagrożonym rozwojem zaburzeń genetycznych aby pomóc im zrozumieć i dostosować się do fizjologicznych, psychologicznych i rodzinnych implikacji wkładu genetycznego w chorobę. Poradnictwo genetyczne łączy badania genetyczne , genealogię genetyczną i epidemiologię genetyczną .
- zaburzenie genetyczne Każda choroba, schorzenie lub inny problem zdrowotny bezpośrednio spowodowany przez jedną lub więcej nieprawidłowości w
- genomie organizmu, które są wrodzone (obecne przy urodzeniu) i nie nabyte w późniejszym okresie życia. Przyczyny mogą obejmować mutację jednego lub więcej genów lub aberracja chromosomalna , taka jak aneuploidia określonego chromosomu. Odpowiedzialna mutacja może wystąpić spontanicznie podczas rozwoju embrionalnego lub może zostać odziedziczona po jednym lub obojgu rodzicach, w którym to przypadku zaburzenie genetyczne jest również klasyfikowane jako zaburzenie dziedziczne . Chociaż sama nieprawidłowość jest obecna przed urodzeniem, rzeczywista choroba, którą powoduje, może rozwinąć się dopiero znacznie później w życiu; niektóre zaburzenia genetyczne niekoniecznie gwarantują ewentualną chorobę, ale po prostu zwiększają ryzyko jej rozwoju przez organizm.
- odległość genetyczna
- Miara rozbieżności genetycznej między gatunkami, populacjami w obrębie gatunku lub osobnikami, stosowana zwłaszcza w filogenetyce do wyrażania czasu, jaki upłynął od istnienia wspólnego przodka lub stopnia zróżnicowania sekwencji DNA obejmujących genomy każdego populacji lub osobnika.
- różnorodność genetyczna
- Całkowita liczba cech lub cech genetycznych w składzie genetycznym populacji, gatunku lub innej grupy organizmów. Jest często używany jako miara zdolności adaptacyjnych grupy do zmieniających się środowisk. zmienności genetycznej , choć różni się od niej .
- dryf genetyczny
- Zmiana częstości występowania istniejącego allelu w populacji spowodowana losową zmiennością rozmieszczenia alleli z pokolenia na pokolenie. Dryf genetyczny jest często interpretowany jako rola, jaką losowy przypadek odgrywa w określaniu, czy dany allel staje się mniej lub bardziej powszechny z każdym pokoleniem, niezależnie od wpływu doboru naturalnego . Taka przypadkowość może spowodować, że pewne allele, nawet korzystne, znikną całkowicie z puli genów , zmniejszając w ten sposób zmienność genetyczną lub może spowodować, że początkowo rzadkie allele, nawet neutralne lub szkodliwe, staną się znacznie częstsze lub nawet utrwalone .
- Inżynieria genetyczna
- Bezpośrednia, celowa manipulacja materiałem genetycznym organizmu przy użyciu dowolnej z różnorodnych metod biotechnologicznych, w tym wprowadzanie lub usuwanie genów , przenoszenie genów w obrębie gatunku i między gatunkami, mutacja istniejących sekwencji oraz konstruowanie nowych sekwencji przy użyciu sztucznych synteza genów . Inżynieria genetyczna obejmuje szeroki zestaw technologii, za pomocą których skład genetyczny poszczególnych komórek, tkanek lub całych organizmów może być zmieniany w różnych celach, zwykle w celu zbadania funkcji i ekspresji poszczególnych genów, do produkcji hormonów, szczepionek i innych leków oraz do tworzenia genetycznie zmodyfikowanych organizmów do użytku w badaniach i rolnictwie.
- epidemiologia genetyczna
- Badanie roli odgrywanej przez czynniki genetyczne w determinowaniu zdrowia i choroby, w szczególności poprzez interakcję czynników genetycznych z czynnikami środowiskowymi, zazwyczaj obserwowaną u genetycznie spokrewnionych osobników, często rodzin lub rodów, ale także populacji i subpopulacji.
- genealogia genetyczna
- Wykorzystanie genealogicznych badań DNA w połączeniu z tradycyjnymi badaniami genealogicznymi metody wnioskowania o poziomie i rodzaju pokrewieństwa genetycznego między jednostkami, znajdowania przodków i konstruowania drzew genealogicznych , genogramów lub innych wykresów genealogicznych.
- autostopem genetycznym
- Rodzaj połączonej selekcji, w którym pozytywna selekcja allelu przechodzącego selektywne przemiatanie powoduje, że allele różnych genów w pobliskich loci również zmieniają częstotliwość , umożliwiając im „podjazd” do utrwalenia wraz z pozytywnie wybranym allelem. Jeśli selekcja w pierwszym locus jest wystarczająco silna, neutralne lub nawet lekko szkodliwe allele w obrębie tej samej grupy sprzężeń mogą podlegać tej samej pozytywnej selekcji, ponieważ fizyczna odległość między pobliskimi loci jest na tyle mała, że rekombinacja zdarzenie między nimi jest mało prawdopodobne. Genetyczny autostop jest często uważany za przeciwieństwo selekcji w tle .
- marker genetyczny
- Specyficzny, łatwy do zidentyfikowania i zwykle wysoce polimorficzny gen lub inna sekwencja DNA ze znaną lokalizacją na chromosomie , która może być wykorzystana do zidentyfikowania posiadającego go osobnika lub gatunku.
- rekombinacja genetyczna
- Wszelka reasortacja lub wymiana materiału genetycznego w obrębie pojedynczego organizmu lub między osobnikami tego samego lub różnych gatunków, zwłaszcza ta, która tworzy zmienność genetyczna . W najszerszym znaczeniu termin ten obejmuje zróżnicowaną klasę naturalnie występujących mechanizmów, za pomocą których sekwencje kwasu nukleinowego są kopiowane lub fizycznie przenoszone do różnych środowisk genetycznych, w tym rekombinacja homologiczna podczas mejozy lub mitozy lub jako normalna część naprawy DNA ; poziome zdarzenia transferu genów, takie jak koniugacja bakteryjna , transdukcja wirusowa lub transformacja ; lub błędy w replikacji DNA lub podział komórek. Sztuczna rekombinacja ma kluczowe znaczenie dla wielu inżynierii genetycznej , które wytwarzają rekombinowane DNA .
- redundancja genetyczna
- Nadmiarowe kodowanie dwóch lub więcej różnych produktów genów , które ostatecznie pełnią tę samą funkcję biochemiczną. Mutacje w jednym z tych genów mogą mieć mniejszy wpływ na sprawność, niż można by się spodziewać, ponieważ nadmiarowe geny często kompensują utratę funkcji i uniemożliwiają jej uzyskanie .
- genetyczna sieć regulacyjna (GRN)
- Wykres przedstawiający złożoność regulacyjną ekspresji genów . Wierzchołki (węzły) są reprezentowane przez różne elementy regulacyjne i produkty genów, podczas gdy krawędzie (połączenia) są reprezentowane przez ich interakcje. Te struktury sieci reprezentują również zależności funkcjonalne, przybliżając tempo transkrypcji genów .
- badania genetyczne
- Szeroka klasa różnych procedur używanych do identyfikacji cech poszczególnych chromosomów, genów lub białek danej osoby w celu określenia pochodzenia lub przodków, zdiagnozowania podatności na choroby dziedziczne lub wykrycia zmutowanych alleli związanych ze zwiększonym ryzykiem rozwoju zaburzeń genetycznych . Testy genetyczne są szeroko stosowane w medycynie, rolnictwie i badaniach biologicznych.
- zmienność genetyczna
- Powstawanie lub obecność osobników różniących się genotypem w obrębie populacji lub innej grupy organizmów, w przeciwieństwie do osobników z różnicami uwarunkowanymi środowiskowo, które powodują jedynie tymczasowe, niedziedziczne zmiany fenotypu . Pomijając inne ograniczenia, populacja o dużej zmienności genetycznej ma większy potencjał udanej adaptacji do zmieniających się warunków środowiskowych niż populacja o niskiej zmienności genetycznej. Zmienność genetyczna jest podobna do różnorodności genetycznej , choć różni się od niej .
- wariacja genetyczna
- Różnice genetyczne zarówno w obrębie populacji, jak i między populacjami, gatunkami lub innymi grupami organizmów. Często jest wizualizowany jako różnorodność różnych alleli w pulach genów różnych populacji.
- organizm zmodyfikowany genetycznie (GMO)
- Każdy organizm, którego materiał genetyczny został zmieniony przy użyciu technik inżynierii genetycznej , w szczególności w sposób, który nie zachodzi naturalnie w wyniku krzyżowania lub naturalnej rekombinacji genetycznej .
- genetyka
- Dziedzina biologii zajmująca się badaniem genów , zmienność genetyczna i dziedziczność w organizmach żywych.
- równowaga genowa
- Mechanizm determinacji płci , który zależy od stosunku liczby chromosomów X (X) do liczby zestawów autosomów (A). Mężczyźni rozwijają się, gdy stosunek X/A wynosi 0,5 lub mniej, kobiety, gdy wynosi on 1,0 lub więcej, a interpłciowość rozwija się, gdy wynosi od 0,5 do 1,0.
- genom
- Cały zestaw materiału genetycznego zawarty w chromosomach organizmu, organelli lub wirus . Termin ten jest również używany w odniesieniu do zbiorowego zestawu loci genetycznych wspólnych dla każdego członka populacji lub gatunku, niezależnie od różnych alleli , które mogą być obecne w tych loci u różnych osobników.
- wielkość genomu
- Całkowita ilość DNA zawarta w jednej kopii genomu , zazwyczaj mierzona masą (w pikogramach lub daltonach ) lub całkowitą liczbą par zasad (w kilozasadach lub megazasadach ). Dla diploidalnych , rozmiar genomu jest często używany zamiennie z wartością C.
- genomowe DNA (gDNA)
- DNA zawarte w chromosomach , w przeciwieństwie do pozachromosomalnego DNA zawartego w oddzielnych strukturach, takich jak plazmidy lub organelle , takie jak mitochondria lub chloroplasty .
- imprinting genomowy
- Zjawisko epigenetyczne powodujące ekspresję genów w sposób zależny od konkretnego rodzica, od którego gen został odziedziczony . Występuje, gdy ślady epigenetyczne, takie jak DNA lub metylacja histonów zostały ustalone lub „odciśnięte” w komórkach rozrodczych organizmu rodzicielskiego, a następnie utrzymywane przez podziały komórkowe w komórkach somatycznych potomstwa organizmu; w rezultacie gen odziedziczony po ojcu u potomstwa może ulegać innej ekspresji niż inna kopia tego samego genu odziedziczona po matce.
- genomika
- Interdyscyplinarna dziedzina zajmująca się badaniem struktury, funkcji, ewolucji, mapowania i edytowania całych genomów , w przeciwieństwie do poszczególnych genów .
- genotoksyczność
- Zdolność pewnych czynników chemicznych do powodowania uszkodzeń materiału genetycznego w żywej komórce (np. poprzez pęknięcia jedno- lub dwuniciowe, sieciowanie lub mutacje punktowe ), co może, ale nie musi, skutkować trwałą mutacją . Chociaż wszystkie mutageny są genotoksyczne, nie wszystkie związki genotoksyczne są mutagenne.
- genotyp
- Cały zestaw alleli obecnych w genomie danego osobnika , który daje początek fenotypowi osobnika .
- częstotliwość genotypów
- Częstotliwość lub odsetek populacji o danym genotypie . Porównaj częstość alleli .
- genotypowanie
- Proces określania różnic w genotypie osobnika poprzez badanie sekwencji DNA w genomie osobnika za pomocą testów biologicznych i porównywanie ich z sekwencjami innego osobnika lub sekwencją referencyjną.
- komórka rozrodcza
- Każda komórka , z której powstają gamety organizmu rozmnażającego się płciowo . Komórki rozrodcze są naczyniami dla materiału genetycznego, który ostatecznie zostanie przekazany potomstwu organizmu i zazwyczaj odróżnia się je od komórek somatycznych , które są całkowicie oddzielone od linii zarodkowej .
- linia zarodkowa
- 1. W organizmach wielokomórkowych populacja komórek , które są zdolne do przekazywania swojego materiału genetycznego potomstwu organizmu i tym samym (przynajmniej teoretycznie) różnią się od komórek somatycznych , które nie mogą przekazywać swojego materiału genetycznego z wyjątkiem własnego bezpośredniego mitotyczny komórki potomne. Komórki linii zarodkowej nazywane są komórkami rozrodczymi .
- 2. Linia komórek rozrodczych obejmująca wiele pokoleń, zawierająca materiał genetyczny przekazany osobnikowi przez przodków.
- Pudełko Goldberg-Hogness
- Zobacz pudełko TATA .
- gRNA
- Patrz przewodnik RNA .
- guanina ( G )
- Nukleozasada purynowa stosowana jako jedna z czterech standardowych zasad nukleinowych zarówno w DNA , jak i RNA Cząsteczki. Guanina tworzy parę zasad z cytozyną .
- zawartość guaniny-cytozyny
- Proporcja zasad azotowych w kwasie nukleinowym , które są guaniną ( G ) lub cytozyną ( C ), zwykle wyrażana w procentach. Cząsteczki DNA i RNA o wyższej zawartości GC są na ogół bardziej termostabilne niż te o niższej zawartości GC ze względu na interakcje molekularne zachodzące podczas układania zasad.
- guanozyna ( G , Guo)
- Jeden z czterech standardowych nukleozydów stosowanych w cząsteczkach RNA , składający się z zasada guaniny z azotem N9 związanym z atomem węgla C1 cukru rybozy . Guanina związana z dezoksyrybozą jest znana jako deoksyguanozyna , która jest wersją stosowaną w DNA .
- przewodnik RNA (gRNA)
H
- spinka do włosów
- Zobacz pętelkę .
- haplodiploidia
- System determinacji płci, w którym płeć pojedynczego organizmu jest określona przez liczbę zestawów chromosomów , które posiada: potomstwo, które rozwija się z zapłodnionych jaj, jest diploidalne i żeńskie , podczas gdy potomstwo, które rozwija się z niezapłodnionych jaj, jest haploidalne i męskie, z o połowę mniej chromosomów niż kobiety. Haplodiploidia jest wspólna dla wszystkich członków rzędu owadów Hymenoptera i kilku innych taksonów owadów.
- haplogrupa
- haploidalny
- (komórki lub organizmu) Posiadanie jednej kopii każdego chromosomu , przy czym żadna kopia nie jest częścią pary. Porównaj diploidalny i poliploidalny .
- haploinsufficiency
- haplotyp
- Zestaw alleli w pojedynczym organizmie, które zostały odziedziczone razem od jednego rodzica.
- helikaza
- Dowolna klasa białek motorycznych zależnych od ATP , które poruszają się kierunkowo wzdłuż szkieletu DNA i katalizują rozdzielanie dwóch komplementarnych nici dwuniciowe , umożliwiające zajście wielu różnych procesów życiowych, np. transkrypcji , replikacji i naprawy .
- hemizygotyczny
- W organizmach diploidalnych , mający tylko jeden allel w danym locus genetycznym (gdzie normalnie byłyby dwa). Hemizygotyczność można zaobserwować, gdy w normalnie diploidalnej komórce lub organizmie występuje tylko jedna kopia chromosomu lub gdy usunięto fragment chromosomu zawierający jedną kopię allelu , lub gdy gen znajduje się na chromosomie płciowym w przypadku płci heterogametycznej (w której chromosomy płciowe nie występują w dopasowanych parach); na przykład u mężczyzn z normalnymi chromosomami prawie wszystkie sprzężone z chromosomem X są uważane za hemizygotyczne, ponieważ istnieje tylko jeden chromosom X , a na chromosomie Y istnieje kilka takich samych genów .
- dziedziczność
- Przechowywanie, przekazywanie i wyrażanie informacji molekularnej w organizmach biologicznych, objawiające się przekazywaniem cech fenotypowych z rodziców na ich potomstwo , poprzez rozmnażanie płciowe lub bezpłciowe . Mówi się, że potomne komórki lub organizmy dziedziczą informację genetyczną swoich rodziców.
- dziedziczność
- 1. Zdolność do dziedziczenia .
- 2. Statystyka stosowana w genetyce ilościowej , która szacuje proporcję zmienności w ramach danego cecha fenotypowa , która wynika ze zmienności genetycznej między osobnikami w określonej populacji. Dziedziczność szacuje się, porównując poszczególne fenotypy blisko spokrewnionych osobników w populacji.
- heterochromatyna
- heterochromosom
- Zobacz allosom .
- heterogeniczny jądrowy RNA (hnRNA)
- heterologiczna ekspresja
- Ekspresja obcego genu lub dowolnej innej obcej sekwencji DNA w organizmie gospodarza, która nie zawiera naturalnie tego samego genu. Wstawienie obcego transgenów do heterologicznych gospodarzy przy użyciu rekombinowanych wektorów jest powszechną biotechnologiczną metodą badania struktury i funkcji genów.
- heterozja
- Ulepszona lub zwiększona funkcja lub jakość jakiejkolwiek cechy biologicznej u potomstwa hybrydy , w odniesieniu do tej samej cechy u jego genetycznie różnych rodziców. Jeśli jedna lub więcej cech rodziców jest zauważalnie wzmocniona u potomstwa w wyniku mieszania wkładu genetycznego rodziców, mówi się, że potomstwo jest heterotyczne .
- heterozygota
- W organizmie diploidalnym , mająca dwa różne allele w danym locus genetycznym . W skrócie genetyki, genotypy heterozygotyczne są reprezentowane przez parę niedopasowanych liter lub symboli, często wielką literę (oznaczającą dominujący allel) i małą literę (oznaczającą allel recesywny ), na przykład „Aa” lub „Bb”. Porównaj homozygoty .
- histon
- o dużej przepustowości
- Dowolne białko z klasy silnie zasadowych białek odpowiedzialnych za upakowanie jądrowego DNA w jednostki strukturalne zwane nukleosomami w komórkach eukariotycznych. Histony są głównymi białkowymi składnikami chromatyny , gdzie łączą się w kompleksy, które działają jak „szpule”, wokół których owija się liniowa cząsteczka DNA. Odgrywają one główną rolę w i ekspresji genów .
- hnRNA
- Patrz heterogeniczny jądrowy RNA .
- holocentryczny
- (liniowego chromosomu lub fragmentu chromosomu) nieposiadający pojedynczego centromeru , ale raczej liczne miejsca składania kinetochoru rozproszone wzdłuż całej długości chromosomu. Podczas podziału komórki chromatydy chromosomów holocentrycznych rozsuwają się równolegle i nie tworzą klasycznych struktur w kształcie litery V typowych dla chromosomów monocentrycznych .
- chromosomy homologiczne
- homeoboksów
- Zestaw dwóch pasujących chromosomów , jednego matczynego i jednego ojcowskiego, które łączą się ze sobą wewnątrz jądra podczas mejozy . Mają te same geny w tych samych loci , ale mogą mieć różne allele .
- rekombinacja homologiczna
- Rodzaj rekombinacji genetycznej , w której sekwencje nukleotydowe są wymieniane między dwiema podobnymi lub identycznymi („homologicznymi”) cząsteczkami DNA , zwłaszcza ta, która zachodzi między chromosomami homologicznymi . Termin ten może odnosić się do rekombinacji, która zachodzi jako część dowolnego z wielu różnych procesów komórkowych, najczęściej naprawy DNA lub krzyżowania chromosomów podczas mejozy u eukariontów i poziomego transferu genów u prokariotów. Porównaj rekombinację niehomologiczną .
- homozygota
- W organizmie diploidalnym , mająca dwa identyczne allele w danym locus genetycznym . W skrócie genetyki, genotypy homozygotyczne są reprezentowane przez parę pasujących liter lub symboli, takich jak „AA” lub „aa”. Porównaj heterozygoty .
- poziomy transfer genów (HGT)
- Dowolny proces, w którym materiał genetyczny jest przenoszony między organizmami jednokomórkowymi i/lub wielokomórkowymi, inny niż poprzez transmisję wertykalną z rodzica na potomstwo, np. koniugacja bakteryjna .
- gen porządkowy
- Dowolny gen konstytutywny , który podlega transkrypcji na względnie stałym poziomie w wielu lub wszystkich znanych stanach i typach komórek. produkty _ genów porządkowych zazwyczaj odgrywa kluczową rolę w utrzymaniu integralności komórkowej i podstawowych funkcji metabolicznych. Ogólnie przyjmuje się, że na ich ekspresję nie mają wpływu warunki eksperymentalne ani patologiczne.
- Geny Hox
- Podzbiór wysoce konserwatywnych genów zawierających homeoboks , które są niezbędne do prawidłowej organizacji planu ciała w rozwijających się embrionach zwierzęcych, zapewniając tworzenie się prawidłowych struktur we właściwych miejscach. Geny Hox są zwykle ułożone na chromosomie w tandemowych macierzach i ulegają ekspresji sekwencyjnie podczas rozwoju, z sekwencją aktywacji genów odpowiadającą fizycznemu układowi tkanek, w których ulegają ekspresji wzdłuż osi przednio-tylnej organizmu.
- Human Genome Project (HGP)
- Międzynarodowy projekt badań naukowych, którego celem jest sekwencjonowanie całego chromosomalnego DNA oraz identyfikacja i mapowanie wszystkich genów w ludzkich komórkach, a ostatecznie złożenie kompletnego genomu referencyjnego dla gatunku ludzkiego. Projekt został rozpoczęty w 1990 roku przez konsorcjum agencji federalnych, uniwersytetów i instytucji badawczych i został uznany za zakończony w 2003 roku. Ponieważ każdy człowiek ma unikalny genom, gotowy genom referencyjny jest mozaiką sekwencji uzyskanych przez pobranie próbek DNA z tysięcy osób na całym świecie i nie reprezentuje żadnej pojedynczej osoby.
- hybryda
- Potomstwo powstałe w wyniku połączenia cech dwóch organizmów różnych rodzajów , gatunków , ras lub odmian poprzez rozmnażanie płciowe . Mieszańce mogą występować naturalnie lub sztucznie, np. podczas selektywnej hodowli udomowionych zwierząt i roślin. Bariery reprodukcyjne zwykle zapobiegają hybrydyzacji między daleko spokrewnionymi organizmami lub przynajmniej zapewniają, że potomstwo hybrydy jest bezpłodne, ale płodne hybrydy mogą skutkować specjacją .
- hybrydyzacja
- 1. Proces, w którym powstaje organizm hybrydowy z dwóch organizmów należących do różnych rodzajów, gatunków, ras lub odmian.
- 2. Proces, w którym dwie lub więcej jednoniciowych cząsteczek kwasu nukleinowego z komplementarne sekwencje nukleotydów łączą się ze sobą w roztworze, tworząc cząsteczki dwuniciowe lub trójniciowe poprzez tworzenie wiązań wodorowych między komplementarnymi zasadami nukleotydowymi każdej nici. W pewnych kontekstach laboratoryjnych, zwłaszcza tych, w których długie nici hybrydyzują z krótkimi starterami oligonukleotydowymi , hybrydyzacja jest często określana jako hybrydyzacja .
- 3. Etap w niektórych testach eksperymentalnych, w którym preparat jednoniciowego DNA lub RNA jest dodawany do powierzchni macierzy i przyłącza się do sondy komplementarnej .
- sonda hybrydyzacyjna
- hipoksantyna ( I )
- Naturalnie występująca niekanoniczna nukleozasada purynowa , która jest używana w niektórych cząsteczkach RNA i tworzy pary ze standardowymi nukleozasadami w zjawisku znanym jako parowanie wobble base . Jego nukleozydowa jest znana jako inozyna , co jest powodem, dla którego jest powszechnie skracana literą I w kolejnych odczytach.
I
- idiochromosom
- Zobacz allosome .
- idiogram
- Schematyczny lub schematyczny kariotyp całego zestawu chromosomów w komórce lub genomie, w którym ilustracje z adnotacjami są używane do przedstawienia każdego chromosomu w jego najbardziej wyidealizowanej formie (np. z liniami prostymi i oczywistymi centromerami ), aby ułatwić łatwą identyfikację sekwencji , cechy strukturalne i odległości fizyczne, które mogą być mniej widoczne na mikrofotografiach rzeczywistych chromosomów.
- idiomer
- Patrz chromomer .
- in silico
- (eksperymentu naukowego lub badań) Przeprowadzone, wyprodukowane lub przeanalizowane za pomocą modelowania komputerowego lub symulacji, w przeciwieństwie do próby w świecie rzeczywistym.
- in situ
- (eksperymentu naukowego lub procesu biologicznego) Występujący lub stworzony do wystąpienia w naturalnym, niekontrolowanym otoczeniu lub w naturalnym lub pierwotnym położeniu lub miejscu, w przeciwieństwie do obcej komórki lub tkanki lub w sztucznym środowisku .
- hybrydyzacja in situ
- in vitro
- (eksperymentu naukowego lub procesu biologicznego) Występujące lub stworzone do wystąpienia w naczyniu laboratoryjnym lub innym kontrolowanym sztucznym środowisku, np. w probówce lub szalce Petriego, w przeciwieństwie do wnętrza żywego organizmu lub środowiska naturalnego .
- in vivo
- (eksperymentu naukowego lub procesu biologicznego) Występujące lub stworzone do wystąpienia wewnątrz komórek lub tkanek żywego organizmu; lub, w najszerszym znaczeniu, w dowolnym naturalnym, niemanipulowanym otoczeniu. Kontrast ex vivo i in vitro .
- linia wsobna
- Dowolna linia określonego gatunku, w którym osobniki są prawie lub całkowicie identyczne genetycznie ze względu na długą historię powtarzającego się chowu wsobnego , w sposób naturalny lub sztuczny. Linie wsobne są zwykle uważane za wsobne po co najmniej 20 pokoleniach chowu wsobnego (np. przez samozapłodnienie lub kojarzenie rodzeństwa), w którym to momencie prawie wszystkie loci w całym genomie są homozygotyczne , a zatem wszystkie osobniki można skutecznie traktować jako klony (pomimo faktu, że osobniki są nadal produkowane w drodze rozmnażania płciowego).
- endogamia
- Rozmnażanie płciowe między rasami lub osobnikami blisko spokrewnionymi genetycznie . Chów wsobny skutkuje homozygotycznością , która może zwiększyć zarówno prawdopodobieństwo dotknięcia potomstwa szkodliwymi cechami recesywnymi , jak i prawdopodobieństwo utrwalenia korzystnych cech w populacji hodowlanej. Zdarzenie rozrodcze i wynikające z niego potomstwo mogą być określane zarówno jako krzyżówka , a potomstwo mówi się, że jest wsobne . Kontrastowe krzyżowanie . częstość
- występowania
- Częstość nowego występowania a choroba genetyczna (lub szerzej jakakolwiek choroba genetyczna lub cecha , szkodliwa lub nie) wśród członków określonej populacji iw określonym okresie czasu.
- Termin odnoszący się do insercji . lub delecji niekompletna jednej lub więcej zasad w sekwencji kwasu nukleinowego
- dominacja
- indel
- induktor
- Białko, które wiąże się z represorem (aby go wyłączyć) lub z aktywatorem (aby go włączyć).
- indukowalny gen
- A gen , którego ekspresja jest albo wrażliwa na zmiany środowiskowe, albo zależna od pozycji komórki gospodarza w cyklu komórkowym .
- dziedziczenie
- Zobacz dziedziczność .
- kodon inicjacji
- Zobacz kodon start .
- insercja
- inozyny
- Rodzaj mutacji , w której jedna lub więcej zasad jest dodawana do sekwencji kwasu nukleinowego . Usuwanie kontrastu .
- sekwencja wstawienia (IS)
- Dowolna sekwencja nukleotydowa wstawiona naturalnie lub sztucznie do innej sekwencji. Termin ten jest stosowany w szczególności w odniesieniu do części elementu zdolnego do transpozycji , która koduje te białka, które są bezpośrednio zaangażowane w proces transpozycji, np. enzym transpozazy . Region kodujący w transpozycyjnej sekwencji insercyjnej jest zwykle otoczony krótkimi odwróconymi powtórzeniami , a struktura większych elementów transpozycyjnych może obejmować parę flankujących sekwencji insercyjnych, które same są odwrócone.
- mutageneza insercyjna
- Zmiana sekwencji DNA poprzez wstawienie jednego lub więcej nukleotydów do sekwencji, naturalnie lub sztucznie. W zależności od dokładnej lokalizacji insercji w sekwencji docelowej, insercje mogą częściowo lub całkowicie inaktywować lub nawet regulować w górę produkt genu lub szlak biochemiczny, lub mogą być neutralne , nie prowadząc do żadnych istotnych zmian. Wiele inżynierii genetycznej polega na wstawianiu egzogennego materiału genetycznego do komórek gospodarza w celu zbadania funkcji i ekspresji genów.
- izolator
- Specyficzna sekwencja DNA, która zapobiega wpływowi na gen poprzez aktywację lub represję pobliskich genów.
- integron
- Ruchomy element genetyczny składający się z kasety genowej zawierającej gen dla rekombinazy specyficznej dla miejsca , miejsc rozpoznawania specyficznych dla integrazy oraz promotora , który reguluje ekspresję jednego lub więcej genów nadających komórce gospodarza cechy adaptacyjne. Integrony zwykle występują w postaci okrągłych episomów Fragmenty DNA, dzięki którym ułatwiają szybką adaptację bakterii, umożliwiając poziomy transfer genów oporności na antybiotyki między różnymi gatunkami bakterii.
- inteina
- Dowolna sekwencja jednego lub więcej aminokwasów w polipeptydzie prekursorowym , która jest wycinana przez składanie białka podczas modyfikacji potranslacyjnej i dlatego jest nieobecna w dojrzałym białku , analogicznie do intronów wyciętych z transkryptów RNA. Ekspozycja kontrastu .
- czynnik interkalujący
- Dowolny związek chemiczny (np. bromek etydyny ), który zaburza dopasowanie i parowanie zasad w komplementarnych niciach cząsteczki DNA poprzez wstawienie się między zasady .
- interkalacja
- Wstawienie, w sposób naturalny lub sztuczny, związków chemicznych między płaskie zasady cząsteczki DNA , które na ogół przerywa wiązanie wodorowe niezbędne do tworzenia par zasad .
- region intercistronowy
- Dowolna sekwencja DNA, która znajduje się między kodonem stop jednego genu a kodonem start następnego genu w policistronowej jednostce transkrypcyjnej. Zobacz także region międzygenowy .
- międzykrzyżowanie
- Krzyżówka , w której zarówno rodzice płci męskiej, jak i żeńskiej są heterozygotami w określonym locus .
- region międzygenowy (IGR)
- Dowolna sekwencja niekodującego DNA , która znajduje się pomiędzy funkcjonalnymi genami .
- odstępnik międzygenowy (IGS)
- Patrz odstępnik .
- interkineza
- Skrócona przerwa w czynnościach związanych z podziałem komórki, która występuje podczas mejozy u niektórych gatunków, pomiędzy pierwszym a drugim podziałem mejotycznym (tj. mejozą I i mejozą II). Żadna replikacja DNA nie zachodzi podczas interkinezy, w przeciwieństwie do normalnej interfazy , która poprzedza mejozę I i mitozę .
- wewnętrzne miejsce wejścia rybosomu (IRES)
- Sekwencja obecna w niektórych informacyjnych RNA , która umożliwia rozpoznanie przez rybosom , a tym samym rozpoczęcie translacji nawet przy braku czapeczki 5' , która w innym przypadku jest wymagana do złożenia kompleksu inicjacyjnego u eukariontów. Elementy IRES często znajdują się w nieulegającym translacji regionie 5' , ale można je również znaleźć w innych pozycjach.
- interfaza
- Wszystkie etapy cyklu komórkowego z wyłączeniem podziału komórki . Typowa komórka spędza większość swojego życia w interfazie, podczas której przeprowadza codzienne czynności metaboliczne, a także pełną replikację swojego genomu w ramach przygotowań do mitozy lub mejozy .
- region wewnątrzgenowy
- Zobacz intron .
- supresja wewnątrzgenowa
- introgresja
- konfliktu wewnątrzgenomowego
- Przeniesienie genu z puli genów jednej populacji lub gatunku do puli innej populacji poprzez wielokrotne krzyżowanie wsteczne mieszańców dwóch populacji z jedną z populacji rodzicielskich . Introgresja jest wszechobecnym i ważnym źródłem zmienności genetycznej w populacjach naturalnych, ale może być również praktykowana celowo w uprawie udomowionych roślin i zwierząt.
- intron
- Dowolna sekwencja nukleotydowa w funkcjonalnym genie , która jest usuwana przez splicing RNA podczas potranskrypcyjnej modyfikacji pierwotnego transkryptu mRNA i dlatego jest nieobecna w końcowym dojrzałym mRNA. Termin ten odnosi się zarówno do sekwencji istniejącej w cząsteczce DNA, jak i do odpowiedniej sekwencji w transkryptach RNA. Ekson kontrastowy .
- intruzja intronów
- rekombinacja za pośrednictwem intronów
- Zobacz tasowanie eksonów .
- powtórzenie odwrócone
- A sekwencja nukleotydowa , po której w dół na tej samej nici występuje jej własne odwrotne dopełnienie . Sekwencja początkowa i odwrotna sekwencja dopełniacza mogą być oddzielone dowolną liczbą nukleotydów lub mogą bezpośrednio przylegać do siebie; w tym drugim przypadku sekwencja złożona jest również nazywana sekwencją palindromową . Odwrócone powtórzenia są z definicji komplementarne , co jest właściwością, która obejmuje je w wielu biologicznych funkcjach i dysfunkcjach. Kontrastowe bezpośrednie powtórzenie .
- w kadrze
- 1. (genu lub sekwencji) Odczyt lub transkrypcja w tej samej ramce odczytu , co inny gen lub sekwencja; niewymagające przesunięcia ramki odczytu, aby były zrozumiałe lub dawały funkcjonalny peptyd .
- 2. (mutacji) Nie powoduje przesunięcia ramki odczytu .
- izochor
- Duży region genomowego DNA o względnie jednorodnym składzie par zasad , odróżniający się od innych regionów proporcją par G - C lub A - T . Genomy większości roślin i kręgowców składają się z różnych klas izochorów bogatych w GC i AT.
- izochromosom
- Typ nieprawidłowego chromosomu , w którym ramiona chromosomu są swoimi lustrzanymi odbiciami. Tworzenie izochromosomu jest równoważne jednoczesnym duplikacji i delecji , tak że dwie kopie albo długiego ramienia , albo krótkiego ramienia zawierają wynikowy chromosom.
- geny izomeryczne
- Dwa lub więcej genów które są równoważne i redundantne w tym sensie, że pomimo kodowania różnych produktów genowych , każdy z nich skutkuje tym samym fenotypem , gdy jest osadzony w tym samym tle genetycznym . Jeśli kilka genów izomerycznych jest obecnych w jednym genotypie , ich wkład w fenotyp może być kumulatywny lub niekumulacyjny.
J
- skaczący gen
- Zobacz element transpozycyjny .
- zróżnicowanie węzłowe
- śmieciowe DNA
- Dowolna sekwencja DNA, która wydaje się nie mieć żadnej znanej funkcji biologicznej lub która służy celowi, który nie ma pozytywnego lub netto negatywnego wpływu na dopasowanie genomu, w którym się znajduje . Termin ten był kiedyś szerzej używany w odniesieniu do całego niekodującego DNA , z którego wiele, jak później odkryto, pełniło jakąś funkcję; we współczesnym użyciu zazwyczaj odnosi się do uszkodzonych lub szczątkowych sekwencji i samolubnych elementów genetycznych , w tym intronów , pseudogeny , międzygenowe DNA oraz fragmenty transpozonów i retrowirusów , które razem stanowią dużą część genomów większości eukariontów. Pomimo tego, że nie przyczyniają się produktywnie do organizmu gospodarza, sekwencje te są w stanie przetrwać w genomach w nieskończoność, ponieważ wady dalszego ich kopiowania są zbyt małe, aby można było na nie oddziaływać doborem naturalnym .
- śmieciowe RNA
- Dowolna sekwencja zakodowana w RNA, zwłaszcza transkrypt , który wydaje się nie mieć żadnej znanej funkcji biologicznej lub którego funkcja nie ma pozytywnego lub negatywnego wpływu netto na dopasowanie genomu, z którego jest transkrybowana. Pomimo tego, że nie ulegają translacji , wiele niekodujących RNA nadal pełni ważne funkcje, podczas gdy śmieciowe RNA są naprawdę bezużyteczne: często są produktem przypadkowej transkrypcji sekwencji śmieciowego DNA lub mogą wynikać z przetwarzania potranskrypcyjnego pierwotnych transkryptów , jak w przypadku wycięte introny . Śmieciowe RNA jest zwykle szybko rozkładane przez rybonukleazy i inne enzymy cytoplazmatyczne.
k
- Kariotyp
- stosunku K a / K s
- Liczba i wygląd chromosomów w jądrze komórki eukariotycznej, zwłaszcza przedstawiony na zorganizowanej mikrofotografii zwanej kariogramem lub idiogramem (w parach i uporządkowane według wielkości i położenia centromeru ) . Termin ten jest również używany w odniesieniu do pełnego zestawu chromosomów w gatunku lub pojedynczym organizmie lub do dowolnego testu, który wykrywa ten dopełniacz lub mierzy liczbę chromosomów.
- kilozasada (kb)
- Jednostka długości kwasu nukleinowego równa 1000 zasad w cząsteczkach jednoniciowych lub 1000 par zasad w cząsteczkach dupleksowych, takich jak dwuniciowy DNA .
- kinetochor
- Kompleks białkowy w kształcie dysku, który gromadzi się wokół centromeru chromosomu podczas prometafazy mitozy i mejozy , gdzie działa jako punkt przyczepu dla mikrotubul aparatu wrzeciona .
- pokrętło
- W cytogenetyce powiększony, silnie barwiący się chromomer , który może być używany jako wizualny znacznik, umożliwiający łatwą identyfikację określonych chromosomów w jądrze.
- knockdown
- Technika inżynierii genetycznej , dzięki której normalna szybkość ekspresji jednego lub więcej genów organizmu zostaje zmniejszona, albo poprzez bezpośrednią modyfikację sekwencji DNA, albo poprzez traktowanie odczynnikiem, takim jak krótki oligonukleotyd DNA lub RNA o sekwencji komplementarny do transkryptu mRNA lub genu.
- knockin
- knockout
- Technika inżynierii genetycznej , w której organizm jest modyfikowany tak, aby zawierał geny , które zostały wyłączone („znokautowane”), tak że ich ekspresja zostaje zakłócona w pewnym momencie szlaku, który wytwarza produkty ich genów , a organizm jest pozbawiony ich normalne skutki. Kontrastowe pukanie .
- Sekwencja konsensusu Kozaka
- Wysoce konserwatywny motyw sekwencji kwasu nukleinowego , który działa jako miejsce rozpoznawania inicjacji translacji w większości eukariotycznych informacyjnych RNA , generalnie sekwencja 10 zasad bezpośrednio otaczająca i obejmująca kodon start : GCC R CCAUGG . Gdy kompleks przedinicjacyjny skanuje transkrypt, rozpoznanie tej sekwencji (lub bliskiego wariantu) powoduje, że kompleks przypisuje się do pełnego rybosomu montaż i rozpoczęcie tłumaczenia. Sekwencja Kozaka różni się od innych rozpoznawanych sekwencji istotnych dla translacji, takich jak miejsca wiązania rybosomu i wewnętrzne miejsca wejścia rybosomu .
Ł
- etykietowanie
- Technika laboratoryjna polegająca na chemicznym przyłączaniu wysoce selektywnej substancji, znanej jako znacznik , znacznik lub sonda , do określonej komórki, białka, aminokwasu lub innej interesującej nas biomolekuły, in vivo lub in vitro . Znacznik jest typowo reaktywną pochodną naturalnie fluorescencyjnego związku (np. zielonego białka fluorescencyjnego ) lub jakąkolwiek inną substancję, która w jakiś sposób sprawia, że jej cel jest rozpoznawalny; inne powszechnie stosowane etykiety obejmują barwniki, enzymy, przeciwciała i cząsteczki radioaktywne. Oznaczane cele są w ten sposób odróżniane od otoczenia, co pozwala na ich łatwe wykrycie, identyfikację, oznaczenie ilościowe lub wyizolowanie do dalszych badań.
- nić opóźniona
- W replikacji DNA powstająca nić , dla której kierunek syntezy polimerazy DNA jest odwrócony od widełek replikacyjnych , co wymaga złożonego i nieciągłego procesu, w przeciwieństwie do usprawnionej, ciągłej syntezy drugiej powstającej nici , która zachodzi jednocześnie. Ponieważ polimeraza DNA działa tylko w od 5' do 3' , ale ogólny kierunek wydłużania łańcucha musi ostatecznie być przeciwny (tj. od 3' do 5' w kierunku widełek replikacyjnych), wydłużenie musi zachodzić poprzez mechanizm pośredni, w prymasa syntetyzuje krótkie startery RNA komplementarny do matrycy DNA, a następnie polimeraza DNA wydłuża zapoczątkowane segmenty do krótkich łańcuchów nukleotydów znanych jako fragmenty Okazaki . Startery RNA są następnie usuwane i zastępowane DNA, a fragmenty Okazaki są łączone przez ligazę DNA . boczny transfer genów
- w chromosomie szczotki lampowej
- (LGT)
- Zobacz poziomy transfer genów .
- Prawo dominacji i jednolitości
- Jedna z trzech fundamentalnych zasad dziedziczenia mendlowskiego , która mówi, że różne allele tego samego genu może być dominujący lub recesywny w stosunku do innych, oraz że organizm z co najmniej jednym dominującym allelem będzie jednolicie wykazywał fenotyp związany z dominującym allelem.
- Prawo niezależnego doboru
- Jedna z trzech fundamentalnych zasad dziedziczenia mendlowskiego , która mówi, że geny odpowiedzialne za różne cechy fenotypowe są niezależnie segregowane podczas mejozy . Geny połączone są godnym uwagi wyjątkiem od tej reguły.
- Prawo segregacji
- Jedna z trzech podstawowych zasad Dziedziczenie mendlowskie , które stwierdza , że podczas mejozy allele każdego genu oddzielają się od siebie w taki sposób, że każda wynikowa gameta niesie tylko jeden allel każdego genu.
- sekwencja liderowa
- Patrz region nieulegający translacji 5' .
- nić wiodąca
- W replikacji DNA powstająca nić , dla której zarówno kierunek syntezy polimerazy DNA , jak i ogólne wydłużenie łańcucha są skierowane w stronę widełek replikacyjnych , tj. oba zachodzą w kierunku od 5' do 3' , co skutkuje pojedynczym, ciągłym procesem wydłużania z niewielką liczbą przerw lub bez przerw. Natomiast druga powstająca nić, znana jako nić opóźniona , jest składana w nieciągłym procesie obejmującym ligację krótkich fragmentów DNA , które są syntetyzowane w przeciwnym kierunku, z dala od widełek replikacyjnych.
- lewe złącze splicingowe
- Granica między lewym końcem (zgodnie z konwencją końcem 5' ) intronu a prawym ( 3' ) końcem sąsiedniego eksonu w transkrypcie pre -mRNA .
- leptonemia
- W mejozie pierwszy z pięciu podetapów profazy I , następujący po interfazie i poprzedzający zygonemę . Podczas leptonemy replikowane chromosomy kondensują się z rozproszonej chromatyny w długie, cienkie pasma, które są znacznie bardziej widoczne w jądrze .
- letalna wartość równoważna
- Średnia liczba recesywnych szkodliwych genów istniejących w stanie heterozygotycznym , które są przenoszone przez członka populacji diploidalnej organizmów, pomnożona przez średnie prawdopodobieństwo, że każdy taki gen spowoduje przedwczesną śmiertelność , gdy jest homozygotą . Na przykład, mówi się, że organizm niosący osiem recesywnych półśmiercionośnych alleli, z których każdy daje tylko 50% prawdopodobieństwo przedwczesnej śmierci, gdy jest homozygotą, ma genetyczne obciążenie czterech „śmiertelnych równoważników”.
- mutacja letalna
- Każda mutacja powodująca przedwczesną śmierć organizmu, który ją nosi. Recesywne mutacje letalne są śmiertelne tylko dla homozygot dominujące letale są śmiertelne nawet u heterozygot .
- ligaza
- suwaka leucynowego
- Klasa enzymów, które katalizują łączenie dużych cząsteczek, takich jak kwasy nukleinowe , poprzez tworzenie między nimi jednego lub więcej wiązań chemicznych, zwykle wiązań C–C , C–O , C–S lub C–N w reakcjach kondensacji . Przykładem jest ligaza DNA , która katalizuje tworzenie wiązań fosfodiestrowych między sąsiednimi nukleotydami na jednej lub obu niciach cząsteczki DNA, reakcja znana jako ligacja .
- rodowód
- ligacyjny
- Liniowa sekwencja ewolucyjna łącząca przodkową komórkę, organizm lub gatunek z określoną komórką, organizmem lub gatunkiem potomnym, w tym wszystkie organizmy pośrednie i obejmująca dowolną liczbę pokoleń ; bezpośrednia progresja zdarzeń reprodukcyjnych (tj. linia pochodzenia) między dwoma osobnikami, w tym osobniki spokrewnione pionowo, np. rodzic (rodzice) i potomstwo, ale zwykle z wyłączeniem osobników spokrewnionych poziomo, które same nie wniosły bezpośrednio materiału genetycznego do żadnego z uwzględnionych osobników np. rodzeństwo.
- połączenie
- Tendencja sekwencji DNA, które są fizycznie blisko siebie na tym samym chromosomie, do wspólnego dziedziczenia podczas mejozy . Ponieważ fizyczna odległość między nimi jest stosunkowo niewielka, prawdopodobieństwo, że dowolne dwie pobliskie części sekwencji DNA (często loci lub markery genetyczne ) zostaną rozdzielone na różne chromatydy podczas krzyżowania chromosomów , jest statystycznie bardzo niskie; mówi się wtedy, że takie loci są bardziej połączone niż loci, które są dalej od siebie. Mówi się, że loci, które istnieją na zupełnie różnych chromosomach, są doskonale niepołączone . Standardową jednostką pomiaru powiązań genetycznych jest centymorgan (cM).
- linker nierównowaga sprzężeń
- DNA
- 1. Krótki, syntetyczny dupleks DNA zawierający sekwencję rozpoznawaną dla określonego enzymu restrykcyjnego . W klonowaniu molekularnym łączniki są często celowo włączane do rekombinowanych cząsteczek, aby umożliwić ich łatwą modyfikację poprzez umożliwienie rozszczepienia i wstawienia obcych sekwencji w ściśle określonych lokalizacjach. Segment zmodyfikowanego plazmidu zawierający wiele takich miejsc restrykcyjnych jest czasem nazywany polilinkerem .
- 2. Fragment chromosomalnego DNA łączący sąsiednie nukleosomy poprzez wiązanie z histonem H1 .
- liczba łącząca
- Liczba przypadków, w których dwie nici kolistej podwójnej helisy DNA krzyżują się ze sobą, co odpowiada liczbie skręcenia (która mierzy skręcenie podwójnej helisy) plus liczba wicia (który mierzy stopień superskręcenia). Numeru łączącego zamkniętą cząsteczkę nie można zmienić bez rozerwania i ponownego połączenia nici. Cząsteczki DNA, które są identyczne, z wyjątkiem ich liczb łączących, są znane jako izomery topologiczne .
- lncRNA
- Patrz długi niekodujący RNA .
- umiejscowienie
- Specyficzna, ustalona pozycja na chromosomie , w której znajduje się określony gen lub marker genetyczny .
- Wynik LOD
- długie ramię
- W skondensowanych chromosomach , w których położenie centromeru tworzy dwa segmenty lub „ramiona” o różnej długości, dłuższe z dwóch ramion chromatydy . Kontrastowe krótkie ramię .
- long interspered jądrowy element (LINE)
- Każdy z dużej rodziny retrotranspozonów innych niż LTR , które razem składają się na jeden z najbardziej rozpowszechnionych ruchomych elementów genetycznych w genomach eukariotycznych. Każda wstawka LINE ma średnio około 7000 par zasad.
- długi niekodujący RNA (lncRNA)
- Klasa niekodującego RNA składająca się ze wszystkich transkryptów o długości ponad 200 nukleotydów , które nie ulegają translacji . Ta granica odróżnia lncRNA od wielu mniejszych niekodujących RNA, takich jak mikroRNA . Zobacz także długi interweniujący niekodujący RNA .
- lionizacja
- Patrz inaktywacja X.
Zobacz też
- Wprowadzenie do genetyki
- Zarys genetyki
- Zarys biologii komórki
- Słowniczek biologii
- Słowniczek chemii
- Słowniczek biologii ewolucyjnej
- ^ „Mówiący glosariusz terminów genetycznych” . genom.gov. 8 października 2017 . Źródło 8 października 2017 r .
- ^ ab . Ussery, David W. „Struktura DNA: rodziny helis A-, B- i Z-DNA” Lyngby, Dania: Duński Uniwersytet Techniczny.
- ^ abcde Klug , William S .; Cummings, Michael R. (1986). Koncepcje genetyki (wyd. 2). Glenview, Illinois: Scott, Foresman and Company. ISBN 0-673-18680-6 .
- ^ A b c d e f g hi j k l m n o p q r s t u v w x y z aa ab ac ad ae af ag ai aj King , Robert C.; Stansfield, William D.; Mulligan, Pamela K. (2006). A Dictionary of Genetics (wyd. 7). Oksford: Oxford University Press. ISBN 978-0-19-530762-7 .
- ^ „Słownik terminów genetycznych NCI” . Narodowy Instytut Raka . Redakcja PDQ® Cancer Genetics.
- ^ a b Lewin, Benjamin (2003). Geny VIII . Upper Saddle River, NJ: Pearson Prentice Hall. ISBN 0-13-143981-2 .
- ^ Nishikawa S. (2007). „Przeprogramowanie według numerów”. Biotechnologia przyrody . 25 (8): 877–878. doi : 10.1038/nbt0807-877 . PMID 17687365 . S2CID 39773318 .
- ^ a b c d e f g hi . Rieger, Rigomar (1991) Glosariusz genetyki: klasyczny i molekularny (wyd. 5). Berlin: Springer-Verlag. ISBN 3540520546 .
- ^ Księżniczka, I .; Maimon, O.; Ben-Gal, I. (2007). „Ocena grupowania ekspresji genów za pomocą wzajemnej miary odległości informacji” . BMC Bioinformatyka . 8 : 111. doi : 10.1186/1471-2105-8-111 . PMC 1858704 . PMID 17397530 .
- ^ „Społeczeństwo danych genomiki funkcjonalnej - społeczeństwo FGED” .
- Bibliografia _ Domraczew, M; Lash, AE (1 stycznia 2002). „Gene Expression Omnibus: ekspresja genów NCBI i repozytorium danych macierzy hybrydyzacji” . Badania kwasów nukleinowych . 30 (1): 207–10. doi : 10.1093/nar/30.1.207 . PMC 99122 . PMID 11752295 .
- Bibliografia _ Wilhite, SE; Ledoux, P; Ewangelista, C; Kim, JEŚLI; Tomaszewski, M; Marshall, KA; Phillippy, KH; Sherman, PM; Holko, M; Jefanow, A; Lee, H.; Zhang, N; Robertson, CL; Serowa, N; Davis, S; Sobolewa, A (styczeń 2013). „NCBI GEO: archiwum zestawów danych genomiki funkcjonalnej - aktualizacja” . Badania kwasów nukleinowych . 41 (problem z bazą danych): D991-5. doi : 10.1093/nar/gks1193 . PMC 3531084 . PMID 23193258 .
- ^ Resta R, Biesecker BB, Bennett RL, Blum S, Hahn SE, Strecker MN, Williams JL (kwiecień 2006). „Nowa definicja poradnictwa genetycznego: raport grupy zadaniowej National Society of Genetic Counselors” . Dziennik poradnictwa genetycznego . 15 (2): 77–83. doi : 10.1007/s10897-005-9014-3 . PMID 16761103 .
- ^ Yakovchuk P, Protozanova E, Frank-Kameneckii MD (2006). „Wkład zasad w układanie w stosy i parowanie zasad w stabilność termiczną podwójnej helisy DNA” . Kwasy nukleinowe Res . 34 (2): 564–74. doi : 10.1093/nar/gkj454 . PMC 1360284 . PMID 16449200 .
- ^ Kozak, M. (luty 1989). „Model skanowania do tłumaczenia: aktualizacja” . Journal of Cell Biology . 108 (2): 229–241. doi : 10.1083/jcb.108.2.229 . ISSN 0021-9525 . PMC 2115416 . PMID 2645293 .
Dalsza lektura
- Budd, A. (2012). „Wprowadzenie do biologii genomu: cechy, procesy i struktury”. Genomika ewolucyjna . Metody w biologii molekularnej . Tom. 855. s. 3–4. doi : 10.1007/978-1-61779-582-4_1 . ISBN 978-1-61779-581-7 . PMID 22407704 .

