Słowniczek genetyki (M – Z)
Ten słowniczek genetyki jest listą definicji terminów i pojęć powszechnie używanych w badaniu genetyki i pokrewnych dziedzin biologii , w tym biologii molekularnej , biologii komórki i biologii ewolucyjnej . Jest przeznaczony jako materiał wprowadzający dla nowicjuszy; bardziej szczegółowe i techniczne szczegóły można znaleźć w artykule odpowiadającym każdemu terminowi. Aby zapoznać się z pokrewnymi terminami, zobacz Glosariusz biologii ewolucyjnej .
Ten glosariusz jest podzielony na dwa artykuły:
- Glosariusz genetyki (0–L) wymienia terminy zaczynające się od cyfr i te zaczynające się na litery od A do L.
- Glosariusz genetyki (M – Z) (ta strona) zawiera terminy zaczynające się na litery od M do Z.
M
- głównego rowka
- (mu)
- Patrz centymorgan .
- klonowanie oparte na mapie
- Zobacz klonowanie pozycyjne .
- matrokliniczny
- (potomstwa ) Pod względem genotypowym lub fenotypowym bardziej przypomina rodzica płci żeńskiej niż rodzic płci męskiej ; pochodzi od matki. Kontrastowy patrokliniczny .
- genetyka medyczna
- dziedzina medycyny i nauk medycznych zajmująca się badaniem, diagnozowaniem i leczeniem zaburzeń dziedzicznych oraz szerzej stosowaniem wiedzy o genetyce człowieka do opieki medycznej.
- mejoza
- Wyspecjalizowany typ podziału komórkowego , który występuje wyłącznie u eukariotów rozmnażających się płciowo , podczas którego po replikacji DNA następują dwie kolejne rundy podziału, w wyniku których ostatecznie powstają cztery genetycznie unikalne haploidalne komórki potomne, z których każda ma połowę liczby chromosomów w porównaniu z pierwotną diploidalną komórką macierzystą . Mejoza występuje tylko w komórkach narządów płciowych i służy do generowania haploidalnych gamet , takich jak plemniki , jaja lub zarodniki , które później łączą się podczas zapłodnienia . Dwa podziały mejotyczne, znane jako mejoza I i mejoza II , mogą również obejmować różne zdarzenia rekombinacji genetycznej między chromosomami homologicznymi .
- wrzeciono mejotyczne
- Zobacz aparat wrzeciona .
- topienie
- Denaturacja dwuniciowego kwasu nukleinowego na dwie pojedyncze nici , zwłaszcza w kontekście reakcja łańcuchowa polimerazy .
- Dziedziczenie mendlowskie
- Teoria dziedziczenia biologicznego oparta na zbiorze zasad pierwotnie zaproponowanych przez Gregora Mendla w 1865 i 1866 roku. Mendel wyprowadził trzy ogólne prawa dotyczące genetycznych podstaw dziedziczenia, które wraz z kilkoma teoriami opracowanymi przez późniejszych naukowców są uważane za podstawę genetyka klasyczna . Porównaj dziedziczenie niemendlowskie .
- informacyjny RNA (mRNA)
- Dowolny z klasy jednoniciowego RNA cząsteczki, które działają jako przekaźniki molekularne, przenosząc informacje o sekwencji zakodowane w genomie DNA do rybosomów , w których zachodzi synteza białek. Podstawowe produkty transkrypcji , mRNA, są syntetyzowane przez polimerazę RNA , która buduje łańcuch rybonukleotydów , które uzupełniają dezoksyrybonukleotydy matrycy DNA ; w ten sposób sekwencja DNA genu kodującego białko jest skutecznie zachowana w surowym transkrypcie , który jest następnie przetwarzany do dojrzałego mRNA przez serię modyfikacji potranskrypcyjnych .
- metabolom
- metacentryczny
- (liniowego chromosomu lub fragmentu chromosomu) Posiadający centromer umieszczony w środku chromosomu, w wyniku czego ramiona chromatydowe mają w przybliżeniu równą długość.
- metagenomika
- Badanie materiału genetycznego pozyskanego bezpośrednio z próbek środowiskowych, w przeciwieństwie do organizmów hodowanych w hodowlach laboratoryjnych .
- metafaza
- Etap mitozy i mejozy , który następuje po prometafazie i przed anafazą , podczas którego centromery replikowanych chromosomów ustawiają się wzdłuż równika komórki, a każdy kinetochor jest przyczepiony do wrzeciona mitotycznego .
- Mikromacierze i ekspresja genów (MAGE)
- Grupa, która „ma na celu zapewnienie standardu reprezentacji danych dotyczących ekspresji genów z mikromacierzy DNA , który ułatwiłby wymianę informacji o mikromacierzach między różnymi systemami danych”.
- mikrochromosom
- Typ bardzo małego chromosomu , na ogół o wielkości mniejszej niż 20 000 par zasad , obecny w kariotypach niektórych organizmów.
- mikrodelecja
- Delecja chromosomalna _ jest zbyt krótki, aby spowodować jakąkolwiek widoczną zmianę morfologii pod mikroskopem świetlnym, chociaż nadal może być wykrywalny innymi metodami, takimi jak sekwencjonowanie .
- mikroRNA (miRNA)
- Rodzaj małej, jednoniciowej , niekodującej cząsteczki RNA, która działa w potranskrypcyjnej regulacji ekspresji genów , w szczególności wyciszaniu RNA , poprzez parowanie zasad z komplementarnymi sekwencjami w transkryptach mRNA , co zwykle skutkuje rozszczepieniem lub destabilizacją transkryptu lub hamuje jego translację przez rybosomy.
- mikrosatelita
- Typ satelitarnego DNA składający się ze stosunkowo krótkiej sekwencji powtórzeń tandemowych , w których pewne motywy (o długości od jednej do sześciu lub więcej zasad ) są powtarzane, zazwyczaj 5–50 razy. Mikrosatelity są szeroko rozpowszechnione w genomach większości organizmów i mają zwykle wyższe wskaźniki mutacji niż inne regiony. Są one klasyfikowane jako o zmiennej liczbie powtórzeń tandemowych (VNTR), wraz z dłuższymi minisatelitami .
- midparent value
- Średnia dwóch wartości rodzicielskich dla a cecha ilościowa u pojedynczego potomstwa lub w określonej krzyżówce .
- Minimum informacji o eksperymencie mikromacierzy (MIAME)
- Standard komercyjny opracowany przez FGED i oparty na MAGE w celu ułatwienia przechowywania i udostępniania danych dotyczących ekspresji genów .
- Minimalna informacja o wysokowydajnym eksperymencie sekwencjonowania (MINSEQE)
- Komercyjny standard opracowany przez FGED do przechowywania i udostępniania wysokoprzepustowego sekwencjonowania dane.
- częstotliwość mniejszego allelu (MAF)
- minisatelity
- mniejsza nić
- minus nić
- Zobacz szablon nici .
- miRNA
- Patrz mikroRNA .
- naprawa niezgodności (MMR)
- niezgodność
- Nieprawidłowe sparowanie zasad nukleinowych na komplementarnych niciach DNA lub RNA , tj. obecność w jednej nici cząsteczki dupleksowej zasady, która nie jest komplementarna (zgodnie z regułami parowania Watsona-Cricka) do zasady zajmującej odpowiednią pozycję w drugiej nici , co zapobiega normalnym wiązaniom wodorowym między zasadami; np. guanina dopasowana do tyminy .
- mutacja zmiany sensu
- Rodzaj mutacji punktowej , w wyniku której a kodon , który koduje inny aminokwas niż w niezmutowanej sekwencji. Porównaj mutację nonsensowną .
- mistranslation
- Wstawienie nieprawidłowego aminokwasu do rosnącego łańcucha peptydowego podczas translacji , tj. włączenie dowolnego aminokwasu, który nie jest określony przez określony kodon w transkrypcie mRNA . Błędna translacja może pochodzić z źle naładowanego transferu RNA lub z nieprawidłowo działającego rybosomu .
- mitoza
- mitochondrialnego DNA (mtDNA)
- W komórkach eukariotycznych część cyklu komórkowego, podczas której następuje podział jądra i replikowane chromosomy są rozdzielone na dwa odrębne jądra. Mitoza jest na ogół poprzedzona etapem S interfazy , kiedy DNA komórki jest replikowane , i albo występuje jednocześnie z cytokinezą, albo następuje po niej cytokineza , kiedy cytoplazma i błona komórkowa dzieli się na dwie nowe komórki potomne.
- rekombinacja mitotyczna
- segregacja mitotyczna
- wrzeciono mitotyczne
- Zobacz aparat wrzeciona .
- mixoploidia
- Obecność więcej niż jednego poziomu ploidalności , tj. więcej niż jednej liczby zestawów chromosomów , w różnych komórkach tej samej populacji komórkowej.
- ruchomy element genetyczny (MGE)
- Każdy materiał genetyczny, który może przemieszczać się między różnymi częściami genomu lub być przenoszony z jednego gatunku lub replikonu do innego w ciągu jednego pokolenia . Wiele typów MGE obejmuje elementy transpozycyjne , plazmidy bakteryjne , elementy bakteriofagowe , które integrują się z genomami gospodarza przez transdukcję wirusową oraz introny samosplicingowe .
- mobilom
- Cały zestaw ruchomych elementów genetycznych w obrębie określonego genomu , komórki, gatunku lub innego taksonu, w tym wszystkie transpozony , plazmidy , profagi i inne samoskładające się cząsteczki kwasu nukleinowego.
- klonowanie molekularne
- Dowolna z różnych metod biologii molekularnej zaprojektowana w celu wielokrotnej replikacji określonej cząsteczki, zwykle sekwencji DNA, wewnątrz komórek naturalnego gospodarza. Zwykle zrekombinowany fragment DNA zawierający interesujący gen jest ligowany z wektorem plazmidowym , który jest następnie przekształcany w kompetentne komórki bakteryjne. Bakteriom niosącym zrekombinowany plazmid pozwala się następnie naturalnie namnażać na pożywce hodowlanej, gdzie plazmidy są replikowane wraz z resztą genomu bakteryjnego; każdy działający gen będący przedmiotem zainteresowania będzie eksprymowany przez komórki bakteryjne, a zatem jego produkty genowe również zostaną sklonowane. Klonowanie molekularne jest podstawowym narzędziem inżynierii genetycznej , przeznaczonym do badania ekspresji genów, amplifikacji określonego produktu genu i generowania selekcyjnego fenotypu .
- genetyka molekularna
- Dział genetyki wykorzystujący metody i techniki biologii molekularnej do badania struktury i funkcji genów oraz produktów genów na poziomie molekularnym . Porównaj klasyczną genetykę .
- monada
- Haploidalny zestaw chromosomów występujący w jądrze niedojrzałej komórki gamet , takiej jak ootyda lub plemnik , tj. komórka, która zakończyła mejozę , ale nie jest jeszcze dojrzałą gametą.
- monocentryczny (z
- chromosomu liniowego lub fragmentu chromosomu) Posiadający tylko jeden centromer . Kontrast dicentryczny i holocentryczny .
- monosomia
- monoploidalna
- Nieprawidłowa i często patologiczna obecność tylko jednego chromosomu normalnej pary diploidalnej. Jest to rodzaj aneuploidii .
- Morfolino
- Syntetyczna polimeryczna cząsteczka łącząca krótkie sekwencje zasad nukleinowych w sztuczne antysensowne oligomery , które są wykorzystywane w inżynierii genetycznej do obniżania ekspresji genów poprzez parowanie z komplementarnymi sekwencjami w naturalnie występujących cząsteczkach RNA lub DNA, zwłaszcza transkryptach mRNA , blokując w ten sposób dostęp innym biomolekułom, takim jak rybosomy i białka. Oligomery morfolinowe same nie ulegają translacji i ani one, ani ich hybrydowe dupleksy z RNA nie są atakowane przez nukleazy ; ponadto, w przeciwieństwie do ujemnie naładowanych fosforanów normalnych kwasów nukleinowych, syntetyczne szkielety Morpholinos są elektrycznie obojętne, co zmniejsza prawdopodobieństwo ich nieselektywnej interakcji z naładowanymi białkami komórki gospodarza. Te właściwości czynią je użytecznymi i niezawodnymi narzędziami do sztucznego generowania zmutowanych fenotypów w żywych komórkach.
- mozaicyzm
- Obecność dwóch lub więcej populacji komórek o różnych genotypach w pojedynczym organizmie, który rozwinął się z pojedynczego zapłodnionego jaja . Organizm mozaikowy może wynikać z wielu rodzajów zjawisk genetycznych, w tym niedysjunkcji chromosomów, endoreduplikacji lub mutacji w poszczególnych liniach komórek macierzystych podczas wczesnego rozwoju zarodka. Mozaicyzm jest podobny do chimeryzmu , ale różni się od niego .
- motyw
- Dowolna wyróżniająca się lub powtarzająca się sekwencja nukleotydów w kwasie nukleinowym lub aminokwasów w a białko , które jest lub co do którego przypuszcza się, że ma znaczenie biologiczne, zwłaszcza takie, które jest wiarygodnie rozpoznawane przez inne biomolekuły lub które ma trójwymiarową strukturę , która umożliwia unikalne lub charakterystyczne interakcje chemiczne, takie jak wiązanie DNA . W kwasach nukleinowych motywy są często krótkie (długość od trzech do dziesięciu nukleotydów), wysoce konserwatywne sekwencje , które działają jako miejsca rozpoznawania dla enzymów wiążących DNA lub RNA zaangażowanych w regulację ekspresji genów .
- mRNA
- Patrz informacyjny RNA .
- mtDNA
- Patrz DNA mitochondrialne .
- Multiomics
- miejsce wielokrotnego klonowania (MCS)
- mutagen
- Każdy czynnik fizyczny lub chemiczny, który zmienia materiał genetyczny (zwykle DNA ) organizmu, a tym samym zwiększa częstotliwość mutacji powyżej naturalnego poziomu tła.
- mutageneza
- 1. Proces, w wyniku którego zmienia się informacja genetyczna organizmu, prowadząc do mutacji . Mutageneza może wystąpić samoistnie lub w wyniku ekspozycji na mutagen .
- 2. W biologii molekularnej każda technika laboratoryjna, za pomocą której celowo dokonuje się jednej lub więcej mutacji genetycznych zaprojektowane w celu wytworzenia zmutowanego genu, elementu regulatorowego, produktu genu lub organizmu zmodyfikowanego genetycznie, tak aby można było szczegółowo zbadać funkcje locus genetycznego, procesu lub produktu.
- mutant
- Organizm, produkt genu lub cecha fenotypowa wynikająca z mutacji typu, którego nie można zaobserwować naturalnie w okazach typu dzikiego .
- mutacja
- Każda trwała zmiana w sekwencji nukleotydowej nici DNA lub RNA lub w sekwencji aminokwasowej peptydu . Mutacje odgrywają rolę zarówno w normalnych, jak i nieprawidłowych procesach biologicznych; ich naturalne występowanie jest integralną częścią procesu ewolucji . Mogą one wynikać z błędów replikacji , uszkodzeń chemicznych, narażenia na promieniowanie wysokoenergetyczne lub manipulacji ruchomymi elementami genetycznymi . Mechanizmy naprawcze ewoluowały w wielu organizmach, aby je naprawić. Dzięki zrozumieniu wpływu mutacji na fenotyp możliwe jest ustalenie funkcji genu lub kolejności, w jakiej występuje.
- odległość mutacji
- Najmniejsza liczba mutacji wymaganych do wyprowadzenia jednego genu, sekwencji lub fenotypu z innego; minimalna liczba insercji, delecji lub substytucji nukleozasad niezbędnych do zmiany jednej sekwencji na inną.
- zdarzenie mutacji
- Faktyczne pochodzenie określonej mutacji w czasie i przestrzeni; przypadek jego pierwotnego wprowadzenia do genomu, w przeciwieństwie do jego manifestacji fenotypowej, która może nastąpić dopiero pokolenia po fakcie.
- wskaźnik mutacji
- Częstotliwość nowych mutacji w określonym locus lub w określonym genie , sekwencji , genomie lub organizmie w określonym czasie, np. podczas jednego pokolenia . Wskaźniki mutacji można obliczyć dla określonej klasy mutacji lub dla wszystkich typów zbiorczo; różnią się znacznie w zależności od organizmu i środowiska organizmu.
- obciążenie mutacyjne
- gorących punktów
- Patrz obciążenie genetyczne .
- gen mutatora
- Dowolny zmutowany gen lub sekwencja, która zwiększa szybkość spontanicznych mutacji jednego lub większej liczby innych genów lub sekwencji. Mutatory są często elementami podlegającymi transpozycji lub mogą być zmutowanymi genami porządkującymi , takimi jak te, które kodują helikazy lub białka zaangażowane w korektę .
- muteina
- Zmutowane białko , tj. białko, którego sekwencja aminokwasowa różni się od prawidłowej z powodu mutacji .
- baranina
- Najmniejsza jednostka cząsteczki DNA, w której zmiana fizyczna lub chemiczna może spowodować mutację ( zwykle pojedynczy nukleotyd ).

Struktura typowego informacyjnego RNA lub mRNA kodującego dojrzałe białko , narysowana w przybliżeniu w skali. Sekwencja kodująca (zielona) jest ograniczona nieulegającymi translacji regionami zarówno na końcu 5' (żółty), jak i na końcu 3' (różowy). Przed eksportem z jądra dodaje się czapeczkę 5' (czerwoną) i ogon poli(A) 3' (czarny), aby pomóc ustabilizować mRNA i zapobiec jego degradacji przez rybonukleazy.

Zarys działania klonowania molekularnego
N
- n orientacja
- Jedna z dwóch możliwych orientacji, w których liniowy fragment DNA może zostać wstawiony do wektora , w szczególności ta, w której mapy genów fragmentu i wektora mają tę samą orientację. Kontrast u orientacji .
- N-koniec
- Koniec liniowego łańcucha aminokwasów (tj. peptyd ), który jest zakończony wolną grupą aminową ( -NH
2 ) pierwszego aminokwasu dodanego do łańcucha podczas translacji . Mówi się, że ten aminokwas jest N-końcowy . Zgodnie z konwencją, sekwencje, domeny, miejsca aktywne lub dowolna inna struktura znajdująca się bliżej N-końca polipeptydu lub jego postaci sfałdowanego białka w stosunku do innych są opisywane jako powyżej . Kontrast C-koniec . - NAD
- Zobacz dinukleotyd nikotynamidoadeninowy .
- NADP
- Patrz fosforan dinukleotydu nikotynamidoadeninowego .
- rodzący się
- W trakcie syntezy; niekompletny; jeszcze nie w pełni przetworzone lub dojrzałe. Termin ten jest powszechnie używany do opisania nici DNA lub RNA , które aktywnie przechodzą syntezę podczas odpowiednio replikacji lub transkrypcji , lub czasami kompletnej, w pełni transkrybowanej cząsteczki RNA przed dokonaniem jakichkolwiek zmian ( np. poliadenylacja lub edycja RNA ) lub łańcuch peptydowy aktywnie poddawany translacji przez rybosom .
- ncDNA
- Zobacz niekodujące DNA .
- ncRNA
- Patrz niekodujący RNA .
- negatywna kontrola
- Rodzaj regulacji genu , w którym represor wiąże się z operatorem w górę od regionu kodującego iw ten sposób zapobiega transkrypcji przez polimerazę RNA . Induktor jest niezbędny do włączenia transkrypcji w kontroli pozytywnej .
- ujemna (-) nić sensowna
- Zobacz nić wzorcowa .
- ujemna
- mutacja neutralna superskręcenia
- 1. Dowolna mutacja sekwencji kwasu nukleinowego które nie jest ani korzystne, ani szkodliwe dla zdolności organizmu do przeżycia i reprodukcji.
- 2. Każda mutacja, w przypadku której dobór naturalny nie wpływa na rozprzestrzenianie się mutacji w obrębie populacji.
- nick
- nick translacja
- nick enzym
- dinukleotyd nikotynamidoadeninowy (NAD)
- fosforan dinukleotydu nikotynamidoadeninowego (NADP, NADP+)
- zasada azotowa
- Dowolny związek organiczny zawierający atom azotu , który ma właściwości chemiczne zasady . Zestaw pięciu określonych zasad azotowych – adenina ( A ), guanina ( G ), cytozyna ( C ), tymina ( T ) i uracyl ( U ) – są szczególnie istotne dla biologii, ponieważ są składnikami nukleotydów , które z kolei są pierwszorzędowe monomery które tworzą kwasy nukleinowe .
- niekodujące DNA (ncDNA)
- Dowolny odcinek DNA , który nie koduje sekwencji, która ostatecznie może ulec transkrypcji i translacji w białko . W większości organizmów tylko niewielka część genomu składa się z DNA kodującego białka, chociaż proporcja ta różni się znacznie w zależności od gatunku. Niektóre niekodujące DNA mogą nadal ulegać transkrypcji do funkcjonalnego niekodującego RNA (jak w przypadku transferowych RNA ) lub mogą służyć ważnym procesom rozwojowym lub regulacyjne ; inne regiony (jak w przypadku tak zwanego „ śmieciowego DNA ”) wydają się nie mieć żadnej znanej funkcji biologicznej.
- niekodujący RNA (ncRNA)
- Każda cząsteczka RNA , która ostatecznie nie ulega translacji do białka . Sekwencja DNA , z której transkrybowany jest funkcjonalny niekodujący RNA, jest często określana jako „gen RNA”. Liczne rodzaje niekodujących RNA niezbędnych do prawidłowego funkcjonowania genomu są wytwarzane konstytutywnie, w tym transfer RNA (tRNA), rybosomalny RNA (rRNA), mikroRNA (miRNA) i mały interferujący RNA (siRNA); inne niekodujące RNA (czasami opisywane jako „śmieciowe RNA”) nie mają żadnej znanej funkcji i prawdopodobnie są produktem fałszywej transkrypcji.
- niehomologiczne łączenie końców (NHEJ)
- dziedziczenie niemendlowskie
- Dowolny wzorzec dziedziczenia , w którym cechy nie ulegają segregacji zgodnie z prawami Mendla , które opisują łatwo obserwowalne dziedziczenie dyskretnie zmiennych cech fenotypowych, na które wpływają pojedyncze geny na jądrze chromosomy . Chociaż prawidłowo wyjaśniają wiele podstawowych obserwacji dotyczących dziedziczenia, prawa Mendla są przydatne tylko w najprostszych i najbardziej ogólnych przypadkach; istnieje wiele procesów i zjawisk genetycznych, zarówno normalnych, jak i nienormalnych, które je naruszają, takich jak niepełna dominacja , kodominacja , powiązania genetyczne , interakcje epistatyczne i cechy poligeniczne , nielosowa segregacja chromosomów , dziedziczenie pozajądrowe , konwersja genów i wiele epigenetycznych zjawiska.
- nielosowa segregacja chromosomów
- nietranskrybowana przerywnik (NTS)
- Patrz przerywnik .
- nić niekodująca
- Patrz nić wzorcowa .
- nondysjunkcja
- Nieprawidłowa segregacja homologicznych chromosomów lub chromatyd siostrzanych podczas podziału komórki . Nondysjunkcja skutkuje komórkami potomnymi, które są aneuploidami , zawierającymi nieprawidłową liczbę jednego lub więcej określonych chromosomów . Może to być spowodowane dowolnym z wielu czynników.
- rekombinacja niehomologiczna
- rekombinacja nieodwrotna
- Zobacz nierówne przejście .
- sekwencja niepowtarzająca się
- Szeroko, dowolna sekwencja nukleotydowa lub region genomu , który nie zawiera powtórzonych sekwencji lub w którym powtórzenia nie stanowią większości; lub dowolny segment DNA wykazujący kinetykę ponownego asocjacji oczekiwaną od unikalnej sekwencji.
- bezsensowna mutacja
- Rodzaj mutacji punktowej , która skutkuje przedwczesnym kodonem stop w transkrybowanej sekwencji mRNA , powodując w ten sposób przedwczesne zakończenie translacji , w wyniku czego powstaje skrócone, niekompletne i często niefunkcjonalne białko .
- nonsensowna mutacja niesynonimiczna
- supresorowa
- Rodzaj mutacji , w której zamiana jednej zasady nukleotydowej na inną skutkuje, po transkrypcji i translacji , sekwencją aminokwasową inną niż sekwencja wytwarzana przez oryginalny niezmutowany gen. Ponieważ mutacje niesynonimiczne zawsze skutkują biologiczną zmianą w organizmie, często podlegają silnej presji selekcyjnej . Porównaj mutację synonimiczną .
- Northern blotting
- Metoda blottingu stosowana w biologii molekularnej do wykrywania RNA w próbce. Porównaj Southern blotting , western blotting i wschodnią blotting .
- nRNA
- Patrz jądrowy RNA .
- membrana nuklearna
- Bariera subkomórkowa składająca się z dwóch dwuwarstwowych błon lipidowych , która otacza jądro w komórkach eukariotycznych .
- jądrowy RNA (nRNA)
- Każda cząsteczka RNA zlokalizowana w jądrze komórki , związana z chromosomami lub występująca swobodnie w nukleoplazmie, w tym mały jądrowy RNA (snRNA), wzmacniający RNA (eRNA) i wszystkie nowo transkrybowane niedojrzałe RNA , kodujące lub nie kodowanie , przed ich eksportem do cytosolu (hnRNA).
- nukleaza
- Dowolna klasa enzymów zdolnych do rozszczepiania wiązań fosfodiestrowych łączących sąsiednie nukleotydy w cząsteczce kwasu nukleinowego (przeciwieństwo ligazy ) . Nukleazy mogą nacinać jedną lub przecinać obie nici cząsteczki dupleksu; mogą ciąć losowo lub w określonych sekwencjach rozpoznawania. Są wszechobecne i niezbędne dla prawidłowego funkcjonowania komórek, a także są szeroko stosowane w biologii molekularnej .
- kwas nukleinowy
- A długi, polimeryczny makrocząsteczka zbudowana z mniejszych monomerów zwanych nukleotydami , które są chemicznie połączone ze sobą w łańcuchu. Dwa specyficzne typy kwasów nukleinowych, DNA i RNA , są używane w systemach biologicznych do kodowania informacji genetycznej rządzącej budową, rozwojem i zwykłymi procesami wszystkich żywych organizmów. Kolejność lub sekwencja nukleotydów w cząsteczkach DNA i RNA zawiera informacje, które są tłumaczone na białka , które kierują wszystkimi reakcjami chemicznymi niezbędnymi do życia.
- sekwencja kwasu nukleinowego
- Dokładna kolejność kolejno połączonych nukleotydów w cząsteczce kwasu nukleinowego, takiej jak DNA lub RNA . Długie sekwencje nukleotydów są głównym środkiem, za pomocą którego systemy biologiczne przechowują informację genetyczną, a zatem dokładna replikacja , transkrypcja i translacja takich sekwencji ma ogromne znaczenie, aby informacje nie zostały utracone lub uszkodzone. Sekwencje kwasu nukleinowego mogą być równoważnie określane jako sekwencje zasad azotowych , nukleozasady , nukleotydy lub pary zasad i odpowiadają bezpośrednio sekwencjom kodonów i aminokwasów .
- nukleozasada
- Dowolna z pięciu podstawowych lub kanonicznych zasad azotowych – adenina ( A ), guanina ( G ), cytozyna ( C ), tymina ( T ) i uracyl ( U ) – które tworzą nukleozydy i nukleotydy , z których te ostatnie są podstawowym budulcem bloki kwasów nukleinowych . Zdolność tych zasad do tworzenia par zasad poprzez wiązania wodorowe , a także ich płaski, zwarty trójwymiarowy profil, pozwala im „układać się” jeden na drugim i prowadzi bezpośrednio do długołańcuchowych struktur DNA i RNA . Podczas zapisywania sekwencji w notacji skróconej, litera N jest często używana do reprezentowania nukleotydu zawierającego rodzajową lub niezidentyfikowaną zasadę nukleinową.
- nukleoid
- Obszar o nieregularnym kształcie w komórce prokariotycznej, który zawiera większość lub całość materiału genetycznego komórki, ale nie jest otoczony błoną jądrową , jak u eukariontów.
- jąderko
- Organelle w jądrze komórek eukariotycznych , które składają się z białek, DNA i RNA i służą jako miejsce syntezy rybosomów .
- nukleoproteina
- Każde białko , które jest chemicznie związane lub sprzężone z cząsteczką kwasu nukleinowego . Przykłady obejmują rybosomy , nukleosomy i wiele enzymów.
- nukleozydaza
- Dowolny z klasy enzymów, które katalizują rozkład nukleozydów na składowe zasady azotowe i cukry pentozowe .
- nukleozyd
- Cząsteczka organiczna złożona z zasady azotowej związanej z pięciowęglowym cukrem ( rybozą lub dezoksyrybozą ). Nukleotyd dodatkowo zawiera jedną lub więcej grup fosforanowych .
- nukleosom
- Podstawowa strukturalna podjednostka chromatyny używana do pakowania jądrowego DNA, takiego jak chromosomy, składająca się z rdzeniowej cząstki ośmiu białek histonowych , wokół których dwuniciowy DNA jest owinięty w sposób podobny do nici nawiniętej na szpulę. Techniczna definicja nukleosomu obejmuje odcinek DNA o długości około 146 par zasad, który wykonuje 1,67 obrotu w lewo, gdy zwija się wokół rdzenia histonowego, a także odcinek łącznikowego DNA (ogólnie 38–80 pz) łącząc ją z sąsiednią cząstką rdzenia, chociaż termin ten jest często używany w odniesieniu do samej cząstki rdzenia. Długie serie nukleosomów są dalej kondensowane przez asocjację z histonem H1 w struktury wyższego rzędu, takie jak włókna 30 nm i ostatecznie superskręcone chromatydy . Ponieważ interakcja histon-DNA ogranicza dostęp do cząsteczki DNA przez inne białka i RNA, takie jak czynniki transkrypcyjne , precyzyjne umiejscowienie nukleosomów wzdłuż sekwencji DNA odgrywa fundamentalną rolę w kontrolowaniu, czy geny ulegają transkrypcji . i wyrażane , a zatem mechanizmy poruszania się i wyrzucania nukleosomów ewoluowały jako środki regulujące ekspresję poszczególnych loci.
- nukleotyd
- Cząsteczka organiczna służąca jako monomer lub podjednostka polimerów kwasów nukleinowych , w tym RNA i DNA . Każdy nukleotyd składa się z trzech połączonych grup funkcyjnych: zasady azotowej , pięciowęglowego cukru ( rybozy lub dezoksyrybozy ) i pojedynczej grupy fosforanowej . Choć technicznie odrębny, termin „nukleotyd” jest często używany zamiennie z zasadą azotową, zasadą nukleinową i parą zasad w odniesieniu do sekwencji tworzących kwasy nukleinowe. Porównaj nukleozyd .
- jądro
- Otoczone błoną organelle występujące w komórkach eukariotycznych , które zawierają większość materiału genetycznego komórki (zorganizowane w postaci chromosomów ) i kierują czynnościami komórki poprzez regulację ekspresji genów .
- allel zerowy
- Dowolny allel , który stał się niefunkcjonalny w wyniku mutacji genetycznej . Mutacja może spowodować całkowity brak wytworzenia produktu genu lub produkt genu, który nie działa prawidłowo; w obu przypadkach allel można uznać za niefunkcjonalny.
- nullizygotyczna
- nullisomia
- Stan komórki lub organizmu, w którym brakuje wszystkich kopii określonego chromosomu , które są normalne dla jego poziomu ploidalności ; np. w organizmie diploidalnym , pozbawionym obu członków normalnej pary. Nullisomia jest często śmiertelna na wczesnym etapie rozwoju.

Zasady nukleinowe (niebieskie) to pięć specyficznych zasad azotowych kanonicznie stosowanych w DNA i RNA. Nukleozasada związana z cukrem pentozowym ( rybozą lub dezoksyrybozą ; kolor żółty) jest znana jako nukleozyd (żółty + niebieski). Nukleozyd związany z pojedynczą grupą fosforanową (czerwony) jest znany jako monofosforan nukleozydu (NMP) lub nukleotyd ( czerwony + żółty + niebieski). Gdy nie są włączone do łańcucha kwasu nukleinowego, wolne nukleozydy mogą wiązać wiele grup fosforanowych: dwa fosforany dają difosforan nukleozydu (NDP), a trzy daje trifosforan nukleozydu (NTP).
O
- ochra
- Jeden z trzech kodonów stop używanych w standardowym kodzie genetycznym ; w RNA jest określony przez tryplet nukleotydowy UAA . Pozostałe dwa kodony stop to bursztyn i opal .
- Fragmenty Okazaki
- Krótkie sekwencje nukleotydów syntetyzowane w sposób nieciągły przez polimerazę DNA , a następnie łączone ze sobą przez ligazę DNA , tworząc nić opóźnioną podczas replikacji DNA . Fragmenty Okazaki są konsekwencją jednokierunkowości polimerazy DNA, która działa tylko w kierunku od 5' do 3'.
- oligogen
- oligomer
- Dowolna cząsteczka polimeru składająca się ze stosunkowo krótkiej serii połączonych monomerów lub podjednostek; np. oligonukleotyd to krótka seria nukleotydów.
- oligonukleotyd
- Stosunkowo krótki łańcuch reszt kwasu nukleinowego . W laboratorium oligonukleotydy są powszechnie stosowane jako startery lub sondy hybrydyzacyjne do wykrywania obecności większych cząsteczek mRNA lub łączone w dwuwymiarowe mikromacierze do wysokowydajnej analizy sekwencjonowania .
- wszechpotężny
- onkogen supresorowy
- Gen , który może powodować raka . W komórkach nowotworowych takie geny są często zmutowane i/lub wyrażane na nienormalnie wysokich poziomach.
- jeden gen – jeden polipeptyd
- Hipoteza, że istnieje duża klasa genów , w których każdy konkretny gen kieruje syntezą jednego określonego polipeptydu lub białka . W przeszłości uważano, że wszystkie geny i białka mogą podlegać tej zasadzie z definicji, ale obecnie wiadomo, że wiele lub większość białek to kompozyty różnych polipeptydów, a zatem produkt wielu genów, a także, że niektóre geny w ogóle nie kodują polipeptydów ale zamiast tego produkują niekodujące RNA , które nigdy nie ulegają translacji.
- opal
- Jeden z trzech kodonów stop używanych w standardowym kodzie genetycznym ; w RNA jest określony przez tryplet nukleotydów UGA . Pozostałe dwa kodony stop to bursztyn i ochra .
- otwarta chromatyna
- Zobacz euchromatyna .
- otwarta ramka odczytu (ORF)
- Część ramki odczytu , która ma zdolność do translacji z DNA lub RNA na białko; dowolny ciągły odcinek kodonów zawierający a kodon start i kodon stop .
- operator
- Sekwencja regulatorowa w operonie , zwykle zlokalizowana pomiędzy sekwencją promotora a genami strukturalnymi operonu, z którą może wiązać się nieinhibitowane białko represorowe , w ten sposób fizycznie blokując polimerazie RNA inicjację transkrypcji sąsiednich cistronów.
- operon
- Funkcjonalna jednostka ekspresji genów składająca się z grupy sąsiadujących ze sobą genów strukturalnych które są zbiorowo pod kontrolą pojedynczego promotora wraz z jedną lub większą liczbą sąsiadujących sekwencji regulatorowych , takich jak operatory , które wpływają na transkrypcję genów strukturalnych. Zestaw genów jest transkrybowany razem, co zwykle skutkuje pojedynczą policistronową cząsteczką informacyjnego RNA , która może następnie podlegać wspólnej translacji lub podlegać splicingowi do tworzenia wielu mRNA, które są tłumaczone niezależnie; w rezultacie geny zawarte w operonie są albo wyrażane razem, albo wcale. Białka regulatorowe, w tym represory , korepresory i aktywatory , zwykle wiążą się specyficznie z sekwencjami regulatorowymi danego operonu; według niektórych definicji geny kodujące te białka regulatorowe są również uważane za część operonu.
- źródło replikacji (ORI)
- sieci operonowej
- Określone miejsce w cząsteczce DNA, w którym rozpoczyna się replikacja DNA . Pochodzenie jest zwykle definiowane przez obecność sekwencji replikatora lub przez specyficzne wzorce chromatyny.
- gen Gen , dla którego nie ma znanych funkcjonalnych homologów poza danym gatunkiem lub linią , i którego historia ewolucyjna jest w związku z tym niejasna.
- sierocy
- orphon
- Dowolna kodująca lub niekodująca sekwencja DNA pochodząca z tandemowej wielogenowej rodziny lub klastra ale jest fizycznie odizolowany od innych genów w rodzinie, ponieważ jest rozproszony w odległym locus w genomie. Orfony są zwykle niefunkcjonalnymi pseudogenami o bardzo zmiennej liczbie kopii .
- ortolog
- Jeden z zestawu genów (lub bardziej ogólnie dowolnej sekwencji DNA wykazującej homologię ), które są obecne w różnych genomach , ale pochodzą od tej samej sekwencji przodków, tj. są bezpośrednio spokrewnione ze sobą przez pionowe pochodzenie z pojedynczego genu lub sekwencji w ostatni wspólny przodek tych genomów. O takich genach lub sekwencjach mówi się, że są ortologiczne . Można wywnioskować, że ortologi są ze sobą spokrewnione na podstawie podobieństwa ich sekwencji; chociaż mogły ewoluować niezależnie w ramach oddzielnych genomów w wyniku mutacji i doboru naturalnego, ich produkty mogą nadal zachowywać podobne struktury, funkcje lub poziomy ekspresji w różnych gatunkach i populacjach. Identyfikacja ortologów okazała się ważna we wnioskowaniu o filogenetycznych między organizmami. Paralog kontrastu .
- krzyżowanie się
- Rozmnażanie płciowe między różnymi rasami lub osobnikami, które może zwiększyć różnorodność genetyczną poprzez wprowadzenie niespokrewnionego materiału genetycznego do populacji hodowlanej. Zdarzenie reprodukcyjne i wynikające z niego potomstwo mogą być określane jako krzyżówka zewnętrzna , a potomstwo mówi się, że jest pozakresowe . Kontrastowy chów wsobny .
- outron
- Sekwencja w pobliżu 5'-końca pierwotnego transkryptu mRNA , która jest usuwana przez specjalną formę splicingu podczas przetwarzanie posttranskrypcyjne . Outrony znajdują się całkowicie poza sekwencjami kodującymi transkrypt , w przeciwieństwie do intronów .
- nadekspresja
- Nieprawidłowo wysoki poziom ekspresji genów , który skutkuje nadmierną liczbą kopii jednego lub więcej produktów genów . Nadekspresja powoduje wyraźny fenotyp związany z genami .
P
- p53
- pachynema
- W mejozie trzeci z pięciu podetapów profazy I , następujący po zygonemie i poprzedzający diplonema . Podczas pachynemy kompleks synaptonemalny ułatwia przejście między synapsami homologicznymi , a centrosomy zaczynają się od siebie oddalać.
- sekwencja palindromiczna
- Sekwencja kwasu nukleinowego dwuniciowej cząsteczki DNA lub RNA , w której jednokierunkowa sekwencja (np. 5' do 3') nukleotydów na jednej nici pasuje do sekwencji w tym samym kierunku (np. 5' do 3') na nici komplementarnej . Innymi słowy, mówi się, że sekwencja nukleotydów jest palindromiczna, jeśli jest równa własnemu odwrotnemu dopełnieniu . Motywy palindromiczne są powszechne w większości genomów i mogą tworzyć spinki do włosów .
- inwersja paracentryczna
- A inwersja chromosomalna , w której odwrócony segment nie obejmuje centromeru chromosomu . Kontrast perycentrycznej inwersji .
- paralog
- Jeden z zestawu genów (lub, bardziej ogólnie, dowolne sekwencje DNA wykazujące homologię ), które są bezpośrednio ze sobą powiązane poprzez jedno lub więcej zdarzeń duplikacji genetycznej; o takich genach lub sekwencjach mówi się, że są paralogiczne . Paralogi wynikają z duplikacji pojedynczej sekwencji w obrębie jednego genomu a następnie późniejsza rozbieżność zduplikowanych sekwencji w wyniku mutacji i doboru naturalnego (albo w obrębie pierwotnego genomu, albo podczas specjacji w różnych genomach). Kontrast ortolog .
- pasożytnicze DNA
- Zobacz samolubny element genetyczny .
- dominacja częściowa
- Zobacz dominację niepełną .
- dziedziczenie cząsteczkowe
- Jedna z definiujących koncepcji dziedziczenia mendlowskiego , według której cechy fenotypowe są lub mogą być dziedziczone poprzez przekazywanie „ dyskretne cząstki „z pokolenia na pokolenie. Cząsteczki te mogą nie mieć wykrywalnych efektów w każdym pokoleniu, ale mimo to zachowują zdolność do ekspresji w kolejnych pokoleniach.
- pasażer
- Fragment DNA będący przedmiotem zainteresowania przeznaczony do składania w „nośnik”, taki jak wektor plazmidowy a następnie sklonowany .patrokliniczny
- (
- z potomstwa ) Pod względem genotypowym lub fenotypowym bardziej podobny do rodzica płci męskiej niż do rodzica płci żeńskiej; pochodzi od ojca. Kontrast matrokliniczny .
- PCR
- Patrz reakcja łańcuchowa polimerazy .
- Produkt PCR
- Patrz amplikon .
- penetracja
- wykresu rodowodowego
- Odsetek osobników z danym genotypem , u których występuje powiązany fenotyp , zwykle podawany w procentach. Ze względu na wiele złożonych interakcji, które regulują ekspresję genów , ten sam allel może wytworzyć obserwowalny fenotyp u jednego osobnika, ale nie u innego. Jeśli mniej niż 100% osobników w populacji niosącej interesujący genotyp również wykazuje ekspresję powiązanego fenotypu, można powiedzieć, że zarówno genotyp, jak i fenotyp wykazują niepełną penetrację . Penetracja określa ilościowo prawdopodobieństwo, że allel spowoduje ekspresję związanego z nim fenotypu w dowolnej formie, tj. w jakimkolwiek stopniu, który odróżnia indywidualnego nosiciela od osobników bez allelu. Porównaj ekspresyjność .
- peptyd
- Krótki łańcuch monomerów aminokwasów połączone kowalencyjnymi wiązaniami peptydowymi . Peptydy są podstawowymi budulcami dłuższych polipeptydowych , a tym samym białek .
- wiązanie peptydowe
- Kowalencyjne wiązanie chemiczne między grupą karboksylową jednego aminokwasu a grupą aminową sąsiedniego aminokwasu w łańcuchu peptydowym , utworzone w reakcji odwodnienia katalizowanej przez peptydylotransferazę , enzym występujący w rybosomie , podczas translacji .
- pericentric
- (genu lub regionu chromosomu) Umieszczony w pobliżu centromeru chromosomu .
- inwersja pericentryczna
- Inwersja chromosomalna , w której odwrócony segment obejmuje centromer chromosomu . Kontrast paracentrycznej inwersji .
- wszechobecna transkrypcja
- farmakogenomika
- Badanie roli odgrywanej przez genom w odpowiedzi organizmu na leki farmaceutyczne, łączące dziedziny farmakologii i genomiki .
- fenom
- Kompletny zestaw fenotypów , które są lub mogą być wyrażane przez genom , komórkę, tkankę, organizm lub gatunek; suma wszystkich jego oczywistych właściwości lub cech chemicznych, morfologicznych i behawioralnych.
- opóźnienie fenotypowe
- Opóźnienie fenotypowej ekspresji mutacji genetycznej spowodowane czasem potrzebnym do ujawnienia się zmian w dotkniętych szlakach biochemicznych.
- fenotyp
- Zbiór obserwowalnych cech morfologicznych, fizjologicznych i behawioralnych an organizmu , które wynikają z ekspresji genotypu organizmu oraz wpływu czynników środowiskowych i interakcji między nimi.
- szkielet fosforanowy
- Liniowy łańcuch naprzemiennych związków fosforanowych i cukrowych, który powstaje w wyniku połączenia kolejnych nukleotydów w tej samej nici cząsteczki kwasu nukleinowego i który służy jako szkielet strukturalny kwasu nukleinowego. Każda pojedyncza nić jest utrzymywana razem przez pojedynczy szkielet składający się z powtarzających się serii wiązań fosfodiestrowych między grupą fosforanową jednego nukleotydu a cukrami rybozy lub dezoksyrybozy sąsiednich nukleotydów. Wiązania te są tworzone przez ligazy i łamane przez dezoksyrybonukleazy .
- wiązanie fosfodiestrowe
- Para wiązań estrowych łączących cząsteczkę fosforanową z dwoma pierścieniami pentozowymi kolejnych nukleozydów na tej samej nici kwasu nukleinowego . Każdy fosforan tworzy wiązanie kowalencyjne z węglem 3' jednej pentozy i węglem 5' sąsiedniej pentozy; powtarzająca się seria takich wiązań, która utrzymuje razem długi łańcuch nukleotydów w DNA i RNA , jest znana jako szkielet fosforanowy lub fosfodiestrowy .
- filogenetyka
- Badanie historii ewolucji i związków między osobnikami lub grupami organizmów, takimi jak gatunki lub populacje, za pomocą metod oceniających podobieństwa i różnice między obserwowanymi cechami dziedzicznymi , w tym cechami morfologicznymi i sekwencjami DNA . Graficzna prezentacja danych z takich analiz jest znana jako filogeneza lub drzewo filogenetyczne .
- piRNA
- Patrz RNA oddziałujący z Piwi .
- Plazmid
- RNA oddziałujący z Piwi
- Każda mała cząsteczka DNA , która jest fizycznie oddzielona od większej części chromosomalnego DNA i może replikować się niezależnie. Plazmidy są najczęściej spotykane jako małe, koliste, dwuniciowe cząsteczki DNA u prokariotów , takich jak bakterie , chociaż czasami występują również u archeonów i eukariontów .
- plejotropia
- Zjawisko, w którym jeden gen wpływa na dwie lub więcej pozornie niezwiązanych ze sobą cech fenotypowych za pomocą jednego z kilku odrębnych, ale potencjalnie nakładających się mechanizmów.
- ploidia
- Liczba kompletnych zestawów chromosomów w komórce , a tym samym liczba możliwych alleli obecnych w komórce w danym locus autosomalnym .
- nić plus
- Patrz nić kodowania .
- mutacja punktowa
- Mutacja , w wyniku której pojedynczy nukleotyd zasada jest zmieniana, wstawiana lub usuwana z sekwencji DNA lub RNA.
- poliadenylacja
- ogona poli(A)
- Dodanie serii wielu rybonukleotydów adenozynowych , znanych jako ogon poli(A) , do 3'-końca pierwotnego transkryptu RNA , zwykle matrycowego RNA . Klasa modyfikacji potranskrypcyjnych , poliadenylacja służy różnym celom w różnych typach komórek i organizmach. U eukariontów dodanie ogona poli (A) jest ważnym krokiem w przetwarzaniu surowego transkryptu w dojrzały mRNA, gotowy do eksportu do cytoplazmy, w której zachodzi translacja ; u wielu bakterii poliadenylacja ma odwrotną funkcję, zamiast tego sprzyja degradacji RNA. poligen
- cecha
- poligeniczna
- Każda cecha fenotypowa będąca pod bezpośrednią kontrolą więcej niż jednego genu . Cechy poligeniczne są często cechami ilościowymi .
- polilinker
- Zobacz wiele stron do klonowania .
- polimeraza
- Dowolny z klasy enzymów, które katalizują syntezę cząsteczek polimerowych , zwłaszcza polimerów kwasu nukleinowego , zazwyczaj poprzez pobudzanie parowania wolnych nukleotydów z nukleotydami z istniejącej komplementarnej matrycy . Polimerazy DNA i polimerazy RNA są niezbędne odpowiednio do replikacji i transkrypcji DNA .
- reakcja łańcuchowa polimerazy (PCR)
- Dowolna z szerokiej gamy metod biologii molekularnej polegająca na szybkim wytwarzaniu milionów lub miliardów kopii określonej sekwencji DNA , umożliwiająca naukowcom selektywne amplifikowanie fragmentów bardzo małej próbki do ilości wystarczającej do szczegółowego zbadania. W najprostszej formie PCR obejmuje inkubację docelowej próbki DNA o znanej lub nieznanej sekwencji z mieszaniną reakcyjną składającą się ze starterów oligonukleotydowych , termostabilnej polimerazy DNA i wolnych trójfosforanów dezoksyrybonukleotydów (dNTP), z których wszystkie są dostarczane w nadmiarze. Ta mieszanina jest następnie naprzemiennie ogrzewana i chłodzona do z góry określonych temperatur przez z góry określony okres czasu, zgodnie z określonym schematem, który jest powtarzany przez wiele cykli, zwykle w termocyklerze, który automatycznie kontroluje wymagane zmiany temperatury. W każdym cyklu, z których najbardziej podstawowy obejmuje denaturacji , fazę wyżarzania i fazę elongacji , kopie zsyntetyzowane w poprzednim cyklu są używane jako matryce do syntezy w następnym cyklu, powodując reakcja łańcuchowa , która powoduje wykładniczy wzrost całkowitej liczby kopii w mieszaninie reakcyjnej. Amplifikacja metodą PCR stała się standardową techniką praktycznie we wszystkich laboratoriach biologii molekularnej.
- polimorfizm
- Regularne i jednoczesne występowanie w tej samej populacji dwóch lub więcej alleli (lub genotypów ) w tym samym locus z częstością , której nie można wytłumaczyć samą mutacją powtarzającą się (na ogół co najmniej 1%), co sugeruje, że allele wielokrotne są stabilnie dziedziczone przez członków populacji.
- polipeptyd
- Długi, ciągły i nierozgałęziony polimerowy łańcuch monomerów aminokwasów połączonych kowalencyjnymi wiązaniami peptydowymi , zwykle dłuższy niż peptyd . Białka na ogół składają się z jednego lub więcej polipeptydów ułożonych w biologicznie funkcjonalny sposób.
- polifeny
- Zobacz plejotropia .
- poliploidalny
- (komórki lub organizmu) mający więcej niż dwie homologiczne kopie każdego chromosomu . Poliploidia może występować jako normalny stan chromosomów w niektórych komórkach lub nawet całych organizmach lub może być wynikiem nieprawidłowego podziału komórki lub mutacji powodującej duplikację całego zestawu chromosomów. Porównaj haploidalne i diploidalne ; zobacz także ploidalność .
- polisom
- Kompleks cząsteczki informacyjnego RNA i dwóch lub więcej rybosomów , których zadaniem jest translacja transkryptu mRNA na polipeptyd .
- polisomia
- Stan komórki lub organizmu mającego co najmniej jedną kopię określonego chromosomu więcej niż jest to normalne dla jego poziomu ploidalności , np. mówi się, że organizm diploidalny z trzema kopiami danego chromosomu wykazuje trisomię . Każda polisomia jest rodzajem aneuploidii .
- genetyka populacji
- Poddziedzina genetyki i biologii ewolucyjnej , która bada różnice genetyczne w populacjach organizmów i między nimi.
- efekt pozycji
- Każdy wpływ na ekspresję lub funkcjonalność genu lub sekwencji, który jest konsekwencją jego lokalizacji lub pozycji w chromosomie lub innej cząsteczce DNA. Dokładna lokalizacja sekwencji w stosunku do innych sekwencji i struktur ma silny wpływ na jej aktywność i inne właściwości, ponieważ różne loci na tej samej cząsteczce mogą mieć zasadniczo różne podłoże genetyczne i środowiska fizyczne/chemiczne, które również mogą zmieniać się w czasie. Na przykład transkrypcja genu znajdującego się bardzo blisko nukleosomu , centromeru lub telomeru jest często tłumiona lub całkowicie uniemożliwiana, ponieważ białka tworzące te struktury blokują dostęp do DNA przez czynniki transkrypcyjne , podczas gdy ten sam gen jest transkrybowany na poziomie znacznie wyższy wskaźnik, gdy znajduje się w euchromatynie . Bliskość do promotory , wzmacniacze i inne elementy regulatorowe , a także regiony częstej transpozycji przez elementy ruchome , mogą również bezpośrednio wpływać na ekspresję; umiejscowienie w pobliżu końca ramienia chromosomu lub wspólnych krzyżowania się może wpływać na czas replikacji i prawdopodobieństwo rekombinacji . Efekty pozycji są głównym przedmiotem badań w dziedzinie dziedziczenia epigenetycznego .
- klonowanie pozycyjne
- Strategia identyfikacji i klonowania potencjalnego genu oparta na znajomości samego locus lub pozycji iz niewielką ilością informacji lub bez informacji o jej produktach lub funkcji, w przeciwieństwie do klonowania funkcjonalnego . Ta metoda zwykle rozpoczyna się od porównania genomów osobników wykazujących fenotyp o nieznanym pochodzeniu (często jest to choroba dziedziczna ) i identyfikacji wspólnych dla nich markerów genetycznych . Regiony zdefiniowane przez markery flankujące jeden lub więcej genów będących przedmiotem zainteresowania są klonowane , a geny znajdujące się pomiędzy markerami można następnie zidentyfikować dowolnymi różnymi sposobami , np . osobników lub poprzez testowanie zdolności domniemanego genu do uratowania zmutowanego fenotypu.
- dodatnia (+) nić sensowna
- Patrz nić kodująca .
- modyfikacja potranskrypcyjna modyfikacja
- potranslacyjna
- pierwotny transkrypt
- Nieprzetworzona, jednoniciowa cząsteczka RNA wytworzona w wyniku transkrypcji sekwencji DNA , jaka istnieje przed modyfikacjami potranskrypcyjnymi, takimi jak alternatywny splicing, przekształca ją w dojrzały produkt RNA, taki jak mRNA , tRNA lub rRNA . Na przykład prekursorowy mRNA lub pre-mRNA jest rodzajem pierwotnego transkryptu, który po przetworzeniu staje się dojrzałym mRNA gotowym do translacji .
- Elementarz
- Krótki, jednoniciowy oligonukleotyd , zwykle o długości 5–100 zasad, który „primuje” lub inicjuje syntezę kwasu nukleinowego przez hybrydyzację z komplementarną sekwencją na nici matrycy , a tym samym zapewnia istniejący 3'-koniec , z którego może rozciągać się polimeraza nowa nić. Systemy naturalne wykorzystują wyłącznie RNA do inicjacji replikacji i transkrypcji DNA , natomiast in vitro syntezy przeprowadzane w wielu technikach laboratoryjnych, takich jak PCR, często wykorzystują startery DNA . W nowoczesnych laboratoriach startery są starannie projektowane, często w parach „do przodu” i „do tyłu”, w celu uzupełnienia specyficznych i unikalnych sekwencji w genomowym DNA , z uwzględnieniem ich temperatur topnienia i hybrydyzacji , a następnie kupowane od dostawców komercyjnych, którzy tworzą oligonukleotydy na zapotrzebowanie przez syntezę de novo .
- starter dimer (PD)
- priming
- Inicjacja kwasu nukleinowego przez hybrydyzację lub hybrydyzację jednego lub większej liczby starterów z komplementarną sekwencją w obrębie nici matrycy .
- probant
- Termin używany w genealogii medycznej i genealogii do określenia konkretnego przedmiotu, który jest badany lub omawiany.
- sonda
- Dowolny odczynnik używany do wykonania pojedynczego pomiaru w teście biochemicznym, takim jak eksperyment z ekspresją genów. Porównaj reportera .
- zestaw sond
- Zbiór dwóch lub więcej sond zaprojektowanych do pomiaru pojedynczego gatunku cząsteczkowego, taki jak zbiór oligonukleotydów zaprojektowanych do hybrydyzacji z różnymi częściami mRNA transkrypty generowane z pojedynczego genu.
- proces koncepcja genu molekularnego
- prometafaza
- Drugi etap podziału komórki w mitozie , po profazie i poprzedzającej metafazie , podczas którego rozpada się błona jądrowa, znajdujące się wewnątrz chromosomy tworzą kinetochory wokół swoich centromerów , mikrotubule wyłaniające się z biegunów wrzeciona mitotycznego docierają do przestrzeni jądrowej i przyłączają się do kinetochorów i białek motorycznych związane z mikrotubulami zaczynają popychać chromosomy w kierunku środka komórki.
- promotor
- Sekwencja lub region DNA, zwykle o długości 100–1000 par zasad, z którym wiążą się czynniki transkrypcyjne w celu rekrutacji polimerazy RNA do sekwencji i zainicjowania transkrypcji jednego lub większej liczby genów. Promotory znajdują się powyżej genów, które transkrybują, w pobliżu miejsca startu transkrypcji.
- promocja
- Zobacz regulacja w górę .
- profaza
- Pierwszy etap podziału komórki zarówno w mitozie , jak i mejozie , zachodzącej po interfazie i przed prometafazą , podczas której DNA chromosomów ulega kondensacji w chromatynę , jąderko rozpada się, centrosomy przemieszczają się na przeciwne końce komórki i tworzy się wrzeciono mitotyczne .
- białko
- Polimeryczna makrocząsteczka złożona z jednego lub więcej długich łańcuchów aminokwasów połączonych ze sobą wiązania peptydowe . Białka to trójwymiarowe struktury powstające, gdy te łańcuchy składają się w określone układy wyższego rzędu po translacji i to właśnie ta pofałdowana struktura określa aktywność chemiczną białka, a tym samym jego funkcję biologiczną. Wszechobecne i fundamentalne we wszystkich żywych organizmach białka są podstawowym środkiem wykonywania funkcji życiowych, uczestnicząc w ogromnej większości reakcji biochemicznych zachodzących wewnątrz i na zewnątrz komórek.
- aminokwas proteinogenny
- Dowolny z 20 kanonicznych aminokwasów które są kodowane przez standardowy kod genetyczny i włączane do peptydów i ostatecznie do białek podczas translacji . Termin może również obejmować dodatkowe dwa aminokwasy kodowane przez niestandardowe kody , które mogą być włączone przez specjalne mechanizmy translacji.
- proteom
- Cały zestaw białek , który jest lub może być eksprymowany przez określony genom , komórka, tkanka lub gatunek w określonym czasie (np. w ciągu jednego życia lub na określonym etapie rozwoju) lub w określonych warunkach (np. w przypadku wystąpienia określonej choroby).
- pseudoallel
- Dowolny z dwóch lub więcej różnych genów lub sekwencji, które mają taki sam lub podobny wkład w fenotyp , a zatem wydają się być autentycznymi allelami , ale w rzeczywistości nie są strukturalnie alleliczne (tj. nie zajmują homologicznych loci na homologicznych chromosomach ).
- pseudogen
- Niefunkcjonalna sekwencja DNA, która przypomina sekwencję funkcjonalną gen . Pseudogeny są zwykle zbędnymi kopiami funkcjonalnych genów, które zostały zduplikowane przez naturalne procesy, z wyjątkiem tego, że brakuje im sekwencji regulatorowych niezbędnych do prawidłowej transkrypcji lub translacji lub zawierają inne defekty, takie jak mutacje przesunięcia ramki odczytu , przedwczesne kodony stop lub brakujące introny .
- pseudopoliploidia
- 1. Stan, w którym liczba chromosomów w zestawie chromosomów ulega podwojeniu (lub potroić itd.), ale bez odpowiedniego wzrostu rzeczywistej ilości materiału genetycznego (tj. poziomu ploidalności ). Dzieje się tak, gdy chromosomy normalnego dopełnienia chromosomu (np. diploidalnego ) ulegają fragmentacji na mniejsze części, zwiększając całkowitą liczbę pojedynczych chromosomów, ale nie tworząc dodatkowych homologicznych kopii tych chromosomów (tak, że komórka pozostaje diploidalna).
- 2. Jakakolwiek liczbowa zależność między zestawami chromosomów w grupach spokrewnionych organizmów, która sugeruje, że niektóre z tych organizmów są poliploidami innych, podczas gdy w rzeczywistości nimi nie są.
- Kwadrat Punnetta
- Diagram tabelaryczny używany do przewidywania możliwych genotypów , które mogą odziedziczyć potomstwo z określonej krzyżówki lub eksperymentu hodowlanego, poprzez podsumowanie wszystkich różnych kombinacji alleli matczynych z allelami ojcowskimi. Otrzymaną tabelę można następnie wykorzystać do określenia prawdopodobieństwa, że potomstwo będzie miało określony genotyp. Użyteczność kwadratów Punnetta jest ograniczona do dyskretnych fenotypów dziedziczonych według prostych wzorców mendlowskich .
- czystej krwi
- puryna
- Dwupierścieniowy heterocykliczny związek organiczny, który wraz z pirymidyną jest jedną z dwóch cząsteczek, z których pochodzą wszystkie zasady azotowe (w tym zasady nukleinowe stosowane w DNA i RNA ). Adenina ( A ) i guanina ( G ) zaliczane są do puryn. Litera R jest czasami używana do wskazania ogólnej puryny; np. w odczytanej sekwencji nukleotydów, R można użyć do wskazania, że nukleozasada purynowa, A lub G , może być podstawiona we wskazanej pozycji.
- domniemany gen
- Specyficzna sekwencja nukleotydów podejrzewana o bycie funkcjonalnym genem na podstawie identyfikacji jego otwartej ramki odczytu . Mówi się, że gen jest „domniemany” w tym sensie, że nie opisano jeszcze żadnej funkcji jego produktów .
- pirymidyna
- Jednopierścieniowy heterocykliczny związek organiczny, który wraz z puryną jest jedną z dwóch cząsteczek, z których pochodzą wszystkie zasady azotowe (w tym nukleozasady stosowane w DNA i RNA ). Cytozyna ( C ), tymina ( T ) i uracyl ( U ) są klasyfikowane jako pirymidyny. Litera Y jest czasami używana do wskazania ogólnej pirymidyny; np. w odczytanej sekwencji nukleotydowej, Y może być użyte do wskazania, że nukleozasada pirymidyny – C , T lub U – może być podstawiona we wskazanej pozycji.
- dimer pirymidyny
- Rodzaj uszkodzenia molekularnego spowodowanego fotochemicznym uszkodzeniem DNA lub RNA , w wyniku którego ekspozycja na promieniowanie ultrafioletowe (UV) indukuje tworzenie wiązań kowalencyjnych między zasadami pirymidynowymi zajmującymi sąsiednie pozycje w tej samej nici polinukleotydowej , co z kolei może powodować lokalne zmiany konformacyjne w strukturze drugorzędowej i zapobiegać parowaniu zasad z przeciwną nicią. W DNA reakcja dimeryzacji zachodzi między sąsiednimi tyminy i cytozyny ( T − T , C − C lub T − C ); może również wystąpić między resztami cytozyny i uracylu w dwuniciowym RNA . Dimery pirymidynowe są zwykle szybko korygowane przez naprawy przez wycinanie nukleotydów , ale nieskorygowane uszkodzenia mogą hamować lub zatrzymywać aktywność polimerazy podczas transkrypcji lub replikacji .
Q
- genetyka ilościowa
- Gałąź genetyki populacyjnej , która bada fenotypy , które zmieniają się w sposób ciągły (takie jak wzrost lub masa), w przeciwieństwie do fenotypów, które mieszczą się w dyskretnie identyfikowalnych kategoriach (takich jak kolor oczu lub obecność lub brak określonej cechy ) . Genetyka ilościowa wykorzystuje metody i koncepcje statystyczne do łączenia stale dystrybuowanych wartości fenotypowych z określonymi genotypami i produktami genów .
- ilościowy PCR (qPCR)
- cecha ilościowa
- locus cechy ilościowej (QTL)
R
- rDNA
- 1. Skrót od rekombinowanego DNA .
- 2. Skrót od rybosomalnego DNA .
- ramka odczytu
- Sposób podziału sekwencji nukleotydów w cząsteczce DNA lub RNA na zestaw kolejnych, nienakładających się na siebie trojaczków, który jest „odczytywany” przez białka podczas transkrypcji i replikacji . W kodowaniu DNA każda trójka jest określana jako kodon , który odpowiada określonemu aminokwasowi podczas tłumaczenie . Ogólnie rzecz biorąc, tylko jedna ramka odczytu (tzw. otwarta ramka odczytu ) w danej sekcji kwasu nukleinowego może być wykorzystana do wytworzenia funkcjonalnych białek, ale istnieją wyjątki w kilku organizmach. Mutacja przesunięcia ramki powoduje przesunięcie w normalnej ramce odczytu i wpływa na wszystkie dalsze kodony.
- PCR w czasie rzeczywistym (rtPCR)
- Patrz ilościowy PCR .
- kinetyka reasocjacji Pomiar
- i manipulacja szybkością ponownego połączenia komplementarnych nici DNA , ogólnie przez ogrzewanie i denaturację dwuniciowej cząsteczki w pojedyncze nici , a następnie obserwowanie ich ponownej hybrydyzacji w niższej temperaturze. Ponieważ para zasad G + C wymaga więcej energii do hybrydyzacji niż para zasad A + T , szybkość ponownego hybrydyzacji między dwiema niciami zależy częściowo od ich sekwencji nukleotydów , a zatem możliwe jest przewidzenie lub oszacowanie sekwencji cząsteczki dupleksu na podstawie czas potrzebny do pełnej hybrydyzacji. Badana jest kinetyka ponownego asocjacji Analiza C0t : fragmenty renaturujące się przy niskich wartościach C0t mają zwykle wysoce powtarzalne sekwencje , podczas gdy wyższe wartości C0t implikują bardziej unikalne sekwencje.
- recesywność
- Związek między allelami genu , w którym jeden allel wywiera wpływ na fenotyp , który jest silniejszy lub „maskowany” przez udział innego allelu w tym samym locus ; mówi się , że pierwszy allel i związana z nim cecha fenotypowa są recesywne , a drugi allel i związana z nim cecha są uważane za dominujące . Często recesywne allele kodują nieefektywne lub dysfunkcyjne białka. Podobnie jak dominacja, recesywność nie jest nieodłączną cechą żadnego allelu lub fenotypu, ale po prostu opisuje jego związek z jednym lub większą liczbą innych alleli lub fenotypów. W skrócie genetyki allele recesywne są często reprezentowane przez małą literę (np. „a”, w przeciwieństwie do dominującej „A”).
- krzyż
- wzajemny Krzyżowanie eksperyment mający na celu sprawdzenie, czy płeć rodziców wpływa na dziedziczenie określonej cechy. W krzyżówkach, w których rodzice różnią się genotypem lub fenotypem lub jednym i drugim, a zatem tylko jeden z rodziców (rodzic płci męskiej lub żeńskiej) wykazuje interesującą cechę, krzyż odwrotny jest odwrotnością, w której rodzic drugiej płci wyraża zamiast tego interesująca cecha. Na przykład, jeśli w pierwszej krzyżówce krzyżuje się samca wykazującego cechę z samicą, która jej nie wykazuje, to w krzyżówce odwrotnej krzyżuje się samicę wykazującą tę cechę z samcem, który jej nie wyraża. Obserwując potomstwo wynikające z każdej krzyżówki, genetycy mogą wyciągać wnioski na temat tego, który chromosom płciowy wpływa na ekspresję cechy.
- wzajemna hybryda
- Potomstwo hybrydy powstałe w wyniku wzajemnej krzyżówki między rodzicami różniącymi się genotypem lub fenotypem lub jednym i drugim .
- translokacja wzajemna
- Rodzaj translokacji chromosomowej , w wyniku której następuje wzajemna wymiana segmentów chromosomów między dwoma lub więcej niehomologicznymi chromosomami . Gdy wymiana materiału jest równomiernie zrównoważona, wzajemne translokacje są zwykle nieszkodliwe.
- recombinant DNA (rDNA)
- Dowolna cząsteczka DNA , w której laboratoryjne metody rekombinacji genetycznej połączyły materiał genetyczny z wielu źródeł, tworząc w ten sposób sekwencję , która inaczej nie zostałaby znaleziona w naturalnie występującym genomie. Ponieważ cząsteczki DNA ze wszystkich organizmów mają tę samą podstawową strukturę chemiczną i właściwości, sekwencje DNA dowolnego gatunku, a nawet sekwencje utworzone de novo przez sztuczną syntezę genów , można włączyć do rekombinowanych cząsteczek DNA. Technologia rekombinacji DNA jest szeroko stosowana m.in inżynieria genetyczna .
- rekombinacja
- rekombinazy
- Patrz: rekombinacja genetyczna , rekombinacja homologiczna i krzyżowanie chromosomów .
- rekombinator
- Dowolna sekwencja nukleotydów , która zwiększa prawdopodobieństwo rekombinacji homologicznej w pobliskich regionach genomu, np. sekwencja Chi u niektórych gatunków bakterii.
- rozpoznanie
- Najmniejsza jednostka cząsteczki DNA zdolna do rekombinacji homologicznej , czyli parę kolejnych nukleotydów, sąsiadujących ze sobą w układzie cis .
- regulon
- Grupa niesąsiadujących ze sobą genów , które są regulowane jako jednostka, generalnie dzięki temu, że ich ekspresja jest kontrolowana przez ten sam element regulatorowy lub zestaw elementów, np. ten sam represor lub aktywator . Termin ten jest najczęściej używany w przypadku prokariotów, gdzie regulon może składać się z genów z wielu operonów .
- powtarzające
- się DNA
- Region lub fragment DNA składający się głównie lub całkowicie z powtarzających się sekwencji nukleotydowych .
- mutacja zastępcza
- Zobacz mutację niesynonimiczną .
- replikacja
- 1. Proces, w którym pewne cząsteczki biologiczne, zwłaszcza kwasy nukleinowe DNA i RNA , wytwarzają swoje kopie.
- 2. Technika stosowana do szacowania zmienności technicznej i biologicznej w eksperymentach do analizy statystycznej danych z mikromacierzy . Repliki mogą być replikami technicznymi , takie jak zamiany barwników lub powtarzane hybrydyzacje macierzowe lub replikaty biologiczne , próbki biologiczne z oddzielnych eksperymentów, które są wykorzystywane do testowania skutków tej samej eksperymentalnej obróbki.
- oko replikacyjne
- Struktura w kształcie oka, która tworzy się, gdy para widełek replikacyjnych , z których każdy odrasta od początku , oddziela nici podwójnej helisy podczas replikacji DNA .
- widełki replikacyjne
- szybkość replikacji
- Szybkość, z jaką dezoksyrybonukleotydy są włączane do wydłużającego się łańcucha przez polimerazy DNA podczas replikacji DNA ; lub bardziej ogólnie szybkość, z jaką dowolny chromosom, genom, komórka lub organizm tworzy kompletną, niezależnie funkcjonalną kopię samego siebie.
- replikator
- 1. Dowolny fragment lub region DNA, który zawiera początek replikacji .
- 2. Każda cząsteczka lub struktura zdolna do kopiowania się ; mianowicie kwasy nukleinowe , ale także kryształy wielu minerałów, np kaolinit .
- replikon
- Dowolna cząsteczka lub region DNA lub RNA replikujący się z pojedynczego miejsca startu replikacji .
- replisom
- Cały kompleks maszynerii molekularnej, który przeprowadza proces replikacji DNA , w tym wszystkie białka, kwasy nukleinowe i inne cząsteczki, które uczestniczą w aktywnych widełkach replikacyjnych .
- reporter
- Zgodny z MIAME termin opisujący odczynnik używany do wykonania pojedynczego pomiaru w eksperymencie z ekspresją genów. MIAME definiuje to jako „sekwencję nukleotydową obecną w określonym miejscu w macierzy”. Reporter może być segmentem jednoniciowego DNA, który jest kowalencyjnie przyłączony do powierzchni macierzy. Porównaj sondę .
- represja
- genu reporterowego
- Patrz regulacja w dół .
- represor
- Białko wiążące DNA , które hamuje ekspresję jednego lub więcej genów poprzez wiązanie się z operatorem oraz blokowanie przyłączania polimerazy RNA do promotora , zapobiegając w ten sposób transkrypcji . Ten proces jest znany jako negatywna regulacja genów .
- ratowanie
- Przywrócenie wadliwej komórki lub tkanki do zdrowego lub normalnego stanu lub przywrócenie lub przywrócenie zmutowanego genu do jego normalnej funkcjonalności, zwłaszcza w kontekście genetyki eksperymentalnej, gdzie eksperyment (np. lek, krzyżówka lub gen mówi się, że przeniesienie), skutkujące takim przywróceniem, ratuje normalność fenotyp .
- pozostałość
- Pojedynczy monomer lub podjednostka większej polimerycznej makrocząsteczki; np. kwas nukleinowy składa się z reszt nukleotydowych , a peptyd lub białko składa się z reszt aminokwasowych .
- element odpowiedzi
- Krótka sekwencja DNA w regionie promotora , która jest zdolna do wiązania określonych czynników transkrypcyjnych w celu regulacji transkrypcji określonych genów.
- restytucja
- Spontaniczne ponowne połączenie pękniętego eksperymentalnie chromosomu , które przywraca pierwotną konfigurację.
- jądro Jądro zawierające dwukrotnie większą niż oczekiwana liczbę chromosomów z powodu błędu w podziale komórki, zwłaszcza niezredukowany, diploidalny produkt mejozy , powstały w wyniku niepowodzenia pierwszego lub drugiego podziału mejotycznego.
- restytucyjne
- enzym restrykcyjny
- Enzym endonukleazy lub egzonukleazy , który rozpoznaje i rozszczepia cząsteczkę kwasu nukleinowego na fragmenty w lub w pobliżu określonych rozpoznawanych sekwencji , zwanych miejscami restrykcyjnymi , poprzez zerwanie wiązań fosfodiestrowych szkieletu kwasu nukleinowego . Enzymy restrykcyjne występują naturalnie w wielu organizmach, ale są również rutynowo stosowane do sztucznej modyfikacji DNA w technikach laboratoryjnych, takich jak klonowanie molekularne .
- fragment restrykcyjny
- Dowolny fragment DNA powstały w wyniku przecięcia nici DNA przez enzym restrykcyjny w jednym lub kilku miejscach restrykcyjnych .
- mapa
- restrykcyjna polimorfizmu długości fragmentów restrykcyjnych (RFLP)
- Diagram znanych miejsc restrykcyjnych w znanej sekwencji DNA, takiej jak wektor plazmidowy , uzyskany przez systematyczne wystawianie sekwencji na działanie różnych enzymów restrykcyjnych , a następnie porównywanie długości powstałych fragmentów , technika znana jako mapowanie restrykcyjne . Zobacz także mapę genów .
- miejsce restrykcyjne
- Krótka, specyficzna sekwencja nukleotydów (zwykle o długości od 4 do 8 zasad), która jest niezawodnie rozpoznawana przez określony enzym restrykcyjny . Ponieważ enzymy restrykcyjne zwykle wiążą się jako homodimery , miejsca restrykcyjne są na ogół sekwencjami palindromowymi obejmującymi obie nici dwuniciowej cząsteczki DNA. Endonukleazy restrykcyjne rozszczepiają szkielet fosforanowy między dwoma nukleotydami w samej rozpoznanej sekwencji, ale inne typy enzymów restrykcyjnych dokonują cięć na jednym końcu sekwencji lub w pobliskiej sekwencji.
- odwrotna genetyka
- odwrotna mutacja
- Każda mutacja w genie lub sekwencji DNA, która przywraca lub ratuje pierwotną funkcję lub fenotyp, który został zmieniony lub zniszczony przez poprzednią mutację w tej samej sekwencji. Kontrastowa mutacja do przodu ; patrz także mutacja supresorowa .
- odwrotna transkryptaza (RT)
- Enzym zdolny do syntezy komplementarnej cząsteczki DNA z matrycy RNA , w procesie określanym jako odwrotna transkrypcja .
- odwrotna transkrypcja
- Synteza DNA cząsteczka z matrycy RNA , przeciwieństwo zwykłej transkrypcji . Proces ten, w którym pośredniczy enzym odwrotna transkryptaza , jest wykorzystywany przez wiele wirusów do replikacji ich genomów, a także przez retrotranspozony i komórki eukariotyczne.
- rewertant
- Gen lub allel, w którym występuje mutacja odwrotna , lub organizm niosący taki gen lub allel.
- rybonukleaza (RNaza)
- Dowolna klasa enzymów nukleaz , które katalizują reakcję hydrolityczną rozszczepianie wiązań fosfodiestrowych w cząsteczkach RNA , a tym samym przecinanie nici rybonukleotydów i powodowanie degradacji polimerów RNA na mniejsze składniki . Porównaj dezoksyrybonukleazę .
- kwas rybonukleinowy (RNA)
- Polimeryczna cząsteczka kwasu nukleinowego złożona z szeregu rybonukleotydów zawierających zestaw czterech zasad nukleinowych : adenina ( A ), guanina ( G ), cytozyna ( C ) i uracyl ( U ). W przeciwieństwie do DNA , RNA częściej występuje jako pojedyncza nić zwinięta na siebie, niż jako sparowana nić podwójna. Różne rodzaje cząsteczek RNA pełnią wiele różnych podstawowych ról biologicznych, w tym kodowanie , dekodowanie , regulację i ekspresję genów , a także działają jako cząsteczki sygnałowe, a w niektórych genomach wirusowych jako sam podstawowy materiał genetyczny.
- rybonukleotyd
- Nukleotyd zawierający rybozę jako pentozowy cukier oraz monomeryczną podjednostkę cząsteczek kwasu rybonukleinowego (RNA). Rybonukleotydy kanonicznie zawierają dowolną z czterech zasad azotowych : adeninę ( A ), guaninę ( G ), cytozynę ( C ) i uracyl ( U ). Porównaj dezoksyrybonukleotyd .
- reduktaza rybonukleotydowa (RNR)
- Enzym, który katalizuje tworzenie dezoksyrybonukleotydów poprzez redukcyjną dehydroksylację rybonukleotydów , w szczególności przez usunięcie grupy hydroksylowej 2' z pierścienia rybozy difosforanów rybonukleozydów (rNDP). RNR odgrywa kluczową rolę w regulowaniu ogólnej szybkości syntezy DNA, tak że stosunek DNA do masy komórkowej jest utrzymywany na stałym poziomie podczas podziału komórki i naprawy DNA .
- rybonukleoproteina (RNP)
- Nukleoproteina , która jest kompleksem jednego lub więcej Cząsteczki RNA i jedno lub więcej białek . Przykłady obejmują rybosomy i enzym rybonukleazę P.
- ryboza Cukier
- monosacharydowy , który jako D-ryboza w postaci pierścienia pentozowego jest jednym z trzech podstawowych składników rybonukleotydów, z których zbudowane są cząsteczki kwasu rybonukleinowego (RNA). Ryboza różni się od swojego strukturalnego analogu dezoksyrybozy , stosowanego w DNA , tylko przy węglu 2 ', gdzie ryboza ma przyłączoną grupę hydroksylową, której brakuje w dezoksyrybozie.
- rybosomalny DNA (rDNA)
- rybosomalny RNA (rRNA)
- rybosom
- Kompleks molekularny, który służy jako miejsce syntezy białek . Rybosomy składają się z dwóch podjednostek ( małej podjednostki , która odczytuje informacje zakodowane w cząsteczkach mRNA i dużej podjednostki , która łączy aminokwasy w sekwencji, tworząc łańcuch polipeptydowy ), z których każda składa się z jednej lub więcej nici rybosomalny RNA i różne białka rybosomalne .
- ryboprzełącznik
- Sekwencja regulatorowa w obrębie informacyjnego transkryptu RNA , która może wiązać się z małą cząsteczką efektorową, uniemożliwiając lub przerywając translację , a tym samym działając jako przełącznik regulujący ekspresję mRNA .
- rybozym
- Cząsteczka RNA o aktywności enzymatycznej, tj. zdolna do katalizowania jednej lub więcej określonych reakcji biochemicznych, podobnie jak enzymy białkowe . Rybozymy działają na wiele sposobów, w tym w rybosomach jako część dużej podjednostki rybosomalnego RNA .
- RNA
- Patrz kwas rybonukleinowy .
- Gen Gen , który koduje dowolny z różnych typów niekodującego RNA (np. rRNA i tRNA ).
- RNA
- Interferencja RNA (RNAi)
- Polimeraza RNA
- Dowolna klasa enzymów polimerazy , które syntetyzują cząsteczki RNA z matrycy DNA . Polimerazy RNA są niezbędne do transkrypcji i występują we wszystkich żywych organizmach oraz wielu wirusach. Budują długie, jednoniciowe polimery zwane transkryptami , dodając po jednym rybonukleotydzie w kierunku od 5' do 3' , opierając się na szablonie dostarczonym przez komplementarne nici do wiernej transkrypcji sekwencji nukleotydów.
- Splicing RNA
- Kompleks wyciszający wywołany przez RNA (RISC) Kompleks
- rybonukleoproteinowy , który wycisza endogenne i egzogenne geny poprzez udział w różnych szlakach interferencji RNA na poziomie transkrypcji i translacji. RISC może wiązać zarówno jednoniciowe , jak i dwuniciowe fragmenty RNA, a następnie rozszczepiać je lub wykorzystywać jako wskazówki do docelowych komplementarnych mRNA do degradacji.
- RNaza
- Patrz rybonukleaza .
- Translokacja robertsonowska (ROB)
- Rodzaj translokacji chromosomalnej , w wyniku której pęknięcie podwójnej nici na lub w pobliżu centromerów dwóch chromosomów akrocentrycznych powoduje wzajemną wymianę segmentów, w wyniku której powstaje jeden duży chromosom metacentryczny (składający się z długich ramion ) i jeden niezwykle mały chromosom (składający się z krótkich ramion ), z których ten ostatni jest często później tracony z komórki z niewielkim skutkiem, ponieważ zawiera bardzo mało genów. Wynikowy kariotyp wykazuje o jeden chromosom mniej niż oczekiwana całkowita liczba chromosomów, ponieważ dwa wcześniej odrębne chromosomy zasadniczo się ze sobą zlały. Nosiciele translokacji Robertsona na ogół nie są powiązani z żadnymi nieprawidłowościami fenotypowymi, ale mają zwiększone ryzyko generowania mejotycznie niezrównoważonych gamet .
- replikacja toczącego się koła (RCR)
- rRNA
- Patrz rybosomalny RNA .
- rtPCR
- 1. Skrót od nazwy łańcuchowej reakcji polimerazy w czasie rzeczywistym , synonim ilościowego PCR .
- 2. Skrót od reakcji łańcuchowej polimerazy z odwrotną transkrypcją .

Wzajemna translokacja między chromosomem 4 a chromosomem 20
S
- mutacja tego samego sensu
- Zobacz mutację synonimiczną .
- Sekwencjonowanie Sangera
- Metoda sekwencjonowania DNA oparta na replikacji in vitro matrycowej sekwencji DNA , podczas której dideoksynukleotydy kończące łańcuch, znakowane fluorochromem , są losowo włączane do wydłużającej się nici; powstałe fragmenty są następnie sortowane według wielkości za pomocą elektroforezy , a konkretny fluorochrom kończący każdy z sortowanych według wielkości fragmentów jest wykrywany za pomocą chromatografii laserowej, ujawniając w ten sposób sekwencję oryginalnej matrycy DNA poprzez kolejność znaczników fluorochromowych, czytając od fragmentów o małej wielkości do fragmentów o dużej wielkości. Chociaż sekwencjonowanie Sangera zostało w niektórych kontekstach zastąpione nowej generacji , pozostaje szeroko stosowane ze względu na jego zdolność do generowania stosunkowo długich odczytów sekwencji (ponad 500 nukleotydów ) i bardzo niski poziom błędów.
- hybrydyzacja hybrydyzacji kwasu nukleinowego in vitro , w której jeden składnik polinukleotydowy ( DNA lub RNA ) jest dostarczany w dużym nadmiarze w stosunku do drugiego, powodując, że wszystkie komplementarne sekwencje drugiego polinukleotydu łączą się w pary z nadmiarowymi sekwencjami i tworzą hybrydowe cząsteczki dupleksu .
- nasycona Reakcja
- scaffolding
- scRNA
- Zobacz mały warunkowy RNA .
- selectable marker
- selective sweep
- Proces, w wyniku którego następuje silna selekcja pozytywna nowej i korzystnej mutacji w populacji powoduje, że mutacja osiąga utrwalenie tak szybko, że pobliskie połączone sekwencje DNA również zostają utrwalone poprzez genetyczny autostop , zmniejszając w ten sposób lub eliminując zmienność genetyczną pobliskich loci w populacji.
- samolubny element genetyczny
- Jakikolwiek materiał genetyczny (np. gen lub jakakolwiek inna sekwencja DNA), który może wzmocnić własną replikację i/lub transmisję do kolejnych pokoleń kosztem innych genów w genomie, nawet jeśli nie ma to żadnego pozytywnego skutku lub nawet negatywnego efektu netto na przydatność genomu jako całości. Samolubne elementy zwykle działają poprzez wytwarzanie samoczynnych produktów genów , które umożliwiają im wielokrotne kopiowanie i wklejanie się do innych części genomu, niezależnie od normalnej replikacji DNA (jak w przypadku elementów transpozycyjnych) . ); ułatwiając nierównomierną zamianę segmentów chromosomów podczas rekombinacji genetycznej (jak w przypadku nierównego crossing-over ); lub przez zakłócenie normalnie równej redystrybucji replikowanego materiału podczas mitotis lub mejozy , tak że prawdopodobieństwo, że element samolubny jest obecny w danej komórce potomnej, jest większe niż normalne 50 procent (jak w przypadku napędów genowych ).
- replikacja semikonserwatywna
- Standardowy tryb replikacji DNA występujący we wszystkich żywych komórkach, w którym każda z dwóch nici oryginalnej dwuniciowej cząsteczki DNA są używane jako nici matrycowe , przy czym polimerazy DNA replikują każdą nić osobno i jednocześnie w kierunkach przeciwrównoległych . W rezultacie każda z dwóch dwuniciowych cząsteczek potomnych składa się z jednej z pierwotnych nici macierzystych i jednej nowo zsyntetyzowanej nici komplementarnej, tak że każda cząsteczka potomna zachowuje dokładną sekwencję informacji (w rzeczywistości te same atomy) z jednego połowę oryginalnej cząsteczki. Porównaj konserwatywną replikację i replikacja rozproszona .
- sens
- Rozróżnienie poszczególnych nici dwuniciowej cząsteczki DNA w celu łatwej i specyficznej identyfikacji każdej nici. Dwie komplementarne nici są rozróżniane jako sensowna i antysensowna lub, równoważnie, nić kodująca i nić matrycowa . To właśnie nić antysensowna/matryca jest faktycznie używana jako matryca do transkrypcji ; nić sensowna/kodująca jedynie przypomina sekwencję kodonów na transkrypcie RNA, co umożliwia określenie na podstawie samej sekwencji DNA oczekiwanej sekwencji aminokwasowej dowolnego białka poddanego translacji z transkryptu RNA. Która nić jest która odnosi się tylko do konkretnego transkryptu RNA, a nie do całej cząsteczki DNA; to znaczy, każda nić może funkcjonować jako nić sensowna/kodująca lub antysensowna/matrycowa.
- kodon sensowny
- Dowolny kodon określający aminokwas , w przeciwieństwie do kodonu stop , który nie określa żadnego konkretnego aminokwasu, ale zamiast tego sygnalizuje koniec translacji.
- sekwencja
- Patrz sekwencja kwasu nukleinowego .
- miejsce ze znacznikiem sekwencji (STS) Dowolna
- sekwencja DNA , która występuje dokładnie raz w określonym genomie i której lokalizacja i sekwencja nukleotydów są znane z pewnością.
- chromosom płci
- Zobacz allosome .
- sprzężenie płci
- Sekwencja Shine-Dalgarno
- W wielu prokariotycznych informacyjnych RNA sekwencja konsensusowa AGGAGGU znajduje się 6–8 zasad powyżej kodonu start translacji , który działa jako miejsce wiązania rybosomu poprzez komplementację sekwencji w rybosomalnym RNA .
- krótkie ramię
- W skondensowanych chromosomach , w których położenie centromeru tworzy dwa segmenty lub „ramiona” o różnej długości, krótsze z dwóch ramion chromatydy . Kontrastowe długie ramię .
- krótkie powtórzenie tandemowe (STR)
- Patrz mikrosatelita .
- krótki przeplatany element jądrowy (SINE)
- shotgun sekwencjonowanie wyciszający
- Sekwencja
- lub region DNA, który może być związany przez represor , blokując w ten sposób transkrypcję pobliskiego genu.
- cichy allel
- Allel , który nie wytwarza wykrywalnego produktu . Porównaj allel zerowy .
- mutacja cicha
- Rodzaj mutacji neutralnej , która nie ma zauważalnego wpływu na fenotyp organizmu . Chociaż termin „cicha mutacja” jest często używany zamiennie z mutacją synonimiczną , mutacje synonimiczne nie zawsze są ciche i odwrotnie. Mutacje zmiany sensu , w wyniku których powstaje inny aminokwas ale mutacje o podobnej funkcjonalności (np. leucyna zamiast izoleucyny ) są również często klasyfikowane jako ciche, ponieważ takie mutacje zwykle nie wpływają znacząco na funkcję białka.
- proste powtórzenie sekwencji (SSR)
- Patrz mikrosatelita .
- polimorfizm pojedynczego nukleotydu (SNP)
- Każda substytucja pojedynczego nukleotydu , która występuje w określonej pozycji w genomie i z mierzalną częstotliwością w populacji; na przykład w określonej pozycji zasady w sekwencji DNA większość osobników w populacji może mieć cytozynę ( C ) , podczas gdy u mniejszości osobników ta sama pozycja może być zajęta przez adeninę ( A ). SNP są zwykle definiowane w odniesieniu do „standardowego” genomu odniesienia; pojedynczy ludzki genom różni się od referencyjnego genomu ludzkiego średnio o 4 do 5 milionów pozycji, z których większość składa się z SNP i krótkich indeli . Zobacz także polimorfizm .
- pęknięcie pojedynczej nici (SSB)
- Utrata ciągłości szkieletu fosforanowo-cukrowego w jednej nici dupleksu DNA . Zobacz także nick ; kontrastowe podwójne pęknięcie nici .
- jednoniciowy
- Złożony z pojedynczej, niesparowanej cząsteczki kwasu nukleinowego , tj. jednej liniowej nici nukleotydów o pojedynczym fosfodiestrowym szkielecie , w przeciwieństwie do dupleksu dwóch takich nici połączonych parowaniem zasad. Zobacz też jednoniciowy DNA i jednoniciowy RNA .
- jednoniciowy DNA (ssDNA)
- Dowolna cząsteczka DNA , która składa się z pojedynczego polimeru nukleotydowego lub „nici”, w przeciwieństwie do pary komplementarnych nici połączonych razem wiązaniami wodorowymi ( dwuniciowy DNA ). W większości przypadków DNA jest bardziej stabilny i częściej występuje w formie dwuniciowej, ale wysokie temperatury, niskie stężenia rozpuszczonych soli oraz bardzo wysokie lub niskie pH mogą powodować rozkład cząsteczek dwuniciowych na dwie cząsteczki jednoniciowe w procesie znany jako „topienie”; reakcja ta jest wykorzystywana przez naturalnie występujące enzymy, takie jak te biorące udział w replikacji DNA, jak również przez techniki laboratoryjne, takie jak reakcja łańcuchowa polimerazy .
- siRNA
- Zobacz małe interferujące RNA .
- chromatydy siostrzane
- Para identycznych kopii ( chromatydy ) powstają w wyniku replikacji DNA chromosomu , zwłaszcza gdy obie kopie są połączone wspólnym centromerem ; para siostrzanych chromatyd nazywana jest diadą . Dwie chromatydy siostrzane są ostatecznie rozdzielane od siebie na dwie różne komórki podczas mitozy lub mejozy .
- site-directed mutagenesis
- mały warunkowy RNA (scRNA)
- Klasa małego RNA cząsteczki zaprojektowane tak, aby warunkowo zmieniać konformację w odpowiedzi na pokrewne bodźce molekularne, często w celu kontrolowania szlaków transdukcji sygnału in vitro lub in vivo .
- mały interferujący RNA (siRNA)
- mały jądrowy RNA (snRNA)
- mały jądrowy RNA (snoRNA)
- mały czasowy RNA (stRNA)
- Podklasa mikroRNA , pierwotnie opisana u nicieni , które regulują czas zdarzeń rozwojowych poprzez wiązanie się z komplementarnymi sekwencjami w Nieulegające translacji regiony 3' informacyjnych RNA i hamowanie ich translacji . W przeciwieństwie do siRNA , które służą podobnym celom, strRNA wiążą się z docelowymi mRNA po rozpoczęciu translacji i bez wpływu na stabilność mRNA, co umożliwia docelowym mRNA wznowienie translacji w późniejszym czasie.
- snoRNA
- Patrz małe jąderkowe RNA .
- snRNA
- Zobacz małe jądrowe RNA .
- solenoid fibre
- rozpuszczalny RNA (sRNA)
- Patrz transfer RNA .
- komórka somatyczna
- Każda komórka biologiczna tworząca ciało organizmu lub, w organizmach wielokomórkowych, każda komórka inna niż gameta , komórka rozrodcza lub niezróżnicowana komórka macierzysta . Komórki somatyczne różnią się teoretycznie od komórek linii zarodkowej , co oznacza, że mutacje , którym uległy, nigdy nie mogą zostać przekazane potomkom organizmu, chociaż w praktyce istnieją wyjątki.
- przeniesienie jądra komórki somatycznej (SCNT)
- skrzyżowanie somatyczne
- Patrz rekombinacja mitotyczna .
- Southern blotting
- Metoda biologii molekularnej stosowana do wykrywania określonej sekwencji w próbkach DNA . Technika ta łączy rozdzielanie fragmentów DNA za pomocą elektroforezy żelowej , przeniesienie DNA na syntetyczną membranę, a następnie identyfikację docelowych fragmentów za pomocą znakowanych radioaktywnie lub fluorescencyjnych sond hybrydyzacyjnych .
- odstępnik
- Dowolna sekwencja lub region niekodującego DNA oddzielający sąsiednie geny , transkrybowane lub nie. Termin ten jest używany w szczególności w odniesieniu do regionów niekodujących między wieloma powtarzającymi się kopiami genów rybosomalnego RNA . Zobacz także region międzygenowy .
- przestrzennie ograniczona ekspresja genów
- Ekspresja jednego lub więcej genów tylko w określonym obszarze anatomicznym lub tkance, często w odpowiedzi na parakrynne sygnał. Granica między jurysdykcjami dwóch ograniczonych przestrzennie genów może generować tam ostry gradient fenotypowy , jak w przypadku wzorów paskowych.
- spektralny kariotyp (SKY)
- aparat wrzeciona
- spliceosom
- splicing
- Patrz inżynieria genetyczna .
- ssDNA
- podzielonego genu
- Zobacz jednoniciowy DNA .
- ssRNA
- Patrz jednoniciowy RNA .
- stabilny niescharakteryzowany transkrypt (SUT)
- standardowy kod genetyczny
- Kod genetyczny używany przez ogromną większość organizmów żywych do translacji sekwencji kwasów nukleinowych na białka . W tym systemie z 64 możliwych permutacji trzyliterowych kodonów , które można utworzyć z czterech nukleotydów , 61 koduje jeden z 20 aminokwasów , a pozostałe trzy kodują sygnały stop . Na przykład kodon CAG koduje aminokwas glutaminę , a kodon UAA jest kodonem stop. Standardowy kod genetyczny jest opisany jako zdegenerowane lub zbędne, ponieważ niektóre aminokwasy mogą być kodowane przez więcej niż jeden inny kodon.
- kodon start
- Pierwszy kodon podlegający translacji przez rybosom z transkryptu dojrzałego informacyjnego RNA , używany jako sygnał do zainicjowania syntezy peptydu . W standardowym kodzie genetycznym kodon start zawsze koduje ten sam aminokwas , metioninę , u eukariontów i zmodyfikowaną metioninę u prokariotów. Najpopularniejszym kodonem startowym jest triplet AUG . Kontrastowy kodon stop .
- genetyka statystyczna
- Dział genetyki zajmujący się rozwojem statystycznych metod wyciągania wniosków z danych genetycznych. Teorie i metodologie genetyki statystycznej często wspierają badania z zakresu genetyki ilościowej , epidemiologii genetycznej i bioinformatyki .
- pętelka
- komórka macierzysta
- Każda komórka biologiczna, która nie zróżnicowała się jeszcze w wyspecjalizowany typ komórek i która może dzielić się poprzez mitozę w celu wytworzenia większej liczby komórek macierzystych.
- lepki
- kodon końca stopu
- Kodon , który sygnalizuje zakończenie syntezy białka podczas translacji informacyjnego transkryptu RNA . W standardowym kodzie genetycznym trzy różne kodony stop są używane do oddzielania rybosomów od rosnącego łańcucha aminokwasów, kończąc w ten sposób translację: UAG (nazywany „bursztynem”), UAA („ochra”) i UGA („opal”). Kontrastowy kodon startowy .
- nić
- Pojedynczy łańcuch nukleotydów zawierający polimer kwasu nukleinowego , występujący pojedynczo (w takim przypadku cząsteczka kwasu nukleinowego jest uważana za jednoniciową ) lub sparowany w dupleksie (w którym to przypadku mówi się, że jest dwuniciowy ).
- rygorystyczność
- Wpływ warunków, takich jak temperatura i pH, na stopień komplementarności wymagany do hybrydyzacji reakcja zachodząca między dwiema jednoniciowymi cząsteczkami kwasu nukleinowego. W najbardziej rygorystycznych warunkach tylko dokładne komplementy mogą z powodzeniem hybrydyzować; dwie hybrydyzujące nici mogą tolerować rosnącą liczbę niedopasowań .
- strRNA
- Zobacz mały czasowy RNA .
- strukturalny
- gen Gen kodujący dowolne białko lub produkt RNA inny niż czynnik regulacyjny . Produkty genów strukturalnych obejmują enzymy , białka strukturalne i niektóre niekodujące RNA .
- submetacentryczny (
- chromosomu liniowego lub fragmentu chromosomu) Posiadający centromer umieszczony blisko środka chromosomu, ale nie dokładnie w jego środku, w wyniku czego ramiona chromatyd mają nieco inną długość. Porównaj metacentryczny .
- substytucja
- Rodzaj mutacji punktowej , w której pojedynczy nukleotyd i przyłączona do niego zasada nukleinowa są zastępowane innym nukleotydem.
- tłumienie
- Patrz regulacja w dół .
- punkt skrętu
- synapsis
- kompleks synaptonemalny
- syndeza
- Synapsa chromosomów podczas mejozy .
- synezis
- Agregacja chromosomów w gęsty węzeł przylegający do jednej strony jądra , powszechnie obserwowana podczas leptonemii u niektórych organizmów.
- mutacja synonimiczna
- Rodzaj mutacji , w której zamiana jednej zasady nukleotydowej na inną skutkuje, po transkrypcji i translacji , sekwencją aminokwasową identyczną z oryginalną niezmutowaną sekwencją. Jest to możliwe dzięki degeneracji kodu genetycznego , który pozwala różnym kodonom na kodowanie tego samego aminokwasu. Chociaż synonimiczne mutacje są często uważane za ciche , nie zawsze tak jest; synonimiczna mutacja może wpływać na wydajność lub dokładność transkrypcji , splicingu , translacji lub dowolnego innego procesu, w którym geny są wyrażane , a tym samym stać się skutecznie niemym. Porównaj mutację niesynonimiczną .
- synteny
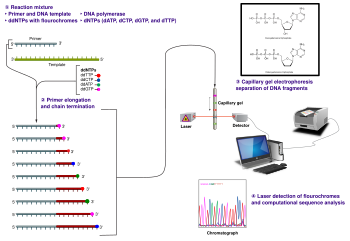
Zarys metody sekwencjonowania Sangera

Trzy różne tryby replikacji DNA . W replikacji semikonserwatywnej każda z dwóch cząsteczek potomnych jest zbudowana z jednej z pierwotnych nici rodzicielskich i jednej nowo zsyntetyzowanej nici. W replikacji konserwatywnej oryginalna cząsteczka macierzysta pozostaje nienaruszona, podczas gdy replikowana cząsteczka składa się z dwóch nowo zsyntetyzowanych nici. W replikacji dyspersyjnej każda z cząsteczek potomnych jest nierówną mieszanką starego i nowego, przy czym niektóre segmenty składają się z dwóch nici rodzicielskich, a inne z dwóch nowo zsyntetyzowanych nici. Tylko replikacja semikonserwatywna zachodzi naturalnie.
Standardowy kod genetyczny określa zestaw 20 różnych aminokwasów z układów trypletowych czterech różnych zasad nukleinowych RNA ( A , G , C i U ). Aby przeczytać ten wykres, wybierz jedną z czterech liter w najbardziej wewnętrznym pierścieniu, a następnie przesuń się na zewnątrz, dodając dwie kolejne litery, aby uzupełnić tryplet kodonów : w sumie można utworzyć 64 unikalne kodony, z których 61 sygnalizuje dodanie jednego z 20 aminokwasów (określonych jednoliterowym skrótem, jak również pełną nazwą i budową chemiczną) do powstającego peptydowego , podczas gdy pozostałe trzy kodony to kodony stop sygnalizujące zakończenie translacji. Wskazano również niektóre właściwości chemiczne aminokwasów i różne sposoby ich modyfikacji.
T
- powtórzenie tandemowe
- Wzór w obrębie sekwencji kwasu nukleinowego , w którym powtarza się jedna lub więcej zasad nukleinowych , a powtórzenia bezpośrednio przylegają do siebie (tj. tandem). Przykładem jest ATGACATGACATGAC , w którym sekwencja ATGAC jest powtarzana trzykrotnie.
- Pudełko TATA
- Wysoce konserwatywna niekodująca sekwencja DNA zawierająca konsensus powtarzających się par zasad T i A , powszechnie spotykana w regionach promotorowych genów u archeonów i eukariontów . Blok TATA często służy jako miejsce inicjacji transkrypcji lub jako miejsce wiązania czynników transkrypcyjnych .
- telestabilność
- Strukturalna destabilizacja podwójnej helisy DNA w locus który jest stosunkowo odległy od miejsca wiązania białka wiążącego DNA .
- telocentryczny (
- chromosomu liniowego lub fragmentu chromosomu) Posiadający centromer umieszczony na końcowym końcu chromosomu (w pobliżu lub w telomerze ), w wyniku czego powstaje tylko jedno ramię. Porównaj akrocentryczny .
- telomer
- Region powtarzających się sekwencji nukleotydów na każdym końcu liniowego chromosomu który chroni koniec chromosomu przed zepsuciem i fuzją z innymi chromosomami. Ponieważ każda runda replikacji powoduje skrócenie chromosomu, telomery działają jak jednorazowe bufory, które są poświęcane na nieustanne obcinanie zamiast pobliskich genów; telomery mogą być również wydłużane przez enzym telomerazę .
- wyciszanie Represja transkrypcji genów w regionach sąsiadujących z telomerami . Wydaje się również, że telomery zmniejszają dostępność subtelomerycznej chromatyny do modyfikacji przez metylotransferazy DNA .
- telomerów
- telofaza
- Końcowy etap podziału komórki zarówno w mitozie , jak i mejozie , występujący po anafazie i przed lub jednocześnie z cytokinezą , podczas którego syntetyzuje się błonę jądrową wokół każdego zestawu chromatyd , jąderka są ponownie składane, a wrzeciono mitotyczne jest rozkładane. Po cytokinezie nowe komórki potomne wznawiają interfazę .
- pasmo szablonowe
- Nić dwuniciowej cząsteczki DNA , która jest używana jako matryca do syntezy RNA podczas transkrypcji . Sekwencja nici matrycy jest komplementarna do powstałego transkryptu RNA. kontrastowa nić kodująca ; zobacz także sens .
- terminalizacja
- W cytologii postępujące przesuwanie się chiasmat z ich pierwotnej pozycji do bardziej dystalnej w miarę postępu mejozy poprzez diplonema i diakinezę .
- kodon terminacji
- Zobacz kodon stop .
- koniec trzech pierwszych
- Zobacz 3'-koniec .
- region nieulegający translacji z trzema liczbami pierwszymi
- Patrz region nieulegający translacji 3' .
- tymidyna ( T , dT )
- Jeden z czterech standardowych nukleozydów stosowanych w cząsteczkach DNA , składający się z zasady tyminy z atomem azotu N9 związanym z atomem węgla C1 cukru dezoksyrybozy . Przedrostek deoksy- jest często pomijany, ponieważ nie ma rybonukleozydowych analogów tymidyny stosowanych w RNA, gdzie zamiast tego zastępuje się ją urydyną .
- tymina ( T )
- Pirymidynowa zasada nukleinowa stosowana jako jedna z czterech standardowych zasad nukleinowych w cząsteczkach DNA . Tymina tworzy parę zasad z adeniną . W RNA tymina w ogóle nie jest używana, zamiast tego jest zastępowana uracylem .
- dimer tyminy
- Zobacz dimer pirymidyny .
- tkankowo specyficzna ekspresja genów
- Funkcja i ekspresja genów ograniczona do określonego rodzaju tkanki lub komórki. Ekspresja specyficzna dla tkanki jest zwykle wynikiem an wzmacniacz , który jest aktywowany tylko w odpowiednim typie komórek.
- topoizomeraza
- Dowolna klasa enzymów wiążących DNA , które katalizują zmiany stanu topologicznego dwuniciowej cząsteczki DNA poprzez nacinanie lub przecinanie szkieletu cukrowo-fosforanowego jednej lub obu nici, rozluźnianie naprężeń skrętnych właściwych dla podwójnej helisy i rozwijanie lub rozplątanie sparowanych pasm przed ponownym ligowaniem nacięć. Ten proces jest zwykle konieczny przed replikacją i transkrypcja . W ten sposób topoizomerazy przekształcają DNA między jego postaciami rozluźnionymi i superskręconymi, połączonymi i niezwiązanymi oraz zawiązanymi i niezwiązanymi, bez zmiany sekwencji lub ogólnego składu chemicznego, tak że cząsteczki substratu i produktu są izomerami strukturalnymi , różniącymi się jedynie kształtem i skręceniem , łączeniem , i/lub wijące się liczby .
- sekwencja końcowa
- Patrz region 3' nie podlegający translacji .
- cecha
- przeł
- Po przeciwnej stronie; naprzeciw; działając z innej cząsteczki. Kontrast cis .
- trans -acting
- Wpływanie na gen lub sekwencję na innej cząsteczce lub nici kwasu nukleinowego . Mówi się, że locus lub sekwencja w określonej cząsteczce DNA, takiej jak chromosom , działa w trans , jeśli ono lub jego produkty wpływają lub działają na inne sekwencje znajdujące się stosunkowo daleko lub na zupełnie innej cząsteczce lub chromosomie. Na przykład białko wiążące DNA działa „w trans ”, jeśli wiąże się lub oddziałuje z sekwencją znajdującą się na dowolnej nici lub cząsteczce innej niż ta, na której jest zakodowana. Kontrast cis-działający .
- trans -splicing
- transkrybowany przerywnik
- Sekwencja przerywnika , która jest transkrybowana i tym samym zawarta w transkrypcie pierwotnego rybosomalnego RNA (w przeciwieństwie do nietranskrybowanego przerywnika ), ale następnie wycinana i odrzucana podczas dojrzewania funkcjonalnych RNA rybosomu .
- transkrypcja
- Produkt z transkrypcja ; to znaczy dowolna RNA , która została zsyntetyzowana przez polimerazę RNA przy użyciu komplementarnej cząsteczki DNA jako matrycy . Kiedy transkrypcja jest zakończona, transkrypty oddzielają się od DNA i stają się niezależnymi transkryptami pierwotnymi . Szczególnie u eukariontów, zwykle konieczne są liczne modyfikacje potranskrypcyjne, aby surowe transkrypty zostały przekształcone w stabilne i trwałe cząsteczki, które są następnie opisywane jako dojrzałe , chociaż nie wszystkie transkrybowane RNA przechodzą dojrzewanie. Wiele transkrypcji jest przypadkowych, fałszywych, niekompletnych lub wadliwych; inne są w stanie pełnić swoje funkcje natychmiast i bez modyfikacji, na przykład niektóre niekodujące RNA .
- transkrypt o nieznanej funkcji (TUF)
- transkryptaza
- Patrz polimeraza RNA .
- transkrypcja
- Pierwszy etap w procesie ekspresji genów , w którym cząsteczka RNA , znana jako transkrypt , jest syntetyzowana przez enzymy zwane Polimerazy RNA wykorzystujące gen lub inną sekwencję DNA jako matrycę . Transkrypcja jest krytycznym i fundamentalnym procesem we wszystkich żywych organizmach i jest niezbędna do wykorzystania informacji zakodowanych w genomie . Wszystkie klasy RNA muszą zostać poddane transkrypcji, zanim będą mogły wywrzeć wpływ na komórkę, chociaż tylko informacyjny RNA (mRNA) musi przejść translację , zanim będzie można wytworzyć funkcjonalne białko , podczas gdy wiele typów niekodujących RNA wypełniać swoje obowiązki bez tłumaczenia. Transkrypcja nie zawsze jest również korzystna dla komórki: gdy zachodzi w niewłaściwym czasie lub w niefunkcjonalnym miejscu , lub gdy ruchome elementy lub zakaźne patogeny wykorzystują maszynerię transkrypcyjną gospodarza, powstałe transkrypty (nie wspominając o marnowaniu cennej energii i zasobów) ) są często szkodliwe dla komórki gospodarza lub genomu.
- czynnik transkrypcyjny (TF)
- Dowolne białko , które kontroluje szybkość transkrypcji informacji genetycznej z DNA na RNA poprzez wiązanie się z określoną sekwencją DNA i promowanie lub blokowanie rekrutacji polimerazy RNA do pobliskich genów . Czynniki transkrypcyjne mogą skutecznie „włączać” i „wyłączać” określone geny, aby upewnić się, że ulegają one ekspresji we właściwym czasie i we właściwych miejscach; z tego powodu są podstawowym i wszechobecnym mechanizmem regulacji genów .
- miejsce startu transkrypcji (TSS)
- Specyficzne miejsce w genie , w którym polimeraza RNA rozpoczyna transkrypcję , określone przez specyficzny nukleotyd lub kodon odpowiadający pierwszemu(-i) rybonukleotydom(-om), który(-e) ma być złożony(-e) w powstającym transkrypcie, tj. kodon start . To miejsce jest zwykle uważane za początek sekwencji kodującej i jest punktem odniesienia do numerowania poszczególnych nukleotydów w genie. Nukleotydom w górę od miejsca startowego przypisuje się liczby ujemne, a nukleotydom w dół są przypisane liczby dodatnie, które są używane do wskazania pozycji pobliskich sekwencji lub struktur względem TSS. Na przykład miejscem wiązania polimerazy RNA może być krótka sekwencja bezpośrednio powyżej TSS, od około -80 do -5, podczas gdy intron w regionie kodującym można zdefiniować jako sekwencję zaczynającą się od nukleotydu +207 i kończącą na nukleotydzie +793.
- jednostka transkrypcyjna
- Segment DNA pomiędzy miejscem inicjacji i terminacji transkrypcji , zawierający sekwencje kodujące dla jednego lub więcej genów . Wszystkie geny w obrębie jednostki transkrypcyjnej są transkrybowane razem w pojedynczy transkrypt podczas pojedynczego zdarzenia transkrypcyjnego; powstały policistronowy RNA może następnie zostać rozszczepiony na oddzielne RNA lub może ulec translacji jako jednostka, a następnie rozszczepiony na oddzielne polipeptydy.
- transkrypcja pękająca
- Przerywany charakter mechanizmów transkrypcji i translacji . Oba procesy zachodzą w „wybuchach” lub „impulsach”, z okresami aktywności genów oddzielonymi nieregularnymi przerwami.
- transkryptom
- Cały zestaw cząsteczek RNA (często odnoszący się do wszystkich rodzajów RNA, ale czasami wyłącznie do informacyjnego RNA ), który jest lub może być wyrażany przez określony genom , komórkę, populację komórek lub gatunek w określonym czasie lub w określonych warunkach. Transkryptom różni się od egzomu i translatomu .
- transdukcja
- Przeniesienie materiału genetycznego między komórkami przez wirusa lub wektor wirusowy, w sposób naturalny lub sztuczny.
- transfekcja
- Celowe eksperymentalne wprowadzenie egzogennych kwasów nukleinowych do komórki lub zarodka. W najszerszym znaczeniu termin ten może odnosić się do dowolnego takiego transferu i czasami jest używany zamiennie z transformacją , chociaż niektóre zastosowania ograniczają użycie transfekcji do wprowadzenia nagiego lub oczyszczonego niewirusowego DNA lub RNA do hodowanych komórek eukariotycznych (zwłaszcza komórek zwierzęcych) skutkujące późniejszym włączeniem obcego DNA do genomu gospodarza lub niedziedziczną modyfikacją ekspresji genów przez obce RNA. W przeciwieństwie zarówno do standardowej transformacji niewirusowej, jak i transdukcji , czasami stosowano transfekcję w odniesieniu do pobierania oczyszczonych wirusowych kwasów nukleinowych przez bakterie lub komórki roślinne bez pomocy wektora wirusowego.
- transfer RNA (tRNA)
- Specjalna klasa cząsteczek RNA , zwykle o długości od 76 do 90 nukleotydów , która służy jako fizyczny adapter umożliwiający translację transkryptów mRNA na sekwencje aminokwasów podczas syntezy białek. Każdy tRNA zawiera specyficzny antykodonu odpowiadający aminokwasowi, który jest kowalencyjnie przyłączony do przeciwległego końca tRNA; w miarę postępu translacji tRNA są rekrutowane do rybosomu , gdzie każdy kodon mRNA jest sparowany z tRNA zawierającym komplementarny antykodon. W zależności od organizmu komórki mogą wykorzystywać aż 41 różnych tRNA z unikalnymi antykodonami; z powodu degeneracji kodonów w kodzie genetycznym kilka tRNA zawierających różne antykodony zawiera ten sam aminokwas.
- transfer-messenger RNA (tmRNA)
- Rodzaj cząsteczki RNA w niektórych bakteriach, która ma podwójne właściwości podobne do tRNA i mRNA , dzięki czemu może jednocześnie pełnić wiele różnych funkcji podczas translacji .
- ligaza tRNA
- Patrz syntetaza aminoacylo-tRNA .
- transformant Komórka lub organizm, który w wyniku
- transformacji pobrał zewnątrzkomórkowy DNA i który może eksprymować kodowane przez niego geny.
- transgen
- transformacyjny
- Dowolny gen lub inny segment materiału genetycznego, który został wyizolowany z jednego organizmu, a następnie przeniesiony w sposób naturalny lub za pomocą dowolnej z różnych technik inżynierii genetycznej do innego organizmu, zwłaszcza innego gatunku. Transgeny są zwykle wprowadzane do linii zarodkowej drugiego organizmu . Są powszechnie używane do badania funkcji genów lub do nadania korzyści, które nie są dostępne w inny sposób w niezmienionym organizmie.
- przejście
- Mutacja punktowa , w której nukleotyd purynowy zostaje zastąpiony inną puryną ( A ↔ G ) lub nukleotyd pirymidynowy zostaje zastąpiony inną pirymidyną ( C ↔ T ). Transwersja kontrastu .
- translacja
- Drugi etap w procesie ekspresji genów , w którym informacyjny RNA Transkrypt powstały podczas transkrypcji jest odczytywany przez rybosom w celu wytworzenia funkcjonalnego białka .
- translatom
- Cały zestaw informacyjnych cząsteczek RNA , które ulegają translacji w określonym genomie , komórce, tkance lub gatunku w określonym czasie lub w określonych warunkach. Podobnie jak transkryptom , jest często używany jako wskaźnik ilościowy do ilościowego określania poziomów ekspresji genów , chociaż transkryptom zawiera również wiele cząsteczek RNA, które nigdy nie ulegają translacji.
- translokacja
- Rodzaj nieprawidłowości chromosomalnej spowodowanej strukturalną rearanżacją dużych fragmentów jednego lub więcej chromosomów . Istnieją dwa główne typy: wzajemny i robertsonowski .
- genetyka transmisyjna
- Dział genetyki zajmujący się badaniem mechanizmów związanych z przenoszeniem genów od rodziców do potomstwa.
- element transpozycyjny (TE)
- transpozaza
- Dowolny z klasy samoczynnych enzymów zdolnych do wiązania się z sekwencjami flankującymi elementu zdolnego do transpozycji , który je koduje, i katalizowania jego przemieszczania się do innej części genomu, zwykle przez mechanizm wycinania/wstawiania lub mechanizm replikacji, w procesie znany jako transpozycja .
- transpozycja
- Proces, w którym sekwencja kwasu nukleinowego znana jako element transpozycyjny zmienia swoją pozycję w genomie poprzez wycięcie i ponowne wstawienie w innym locus (wytnij i wklej) lub powielając się bez przenoszenia elementu z jego pierwotnego miejsca (kopiuj-wklej). Reakcje te są katalizowane przez enzym znany jako transpozaza , który jest kodowany przez gen w samym elemencie zdolnym do transpozycji; w ten sposób produkty elementu są samoczynne i mogą autonomicznie kierować własną reprodukcją. Transponowane sekwencje mogą być ponownie wstawiane w losowych loci lub w miejscach docelowych specyficznych dla sekwencji, albo na tej samej cząsteczce DNA, albo na różnych cząsteczkach.
- transwekcja
- transwersja
- Mutacja punktowa , w której puryna nukleotyd jest podstawiony na nukleotyd pirymidynowy lub odwrotnie (np. A ↔ C lub A ↔ T ). Przejście kontrastu .
- powtórzenie trinukleotydu
- Dowolna sekwencja, w której pojedyncza trójka nukleotydów jest wielokrotnie powtarzana w tandemie , czy to w genie, czy w sekwencji niekodującej. W większości loci pewien stopień powtórzeń jest normalny i nieszkodliwy, ale mutacje, które powodują określone trojaczki (zwłaszcza formy C n G ) w celu zwiększenia liczby kopii powyżej normalnego zakresu są wysoce niestabilne i odpowiedzialne za różne zaburzenia genetyczne .
- tryplet
- Jednostka trzech kolejnych nukleotydów w cząsteczce DNA lub RNA . Trójka w sekwencji kodującej, która koduje określony aminokwas, jest znana jako kodon .
- trisomia
- Rodzaj polisomii , w którym diploidalna komórka lub organizm ma trzy kopie określonego chromosomu zamiast zwykłej dwójki.
- tRNA
- Patrz transfer RNA .
- prawdziwie rasowy
- Wykazujący spójne, przewidywalne i powtarzalne cechy potomstwa, które wynikają z krzyżowania dowolnych dwóch czystorasowych rodziców należących do tej samej rasy lub odmiany (a nie cechy innych ras lub odmian, które mogły wcześniej zostać skrzyżowane w linii ). „Prawdziwa hodowla” oznacza, że rodzice z tej samej linii dadzą potomstwo, które będzie miało wszystkie cechy rodziców.
- przekręcana liczba

Uproszczony schemat transkrypcji . Polimeraza RNA (RNAP) syntetyzuje transkrypt RNA (niebieski) w kierunku od 5' do 3', wykorzystując jedną z nici DNA jako matrycę, podczas gdy kompleks wielu czynników transkrypcyjnych wiąże się z promotorem powyżej genu.
u
- ubikwitynacja
- umbra
- Zobacz opal .
- nienaładowany tRNA
- Przenoszący RNA bez przyłączonego aminokwasu . tRNA naładowane kontrastem .
- underdominance
- underwinding
- Zobacz ujemne superskręcenie .
- nierówne przejście przez
- jednorodzicielskie dziedziczenie
- unikalnego DNA
- Klasa sekwencji DNA określonych przez analizę Ct 0 sekwencji jako obecnych tylko raz w analizowanym genomie, w przeciwieństwie do powtarzalnych . Większość genów strukturalnych i ich intronów jest unikalna.
- mutacja Mutacja o wysokiej częstości rewersji .
- niestabilna
- region nieulegający translacji (UTR)
- Jedna z dwóch sekwencji niekodujących, które są transkrybowane wraz z sekwencją kodującą białko , a zatem zawarte w informacyjnym RNA , ale które ostatecznie nie ulegają translacji podczas syntezy białek. Typowy transkrypt mRNA zawiera jeden taki region bezpośrednio powyżej sekwencji kodującej, znany jako region 5' nie podlegający translacji (5'-UTR) i jeden poniżej sekwencji kodującej, znany jako region 3' nie podlegający translacji (3'-UTR) . Regiony te nie są usuwane podczas przetwarzania potranskrypcyjnego (w przeciwieństwie do intronów ) i są zwykle uważane za wyłączne z czapeczki 5 ' i poliadenylowanego ogona 3' (oba są późniejszymi dodatkami do pierwotnego transkryptu, a nie same są produktami transkrypcji). UTR są konsekwencją faktu, że transkrypcja zwykle zaczyna się znacznie powyżej kodonu start sekwencji kodującej białko i kończy się długo po transkrypcji kodonu stop , podczas gdy translacja jest bardziej precyzyjna.
- regulacja w górę
- Każdy proces, naturalny lub sztuczny, który zwiększa poziom ekspresji określonego genu . Mówi się, że gen, którego ekspresja jest obserwowana na stosunkowo wysokim poziomie (na przykład poprzez wykrycie wyższych poziomów jego mRNA ) w jednej próbce w porównaniu z inną próbką, jest regulowany w górę . Zmniejszenie kontrastu .
- w górę
- W kierunku lub bliżej końca 5' łańcucha nukleotydów lub N -końca łańcucha peptydowego . Kontrast poniżej .
- sekwencja aktywująca w górę (UAS)
- uracyl ( U ) Pirymidynowa jedna z czterech standardowych zasad nukleinowych w cząsteczkach RNA . Uracyl tworzy parę zasad z adeniną . W DNA uracyl w ogóle nie jest używany, a zamiast tego jest zastąpiony tyminą .
- zasada nukleinowa stosowana jako
- urydyna ( U , Urd)
- Jeden z czterech standardowych nukleozydów stosowanych w RNA cząsteczki składające się z zasady uracylu z azotem N9 związanym z atomem węgla C1 cukru rybozy . W DNA urydyna jest zastąpiona tymidyną .
V
- zróżnicowana liczba powtórzeń tandemowych (VNTR)
- variegation
- Zmienność lub nieregularność określonego fenotypu , zwłaszcza rzucająca się w oczy widoczna cecha , taka jak kolor lub pigmentacja, występująca jednocześnie w różnych częściach tego samego pojedynczego organizmu z różnych przyczyn, takich jak X- inaktywacja , rekombinacja mitotyczna , aktywność elementu transpozycyjnego , efekty pozycji lub infekcja patogenami.
- wektor
- variome
- Dowolne DNA cząsteczka używana jako nośnik do sztucznego przenoszenia obcego materiału genetycznego do innej komórki, gdzie może być replikowany i/lub wyrażany . Wektory to zazwyczaj zmodyfikowane rekombinowanego DNA składające się z wstawki (często transgenu ) i dłuższej sekwencji „kręgosłupa” zawierającej początek replikacji , miejsce wielokrotnego klonowania i marker selekcyjny . Wektory są szeroko stosowane w laboratoriach biologii molekularnej do izolacji, klonowania lub wyrazić wstawkę w komórce docelowej.
- komórka roślinna
- Patrz komórka somatyczna .
W
- Sekwencjonowanie całego genomu metodą (WGS)
- western blotting
- Proces określania całości lub prawie całości sekwencji DNA składających się na genom organizmu za pomocą jednej procedury lub eksperymentu, generalnie obejmujący całe chromosomalne i pozachromosomalne (np. mitochondrialne ) DNA.
- typ dziki (WT)
- Fenotyp typowej formy gatunku występującej w przyrodzie; produkt standardowego „normalnego” allelu w danym locus , w przeciwieństwie do produktu wytwarzanego przez niestandardowy zmutowany allel.
- chwiejna liczba
- wijących się par bazowych
X
- Chromosom X
- Jeden z dwóch chromosomów płciowych obecnych w organizmach wykorzystujących system determinacji płci XY i jedyny chromosom płciowy w systemie X0 . Chromosom X występuje zarówno u mężczyzn, jak i kobiet i zwykle zawiera znacznie więcej genów niż jego odpowiednik, chromosom Y.
- Inaktywacja chromosomu X
- Proces, w którym jedna z dwóch kopii chromosomu X zostaje wyciszona poprzez nieodwracalną kondensację w nieaktywną transkrypcyjnie heterochromatynę w komórkach samic ssaków teriańskich. Forma kompensacji dawki , inaktywacja X zapobiega wytwarzaniu przez kobiety dwukrotnie większej liczby produktów genowych z genów na chromosomie X niż mężczyźni, którzy mają tylko jedną kopię chromosomu X. To, który chromosom X jest inaktywowany, jest ustalane losowo we wczesnym zarodku, co umożliwia istnienie linii komórkowych z różnymi nieaktywnymi chromosomami X w tym samym organizmie.
- Cecha sprzężona z X
Y
- Chromosom Y
- Jeden z dwóch chromosomów płciowych obecnych w organizmach wykorzystujących system determinacji płci XY . Chromosom Y występuje tylko u mężczyzn i jest zwykle znacznie mniejszy niż jego odpowiednik, chromosom X.
- Rozwidlenie Y
- Zobacz rozwidlenie replikacyjne .
- sztuczny chromosom drożdży (YAC)
Z
- Zygonema
- palca cynkowego
- Z-DNA
- W mejozie drugi z pięciu podetapów profazy I , następujący po leptonemie i poprzedzający pachynemę . Podczas zygonemy synapsy , która fizycznie wiąże ze sobą homologiczne chromosomy , a centrosom komórki dzieli się na dwa centrosomy potomne, z których każdy zawiera pojedynczą centriolę .
- zygotyczność
- Stopień, w jakim wiele kopii genu , chromosomu lub genomu mają tę samą sekwencję genetyczną; np. w organizmie diploidalnym z dwiema kompletnymi kopiami genomu (jedną matczyną i jedną ojcowską), stopień podobieństwa alleli obecnych w każdej kopii. Mówi się, że osoby posiadające dwa różne allele dla określonego genu są heterozygotami dla tego genu; mówi się, że osobniki posiadające dwa identyczne allele są homozygotyczne pod względem tego genu. Zygotyczność można również rozpatrywać zbiorczo dla grupy genów lub dla całego zestawu genów i loci genetycznych składających się na genom.
- zygota
- Rodzaj eukariota komórka powstała w bezpośrednim wyniku zapłodnienia między dwiema gametami . W organizmach wielokomórkowych zygota jest najwcześniejszym etapem rozwoju.
Zobacz też
- Wprowadzenie do genetyki
- Zarys genetyki
- Zarys biologii komórki
- Słowniczek biologii
- Słowniczek chemii
- Słowniczek biologii ewolucyjnej
Dalsza lektura
- Budd, A. (2012). „Wprowadzenie do biologii genomu: cechy, procesy i struktury”. Genomika ewolucyjna . Metody w biologii molekularnej . Tom. 855. s. 3–4. doi : 10.1007/978-1-61779-582-4_1 . ISBN 978-1-61779-581-7 . PMID 22407704 .