Koronawirus związany z MERS
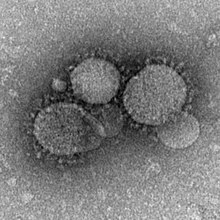
|
|
| koronawirusa MERS-CoV związane z zespołem oddechowym na Bliskim Wschodzie | |
|---|---|
| widziane przez mikroskopię elektronową z barwnikiem ujemnym. Wiriony zawierają charakterystyczne maczugowate wypustki wychodzące z błony wirusowej. | |
| Klasyfikacja wirusów | |
| (nierankingowe): | Wirus |
| królestwo : | Rybowiria |
| Królestwo: | Orthornawirusy |
| Gromada: | Pisuviricota |
| Klasa: | Pisoniviricetes |
| Zamówienie: | Nidowirusy |
| Rodzina: | Koronawirusy |
| Rodzaj: | Betakoronawirus |
| Podrodzaj: | Merbekowirus |
| Gatunek: |
Koronawirus związany z bliskowschodnim zespołem oddechowym
|
Koronawirus związany z zespołem oddechowym na Bliskim Wschodzie ( MERS-CoV ) lub EMC/2012 ( HCoV-EMC/2012 ) to wirus wywołujący zespół oddechowy na Bliskim Wschodzie (MERS). Jest to gatunek koronawirusa , który zaraża ludzi, nietoperze i wielbłądy. Wirus infekujący jest otoczkowym , jednoniciowym wirusem RNA o dodatniej czułości , który dostaje się do komórki gospodarza poprzez wiązanie się z receptorem DPP4 . Gatunek należy do rodzaju Betacoronavirus i podrodzaj Merbecovirus .
Początkowo nazywany po prostu nowym koronawirusem lub nCoV, został po raz pierwszy zgłoszony w czerwcu 2012 roku po sekwencjonowaniu genomu wirusa wyizolowanego z próbek plwociny osoby, która zachorowała w 2012 roku podczas wybuchu nowej grypopodobnej choroby układu oddechowego. Do lipca 2015 r. przypadki MERS-CoV zgłoszono w ponad 21 krajach w Europie , Ameryce Północnej i Azji , a także na Bliskim Wschodzie . MERS-CoV jest jednym z kilku wirusów zidentyfikowanych przez Światową Organizację Zdrowia (WHO) jako prawdopodobną przyczynę przyszłej epidemii. Wymieniają go do pilnych badań i rozwoju.
Wirusologia
Wirus MERS-CoV należy do grupy beta koronawirusa, Betacoronavirus , linia C. Genomy MERS-CoV są filogenetycznie podzielone na dwa klady , klad A i B. Najwcześniejsze przypadki dotyczyły klastrów kladu A, podczas gdy większość bardziej ostatnie przypadki dotyczą genetycznie odrębnego kladu B.
MERS-CoV jest jednym z siedmiu znanych koronawirusów infekujących ludzi, w tym HCoV-229E , HCoV-NL63 , HCoV-OC43 , HCoV-HKU1 , oryginalny SARS-CoV (lub SARS-CoV-1) i SARS-CoV-2 . Jest często określany jako wirus podobny do SARS. Do listopada 2019 roku zgłoszono 2494 przypadków MERS z 858 zgonami, co oznacza śmiertelność powyżej 30%.
Wczesne przypadki i zdarzenie uboczne
Pierwszy potwierdzony przypadek odnotowano w Jeddah w Arabii Saudyjskiej w kwietniu 2012 r. Egipski wirusolog Ali Mohamed Zaki wyizolował i zidentyfikował nieznanego wcześniej koronawirusa z płuc mężczyzny . Następnie Zaki opublikował swoje odkrycia 24 września 2012 r. na poczcie ProMED . Wyizolowane komórki wykazywały efekt cytopatyczny (CPE), w postaci zaokrąglenia i tworzenia syncytii .
Drugi przypadek wykryto we wrześniu 2012 r., kiedy 49-letni mężczyzna mieszkający w Katarze miał podobne objawy grypy. Sekwencja wirusa była prawie identyczna jak w pierwszym przypadku. W listopadzie 2012 roku podobne przypadki pojawiły się w Katarze i Arabii Saudyjskiej. Odnotowano dodatkowe przypadki z powiązanymi zgonami i rozpoczęto szybkie badania i monitorowanie nowego koronawirusa. Nie wiadomo, czy infekcje są wynikiem pojedynczego odzwierzęcego z późniejszym przeniesieniem z człowieka na człowieka, czy też wiele geograficznych miejsc infekcji reprezentuje wiele zdarzeń odzwierzęcych z nieznanego wspólnego źródła. [ potrzebne źródło ]
Badanie przeprowadzone przez Ziada Memisha z Riyadh University i jego współpracowników sugeruje, że wirus pojawił się w okresie między lipcem 2007 a czerwcem 2012, prawdopodobnie z siedmioma oddzielnymi transmisjami odzwierzęcymi. [ potrzebne źródło ] Wśród rezerwuarów zwierzęcych CoV ma dużą różnorodność genetyczną, jednak próbki od pacjentów sugerowały podobny genom, a zatem wspólne źródło, chociaż dane były ograniczone. Na podstawie analizy zegara molekularnego ustalono , że wirusy z EMC/2012 i Anglii/Kataru/2012 datowane są na początek 2011 r., co sugeruje, że te przypadki pochodzą od pojedynczego zdarzenia odzwierzęcego. Okazało się, że MERS-CoV krążył w populacji ludzkiej przez ponad rok bez wykrycia i sugerował niezależną transmisję z nieznanego źródła.
tropizm
U ludzi wirus wykazuje silny tropizm w stosunku do nierzęskowych komórek nabłonka oskrzeli i wykazano, że skutecznie unika wrodzonych odpowiedzi immunologicznych i antagonizuje produkcję interferonu (IFN) w tych komórkach. Ten tropizm jest wyjątkowy, ponieważ większość wirusów układu oddechowego atakuje komórki rzęskowe.
Ze względu na podobieństwo kliniczne między MERS-CoV i SARS-CoV zaproponowano, że mogą wykorzystywać ten sam receptor komórkowy; egzopeptydaza, enzym konwertujący angiotensynę 2 ( ACE2 ). Jednak później odkryto, że neutralizacja ACE2 przez rekombinowane przeciwciała nie zapobiega zakażeniu MERS-CoV. Dalsze badania wykazały, że peptydaza dipeptydylowa 4 ( DPP4 ; znana również jako CD26 ) jest funkcjonalnym receptorem komórkowym dla MERS-CoV. W przeciwieństwie do innych znanych receptorów koronawirusa, aktywność enzymatyczna DPP4 nie jest wymagany do infekcji. Jak można się było spodziewać, sekwencja aminokwasowa DPP4 jest wysoce konserwatywna u różnych gatunków i ulega ekspresji w ludzkim nabłonku oskrzeli i nerkach. Wydaje się, że geny DPP4 nietoperzy podlegały w wysokim stopniu ewolucji adaptacyjnej w odpowiedzi na infekcje koronawirusowe, więc linia prowadząca do MERS-CoV mogła krążyć w populacjach nietoperzy przez długi czas, zanim została przeniesiona na ludzi.
Przenoszenie
W dniu 13 lutego 2013 r. Światowa Organizacja Zdrowia stwierdziła, że „ryzyko trwałej transmisji między osobami wydaje się być bardzo niskie”. Komórki infekowane przez MERS-CoV w płucach stanowią tylko 20% komórek nabłonka oddechowego, więc prawdopodobnie trzeba będzie wdychać dużą liczbę wirionów, aby wywołać infekcję.
Anthony Fauci z National Institutes of Health w Bethesda w stanie Maryland stwierdził, że MERS-CoV „w ogóle nie rozprzestrzenia się w sposób trwały z człowieka na człowieka”, jednocześnie zwracając uwagę na możliwość mutacji wirusa w szczep, który przenosi się z człowieka do osoby. Jednak infekcja pracowników służby zdrowia doprowadziła do obaw związanych z przenoszeniem się wirusa z człowieka na człowieka.
Centra Kontroli i Zapobiegania Chorobom (CDC) wymieniają MERS jako przenoszone z człowieka na człowieka. Twierdzą, że „wykazano, że MERS-CoV rozprzestrzenia się między osobami, które są w bliskim kontakcie. Zaobserwowano również przenoszenie się z zakażonych pacjentów na personel medyczny. Badane są skupiska przypadków w kilku krajach”.
Jednak 28 maja CDC ujawniło, że mężczyzna z Illinois, o którym pierwotnie sądzono, że był pierwszym przypadkiem rozprzestrzeniania się wirusa z człowieka na człowieka (od mężczyzny z Indiany na spotkaniu biznesowym), w rzeczywistości uzyskał wynik negatywny na obecność MERS -CoV. Po przeprowadzeniu dodatkowych i bardziej ostatecznych testów z użyciem testu na przeciwciała neutralizujące, eksperci z CDC doszli do wniosku, że pacjent z Indiany nie rozprzestrzenił wirusa na pacjenta z Illinois. Testy wykazały, że mężczyzna z Illinois nie był wcześniej zakażony. MERS może być bezobjawowy, a wczesne badania wykazały, że do 20% przypadków nie wykazuje oznak aktywnej infekcji, ale we krwi występują przeciwciała MERS-CoV.
Ewolucja
Wygląda na to, że wirus pochodzi od nietoperzy. Sam wirus został wyizolowany z nietoperza. Wirus ten jest blisko spokrewniony z Tylonycteris HKU4 i koronawirusem nietoperza Pipistrellus HKU5 . Dowody serologiczne wskazują, że wirusy te infekują wielbłądy od co najmniej 20 lat. Najnowszy wspólny przodek kilku szczepów ludzkich datuje się na marzec 2012 r. (95% przedział ufności od grudnia 2011 r. do czerwca 2012 r.).
Uważa się, że wirusy były obecne u nietoperzy od jakiegoś czasu i do połowy lat 90. rozprzestrzeniły się na wielbłądy. Wydaje się, że wirusy rozprzestrzeniły się z wielbłądów na ludzi na początku 2010 roku. Pierwotny gatunek żywiciela nietoperza i czas początkowej infekcji u tego gatunku nie zostały jeszcze określone. [ Potrzebne źródło ] Badanie sekwencji 238 izolatów sugeruje, że wirus ten wyewoluował w trzy klady różniące się wykorzystaniem kodonów , żywicielem i rozmieszczeniem geograficznym.
Naturalny zbiornik
Uważa się, że wirus pochodzi od nietoperzy, a jednym z kandydatów jest egipski nietoperz grobowy . Praca epidemiologa Iana Lipkina z Columbia University w Nowym Jorku wykazała, że wirus wyizolowany z nietoperza wydaje się pasować do wirusa występującego u ludzi. Betakoronawirusy 2c wykryto u Nycteris w Ghanie i nietoperzy Pipistrellus w Europie, które są filogenetycznie spokrewnione z wirusem MERS-CoV. Jednak główny naturalny rezerwuar, w którym ludzie zarażają się wirusem, pozostawał nieznany do 9 sierpnia 2013 r., Raport w czasopiśmie The Lancet Infectious Diseases wykazało, że 50 z 50 (100%) surowicy krwi wielbłądów omańskich i 15 ze 105 (14%) wielbłądów hiszpańskich miało przeciwciała swoiste dla białka przeciwko białku MERS-CoV. Surowica krwi europejskich owiec, kóz, bydła i innych wielbłądowatych nie miała takich przeciwciał.
Eurosurveillance badanie seroepidemiologiczne autorstwa RA Perera i in. gdzie zbadali 1343 ludzkie i 625 surowic zwierzęcych, wykazali obfitą obecność przeciwciał specyficznych dla MERS-CoV u 108 ze 110 egipskich jednogarbnych wielbłądów, ale nie u innych zwierząt, takich jak kozy, krowy czy owce w tym regionie. Są to pierwsze i znaczące doniesienia naukowe wskazujące na rolę „wielbłądów jednogarbnych” jako rezerwuaru MERS-CoV. [ potrzebne źródło ]
Badania powiązały wielbłądy , pokazując, że infekcja koronawirusem u cieląt i dorosłych wielbłądów dromaderów jest w 99,9% zgodna z genomami ludzkiego kladu B MERS-CoV. Co najmniej jedna osoba, która zachorowała na MERS, miała kontakt z wielbłądami lub ostatnio piła wielbłądzie mleko . Kraje takie jak Arabia Saudyjska i Zjednoczone Emiraty Arabskie produkują i konsumują duże ilości wielbłądziego mięsa . Istnieje możliwość, że nietoperze afrykańskie lub australijskie niosą wirusa i przenoszą go na wielbłądy. Importowane wielbłądy z tych regionów mogły przenosić wirusa na Bliski Wschód.
W 2013 r. MERS-CoV został zidentyfikowany u trzech członków stada wielbłądów dromaderów trzymanych w stodole w Katarze, co powiązano z dwoma potwierdzonymi przypadkami u ludzi, którzy od tego czasu wyzdrowiali. Obecność MERS-CoV u wielbłądów została potwierdzona przez Narodowy Instytut Zdrowia Publicznego i Środowiska (RIVM) Ministerstwa Zdrowia oraz Centrum Medyczne Erasmusa (WHO Collaborating Centre) w Holandii. Podczas pobierania próbek żaden z wielbłądów nie wykazywał żadnych oznak choroby. Najwyższa Rada Zdrowia Kataru zaleciła w listopadzie 2013 r., aby osoby z chorobami współistniejącymi, takimi jak choroby serca, cukrzyca, choroby nerek, choroby układu oddechowego, osoby z obniżoną odpornością i osoby starsze, unikały bliskiego kontaktu ze zwierzętami podczas odwiedzania gospodarstw i targowisk oraz aby przestrzegaj zasad higieny, takich jak mycie rąk.
Dalsze badanie wielbłądów jednogarbnych z Arabii Saudyjskiej, opublikowane w grudniu 2013 r., ujawniło obecność MERS-CoV u 90% ocenianych wielbłądów jednogarbnych (310), co sugeruje, że wielbłądy jednogarbne nie tylko mogą być głównym rezerwuarem MERS-CoV, ale także zwierzęce źródło MERS.
Zgodnie z aktualizacją podsumowującą MERS-CoV z 27 marca 2014 r., ostatnie badania potwierdzają, że wielbłądy są głównym źródłem infekowania ludzi przez MERS-CoV, podczas gdy nietoperze mogą być ostatecznym rezerwuarem wirusa. Dowody obejmują częstotliwość, z jaką wirus został znaleziony u wielbłądów, na które narażono ludzkie przypadki, poważne dane, które pokazują powszechną transmisję u wielbłądów, oraz podobieństwo CoV wielbłąda do ludzkiego CoV.
W dniu 6 czerwca 2014 r. gazeta Arab News zwróciła uwagę na najnowsze wyniki badań w New England Journal of Medicine, w których 44-letni Saudyjczyk, który trzymał stado dziewięciu wielbłądów, zmarł na MERS w listopadzie 2013 r. Jego przyjaciele powiedzieli, że byli świadkami on nakładał miejscowe lekarstwo na nos jednego ze swoich chorych wielbłądów - cztery z nich podobno cierpiały na wydzielinę z nosa - siedem dni przed tym, jak on sam został dotknięty MERS. Naukowcy zsekwencjonowali wirusa znalezionego w jednym z chorych wielbłądów i wirusa, który zabił człowieka, i odkryli, że ich genomy są identyczne. W tym samym artykule, Arab News poinformował, że na dzień 6 czerwca 2014 r. w Królestwie Arabii Saudyjskiej zgłoszono 689 przypadków MERS, w których zginęły 283 osoby.
Taksonomia
MERS-CoV jest bliżej spokrewniony z koronawirusami nietoperzy HKU4 i HKU5 (linia 2C) niż z SARS-CoV (linia 2B) (2, 9), dzieląc ponad 90% identyczności sekwencji z ich najbliższymi krewnymi, koronawirusami nietoperzy HKU4 i HKU5 i dlatego są uważane za należące do tego samego gatunku przez Międzynarodowy Komitet Taksonomii Wirusów (ICTV). [ potrzebne źródło ]
- Mnemoniczny:
- Identyfikator taksonu:
- Nazwa naukowa: koronawirus zespołu oddechowego na Bliskim Wschodzie
- Nazwa zwyczajowa: MERS-CoV
- Synonim: koronawirus zespołu ostrej niewydolności oddechowej
- Inne nazwy:
- nowy koronawirus (nCoV)
- Londyn1 powieść CoV/2012
- Erasmus Medical Center/2012 dla ludzkiego koronawirusa ( HCoV-EMC/2012 )
- Ranga:
- Rodowód:
-
› Wirusy
- › Wirusy ssRNA
-
› Grupa: IV; jednoniciowe wirusy RNA o dodatniej czułości ›
-
Rząd: Nidovirales
-
› Rodzina: Coronaviridae
-
› Podrodzina: Coronavirinae
-
› Rodzaj: Betacoronavirus
- › Gatunek: Betacoronavirus 1 (powszechnie nazywany ludzkim koronawirusem OC43 ), ludzki koronawirus HKU1 , mysi koronawirus , koronawirus nietoperza Pipistrellus HKU5 , Koronawirus nietoperza Rousettus HKU9 , Koronawirus związany z zespołem ciężkiej ostrej niewydolności oddechowej , Koronawirus nietoperza Tylonycteris HKU4 , MERS-CoV , Koronawirus zespołu ciężkiej ostrej niewydolności oddechowej 2
-
› Rodzaj: Betacoronavirus
-
› Podrodzina: Coronavirinae
-
› Rodzina: Coronaviridae
-
Rząd: Nidovirales
-
› Grupa: IV; jednoniciowe wirusy RNA o dodatniej czułości ›
- › Wirusy ssRNA
- Nosiciele wirusów:
- Homo sapiens (człowiek)
- wielbłądy
- nietoperze
Szczepy:
- Izolować:
- Izolować:
- NCBI
Badania i patenty
Urzędnicy saudyjscy nie wyrazili zgody, aby dr Zaki, pierwszy izolator ludzkiego szczepu, wysłał próbkę wirusa do Fouchier i byli rozgniewani, gdy Fouchier zażądał patentu na pełną sekwencję genetyczną MERS-CoV.
Redaktor The Economist zauważył: „Niepokój o bezpieczeństwo nie może spowalniać pilnej pracy. Badanie śmiercionośnego wirusa jest ryzykowne. Brak badania jest bardziej ryzykowny”. Dr Zaki został zwolniony z pracy w szpitalu w wyniku pominięcia saudyjskiego Ministerstwa Zdrowia w swoim ogłoszeniu i udostępnienia swojej próbki i wyników.
Na dorocznym spotkaniu Światowego Zgromadzenia Zdrowia w maju 2013 r. szefowa WHO Margaret Chan zadeklarowała tę własność intelektualną , czy patenty na szczepy nowego wirusa, nie powinny utrudniać narodom ochrony swoich obywateli poprzez ograniczanie badań naukowych. Wiceminister zdrowia Ziad Memish wyraził obawy, że naukowcy posiadający patent na MERS-CoV nie pozwolą innym naukowcom na wykorzystanie opatentowanego materiału i dlatego opóźniają rozwój testów diagnostycznych. Erasmus MC odpowiedział, że wniosek patentowy nie ogranicza badań zdrowia publicznego nad MERS i MERS-CoV oraz że wirusy i testy diagnostyczne zostały wysłane – bezpłatnie – do wszystkich, którzy zażądali takich odczynników.
Mapowanie
Istnieje szereg wysiłków związanych z mapowaniem, które koncentrują się na śledzeniu koronawirusa MERS. W dniu 2 maja 2014 r. Uruchomiono mapę Corona, aby śledzić koronawirusa MERS w czasie rzeczywistym na mapie świata. Dane są oficjalnie przekazywane przez WHO lub Ministerstwo Zdrowia danego kraju. HealthMap śledzi również raporty przypadków z uwzględnieniem wiadomości i mediów społecznościowych jako źródeł danych w ramach HealthMap MERS. Korea Południowa została zarażona w połowie 2015 roku, z 38 zgonami wśród 186 przypadków infekcji. [ potrzebne źródło ]
Zobacz też
- Europejskie Centrum ds. Zapobiegania i Kontroli Chorób
- Nowy wirus
- Superrozrzutnik
- Zjadliwość
- Koalicja na rzecz innowacji w zakresie gotowości na wypadek epidemii
Linki zewnętrzne
- Pojawienie się koronawirusa bliskowschodniego zespołu oddechowego
- Kompletny genom MERS-CoV
- Pojawiające się wirusy
- Molekularna ilustracja MERS-koronawirusa
- Filipiny nadal wolne od MERS-CoV – DOH
- Śmiertelny koronawirus z Bliskiego Wschodu znaleziony w egipskim nietoperzu grobowym

