Haplogrupa A-L1085
| Haplogrupa A-L1085 | |
|---|---|
| Możliwy czas pochodzenia | 140 000 YBP, 125 000 - 382 000 YBP |
| Możliwe miejsce pochodzenia | Afryka Środkowa - Północno-Zachodnia |
| Przodek | Homo Y-MRCA |
| Potomków | A-V148 (A0), A-P305 (A1) |
| Najwyższe częstotliwości |
Namibia (Tsumkwe San, Nama ) 60-70% Sudan Południowy ( Dinka , Shilluk , Nuer ) 33%-61,5% Etiopia ( Beta Izrael ) 41%-46% |
Haplogrupa A-L1085 , znana również jako haplogrupa A0-T, jest haplogrupą ludzkiego Y-DNA . Jest częścią linii ojcowskiej prawie wszystkich żyjących obecnie ludzi. SNP L1085 odegrał dwie role w genetyce populacji : po pierwsze, większość haplogrup Y-DNA oddzieliła się od niego i; po drugie, definiuje niezróżnicowany klad podstawowy A-L1085 *.
A0-T ma dwie główne gałęzie: A-V148 (znana również jako haplogrupa A0) i haplogrupa A-P305 (haplogrupa A1).
Pochodzenie

Wiele propozycji dotyczących pochodzenia haplogrupy A-L1085 sugeruje, że była ona związana z przodkami populacji łowców-zbieraczy z Afryki Południowej. Dzieje się tak, ponieważ rodowody haplogrupy A-L1085 są częste wśród ludu San .
Jednak linie rodowe A-L1085 z Afryki Południowej są podkladami linii A występujących w innych częściach Afryki, głównie wśród ludów nilotycznych , ale także wśród innych Afrykanów. Sugeruje to, że linie rodowe A-L1085 przybyły do Afryki Południowej z innych miejsc. Dwie najbardziej podstawowe linie haplogrupy A-L1085, A-V148 i A-P305 wykryto w Afryce Zachodniej, Afryce Północno-Zachodniej i Afryce Środkowej. Crucianiego i in. 2011 sugeruje, że te linie mogły pojawić się gdzieś pomiędzy Afryką Środkową i Północno-Zachodnią, chociaż taka interpretacja jest nadal wstępna ze względu na niepełny zasięg geograficzny afrykańskich chromosomów Y.
Wstępne badania wykazały, że linie haplogrupy A-L1085 pojawiły się około 60 000 lat temu, co było znacznie nowsze niż TMRCA w przypadku linii mitochondrialnego DNA, które łączą się do 150-200 kya. Crucianiego i in. Rok 2011 wraz z poważną restrukturyzacją gałęzi cofnął korzeń drzewa chromosomu Y do 142 000 lat temu.
W listopadzie 2012 r. nowe badanie przeprowadzone przez Scozzari i in. potwierdził „hipotezę pochodzenia haplogrupy A1b w północno-zachodniej ćwiartce kontynentu afrykańskiego i wraz z niedawnymi odkryciami starożytnych linii chromosomu Y w środkowo-zachodniej Afryce dostarcza nowych dowodów dotyczących geograficznego pochodzenia ludzkiego MSY różnorodność".
Rozkład geograficzny
Afryka Centralna
Haplogrupa A-M13 została zaobserwowana w populacjach północnego Kamerunu (2/9 = 22% Tupuri , 4/28 = 14% Mandara, 2/17 = 12% Fulbe ) i wschodniej DRK (2/9 = 22% Alur , 1 /18 = 6% Hema , 1/47 = 2% Mbuti ).
Haplogrupę A-M91 (xA-M31, A-M6, A-M32) zaobserwowano u ludu Bakola w południowym Kamerunie (3/33 = 9%).
Bez testowania jakiejkolwiek podklady, haplogrupę A-L1085 zaobserwowano w próbkach kilku populacji Gabonu , w tym 9% (3/33) próbki Baka , 3% (1/36) próbki Ndumu, 2% (1/46) próbki Dumy , 2% (1/57) próbki Nzebi i 2% (1/60) próbki Tsogo .
Wschodnia Afryka
Haplogrupa A-M13 jest powszechna wśród Południowych Sudańczyków (53%), zwłaszcza Dinka (61,5%). Haplogrupę A-M13 zaobserwowano również w innej próbce populacji Sudanu Południowego z częstotliwością 45% (18/40), w tym 1/40 A-M171. Haplogrupa A została również zgłoszona w 14,6% (7/48) próbki Amhara , 10,3% (8/78) próbki Oromo , 13,6% (12/88) innej próbki z Etiopii i 41% próbki z Beta Israel (Cruciani i in. 2002), a także Bantus w Kenii (14%, Luis i in. 2004) oraz Iraku w Tanzanii (3/43 = 7,0% (Luis et al. 2004) do 1/6 = 17% (Knight et al. 2003)).
północna Afryka
Podkladę A1 zaobserwowano u libijskich Berberów , podczas gdy podkladę A-M13 zaobserwowano u około 3% egipskich mężczyzn.
Południowa Afryka
Jedno z badań wykazało haplogrupę A w próbkach różnych grup mówiących po khoisan z częstotliwością w zakresie od 10% do 70%. Ta konkretna haplogrupa nie została znaleziona w próbce Hadzabe z Tanzanii, populacji czasami grupowanej z innymi grupami Khoisan ze względu na obecność spółgłosek kliknięcia w ich języku, ale której język został dokładnie wykazany jako izolat tak niezwiązany z nimi jak inne języków dzięki pracy lingwistki Bonny Sands.
Europa
Haplogrupę A zaobserwowano jako A1 u europejskich mężczyzn w Anglii. Chromosom AY obserwowano również z niską częstotliwością w Azji Mniejszej, na Bliskim Wschodzie i na niektórych wyspach Morza Śródziemnego, wśród Greków Morza Egejskiego, Sycylijczyków (0,2% A1a w Capo d'Orlando i 0,5% A1b na całej wyspie) , Palestyńczyków , Jordańczycy i Jemeńczycy. Bez testowania jakiejkolwiek podklady, haplogrupę A1b zaobserwowano w próbce Greków z Mitilini na wyspie Lesbos na Morzu Egejskim , a A1b zaobserwowano również u 0,1% iberyjskich Żydów . Autorzy jednego z badań zgłosili znalezienie czegoś, co wydaje się być haplogrupą A u 3,1% (2/65) próby Cypryjczyków , chociaż nie wykluczyli ostatecznie możliwości, że którakolwiek z tych osób może należeć do haplogrupy B.
Dystrybucja podkladów
A-V148* (A0)*)
A-V148 jest jedną z dwóch głównych gałęzi w A0-T.
A-P305* (A1*)
Haplogrupa A-P305* jest w dużej mierze ograniczona do części Afryki , choć kilka przypadków odnotowano w Europie i Azji Zachodniej .
A-P305 występuje w najwyższych ilościach u Pigmejów Bakola ( Kamerun Południowy ) na poziomie 8,3% i Berberów z Algierii na poziomie 1,5% oraz w Ghanie . Klad osiąga również wysokie częstotliwości w łowców-zbieraczy Buszmenów w Afryce Południowej , a tuż za nim wiele grup nilotycznych w Afryce Wschodniej . Jednak najstarsze podklady haplogrupy A występują wyłącznie w środkowo - północno- zachodniej Afryce , gdzie uważa się, że on, a co za tym idzie Adam z chromosomem Y, powstał około 140 000 lat temu. Klad obserwowano również ze znaczną częstotliwością w niektórych populacjach w Etiopii , a także w niektórych grupach Pigmejów w Afryce Środkowej.
Haplogrupa A-L1085 jest mniej powszechna wśród osób mówiących w Nigrze i Kongo , którzy w dużej mierze należą do kladu E1b1a . Ogólnie uważa się, że haplogrupa E pochodzi z Afryki północno-wschodniej, a później została wprowadzona do Afryki Zachodniej , skąd rozprzestrzeniła się około 5000 lat temu do Afryki Środkowej, Południowej i Południowo-Wschodniej wraz z ekspansją Bantu . Według Wooda i in. (2005) oraz Rosa i in. (2007), takie stosunkowo niedawne ruchy populacji z Afryki Zachodniej zmieniły istniejącą wcześniej różnorodność chromosomów Y populacji w Afryce Środkowej, Południowej i Południowo-Wschodniej, zastępując poprzednie haplogrupy na tych obszarach dominującymi obecnie liniami E1b1a. Ślady przodków można jednak dziś zaobserwować w tych regionach dzięki obecności haplogrup DNA Y A-M91 i B-M60 , które są powszechne w niektórych populacjach reliktowych, takich jak Pigmeje Mbuti i Khoisan .
| Afryka | ||
| Badana populacja |
Częstotliwość (W %) |
|
| Tsumkwe San (Namibia) | 66% | |
| Nama (Namibia) | 64 | |
| Dinka (Sudan) | 62 | |
| Shilluk (Sudan) | 53 | |
| Nuba (Sudan) | 46 | |
| Khoisan | 44 | |
| Żydzi etiopscy | 41 | |
| !Kung /Sekele | ~40 | |
| Borgu (Sudan) | 35 | |
| Nuer (Sudan) | 33 | |
| Futro (Sudan) | 31 | |
| Masajowie (Kenia) | 27 | |
| Nara (Erytrea) | 20 | |
| Masalit (Sudan) | 19 | |
| Amhara (Etiopia) | ~16 | |
| Etiopczycy | 14 | |
| Bantu (Kenia) | 14 | |
| Mandara (Kamerun) | 14 | |
| Hausa (Sudan) | 13 | |
| Khwe (Republika Południowej Afryki) | 12 | |
| Fulbe (Kamerun) | 12 | |
| Dama (Namibia) | 11 | |
| Oromo (Etiopia) | 10 | |
| Kunama (Erytrea) | 10 | |
| Południowo-semicki (Etiopia) | 10 | |
| Arabowie (Egipt) | 3 | |
W złożonej próbie 3551 afrykańskich mężczyzn haplogrupa A miała częstość 5,4%. Najwyższe częstotliwości haplogrupy A odnotowano wśród Khoisan z Afryki Południowej, Beta Izraela i Nilo-Saharyjczyków .
A-M31
Subklada A-M31 została znaleziona w około 2,8% (8/282) z puli siedmiu próbek różnych grup etnicznych w Gwinei Bissau , zwłaszcza wśród Papel-Manjaco-Mancanha (5/64 = 7,8%). Wcześniejsze badanie, Gonçalves i in. 2003, donieśli o znalezieniu A-M31 w 5,1% (14/276) próbki z Gwinei Bissau iw 0,5% (1/201) pary próbek z Cabo Verde . Autorzy innego badania poinformowali o znalezieniu haplogrupy A-M31 u 5% (2/39) próbki Mandinki z Senegambii i 2% (1/55) próbki Dogonów z Mali . Haplogrupa A-M31 została również znaleziona w 3% (2/64) próby Berberów z Maroka i 2,3% (1/44) próby nieokreślonej przynależności etnicznej z Mali .
Co najmniej siedmiu mężczyzn o przodkach pochodzących z Yorkshire w Anglii i mających charakterystyczne nazwisko Revis zostało zidentyfikowanych jako należących do podklady A-M31. Doniesienia prasowe sugerowały, że mężczyźni byli fenotypowo „europejscy” i nieświadomi żadnego afrykańskiego pochodzenia. Późniejsze badania sugerowały, że mieli wspólnego patrylinearnego przodka w XVIII wieku.
A-M6
A-M6 (dawniej A2) zwykle występuje wśród ludów Khoisan. Autorzy jednego badania zgłosili znalezienie haplogrupy A-M6 (xA-P28) u 28% (8/29) próbki Tsumkwe San i 16% (5/32) próbki! Kung / Sekele oraz haplogrupy A-P28 w 17% (5/29) próbki Tsumkwe San, 9% (3/32) próbki !Kung /Sekele, 9% (1/11) próbki Nama i 6% (1/18) próbki Dama . Autorzy innego badania zgłosili znalezienie haplogrupy A-M6 u 15,4% (6/39) próbki samców Khoisan, w tym 5/39 A-M6 (xA-M114) i 1/39 A-M114.
A-M32
Klad A-M32 (dawniej A3) zawiera najbardziej zaludnione gałęzie haplogrupy A-L1085 i występuje głównie w Afryce Wschodniej i Afryce Południowej .
A-M28
Podklada A-M28 (dawniej A3a) była rzadko obserwowana w Rogu Afryki . U 5% (1/20) mieszanej próby osób posługujących się językami południowo-semickimi z Etiopii, 1,1% (1/88) próby Etiopczyków i 0,5% (1/201) Somalijczyków.
A-M51
Podklada A-M51 (dawniej A3b1) występuje najczęściej wśród ludów Khoisan (6/11 = 55% Nama , 11/39 = 28% Khoisan, 7/32 = 22% ! Kung / Sekele, 6/29 = 21% Tsumkwe San, 1/18 = 6% Damy). Jednak stwierdzono go również z mniejszą częstotliwością wśród ludów Bantu w Afryce Południowej , w tym 2/28 = 7% Sotho-Tswana , 3/53 = 6% mieszkańców Afryki Południowej niebędących Khoisan, 4/80 = 5% Xhosa i 1 /29 = 3% Zulu .
A-M13
Podklada A-M13 (dawniej A3b2), która jest powszechnie spotykana w Afryce Wschodniej i północnym Kamerunie, różni się od tej znalezionej w próbkach Khoisan i jest z nimi tylko zdalnie spokrewniona. To odkrycie sugeruje starożytną rozbieżność.
W Sudanie haplogrupa A-M13 została znaleziona w 28/53 = 52,8% Sudańczyków Południowych , 13/28 = 46,4% Nuba w środkowym Sudanie, 25/90 = 27,8% Sudańczyków Zachodnich , 4/32 = 12,5% miejscowej ludności Hausa i 5/216 = 2,3% mieszkańców Sudanu Północnego.
W Etiopii jedno badanie wykazało znalezienie haplogrupy A-M13 w 14,6% (7/48) próbki Amhary i 10,3% (8/78) próbki Oromo . Inne badanie wykazało znalezienie haplogrupy A-M118 u 6,8% (6/88) i haplogrupy A-M13 (xA-M171, A-M118) u 5,7% (5/88) mieszanej próby Etiopczyków, co stanowi łącznie 12,5% (11/88) A-M13.
Haplogrupa A-M13 była również czasami obserwowana poza Afryką Środkową i Wschodnią, jak w regionie Morza Egejskiego w Turcji (2/30 = 6,7%), jemeńscy Żydzi (1/20 = 5%), Egipt (4/147 = 2,7 %, 3/92 = 3,3%), Palestyńczycy (2/143 = 1,4%), Sardynia (1/77 = 1,3%, 1/22 = 4,5%), stolica Jordanii , Amman (1/101=1 %) i Omanu (1/121 = 0,8%).
Filogenetyka
Historia filogenetyczna
Przed 2002 rokiem w literaturze akademickiej istniało co najmniej siedem systemów nazewnictwa drzewa filogenetycznego chromosomu Y. Doprowadziło to do sporego zamieszania. W 2002 roku główne grupy badawcze połączyły siły i utworzyły Konsorcjum Y-Chromosom Consortium (YCC). Opublikowali wspólny dokument, który stworzył jedno nowe drzewo, którego wszyscy zgodzili się użyć. Później grupa naukowców-obywateli zainteresowanych genetyką populacyjną i genealogią genetyczną utworzyła grupę roboczą w celu stworzenia amatorskiego drzewa, którego celem jest przede wszystkim aktualność. Poniższa tabela zawiera wszystkie te prace w miejscu charakterystycznego drzewa YCC z 2002 roku. Pozwala to badaczowi przeglądającemu starszą opublikowaną literaturę na szybkie poruszanie się między nomenklaturami.
| YCC 2002/2008 (stenografia) | (α) | (β) | (γ) | (δ) | (ε) | (ζ) | (η) | YCC 2002 (odręczny) | YCC 2005 (odręczny) | YCC 2008 (odręczny) | YCC 2010r (odręczny) | ISOGG 2006 | ISOGG 2007 | ISOGG 2008 | ISOGG 2009 | ISOGG 2010 | ISOGG 2011 | ISOGG 2012 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A-M31 | 7 | I | 1A | 1 | – | H1 | A | A1 | A1 | A1 | A1a | A1 | A1 | A1a | A1a | A1a | A1a | A1a |
| A-M6 | 27 | I | 2 | 3 | – | H1 | A | A2* | A2 | A2 | A2 | A2 | A2 | A2 | A2 | A2 | A2 | A1b1a1a |
| A-M114 | 27 | I | 2 | 3 | – | H1 | A | A2a | A2a | A2a | A2a | A2a | A2a | A2a | A2a | A2a | A2a | A1b1a1a1a |
| A-P28 | 27 | I | 2 | 4 | – | H1 | A | A2b | A2b | A2b | A2b | A2b | A2b | A2b | A2b | A2b | A2b | A1b1a1a1b |
| A-M32 | * | * | * | * | * | * | * | * | A3 | A3 | A3 | A3 | A3 | A3 | A3 | A3 | A3 | A1b1b |
| A-M28 | 7 | I | 1A | 1 | – | H1 | A | A3a | A3a | A3a | A3a | A3a | A3a | A3a | A3a | A3a | A3a | A1b1b1 |
| A-M51 | 7 | I | 1A | 1 | – | H1 | A | A3b1 | A3b1 | A3b1 | A3b1 | A3b1 | A3b1 | A3b1 | A3b1 | A3b1 | A3b1 | A1b1b2a |
| A-M13 | 7 | I | 1A | 2 | ue1 | H1 | A | A3b2* | A3b2 | A3b2 | A3b2 | A3b2 | A3b2 | A3b2 | A3b2 | A3b2 | A3b2 | A1b1b2b |
| A-M171 | 7 | I | 1A | 2 | ue1 | H1 | A | A3b2a | A3b2a | A3b2a | A3b2a | A3b2a | A3b2a | A3b2a | A3b2a | A3b2a | A3b2a | REMOVED |
| A-M118 | 7 | I | 1A | 2 | ue1 | H1 | A | A3b2b | A3b2b | A3b2b | A3b2b | A3b2b | A3b2b | A3b2b | A3b2b | A3b2b | A3b2b | A1b1b2b1 |
Oryginalne publikacje naukowe
Następujące zespoły badawcze zgodnie z ich publikacjami były reprezentowane w tworzeniu Drzewa YCC.
Crucianiego 2011
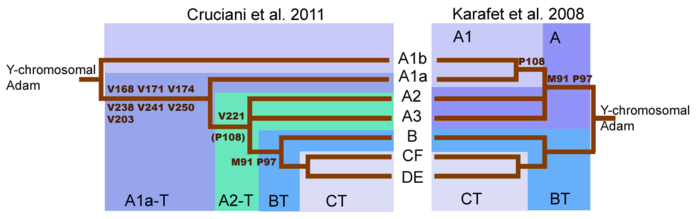
Główna zmiana w rozumieniu drzewa Haplogrupy A pojawiła się wraz z publikacją ( Cruciani 2011 ) . Wstępne sekwencjonowanie ludzkiego chromosomu Y sugerowało, że pierwszy podział w drzewie genealogicznym chromosomu Y nastąpił z mutacją M91, która oddzieliła haplogrupę A od haplogrupy BT . Jednak obecnie wiadomo, że najgłębszy podział w drzewie chromosomu Y występuje między dwiema wcześniej zgłoszonymi podkladami haplogrupy A, a nie między haplogrupą A i haplogrupą BT. Podklady A1b i A1a-T schodzą teraz bezpośrednio z korzenia drzewa. Przegrupowanie drzewa genealogicznego chromosomu Y oznacza, że linie rodowe sklasyfikowane jako haplogrupa A niekoniecznie tworzą klad monofiletyczny . Haplogrupa A odnosi się zatem do zbioru linii, które nie posiadają markerów definiujących haplogrupę BT, chociaż wiele linii w ramach haplogrupy A jest tylko bardzo daleko spokrewnionych.
Mutacje M91 i P97 odróżniają haplogrupę A od haplogrupy BT . W chromosomach haplogrupy A marker M91 składa się z odcinka 8 T jednostek nukleozasadowych. W haplogrupie BT i chromosomach szympansa marker ten składa się z 9 jednostek zasad nukleinowych T. Ten wzorzec sugerował, że odcinek 9T haplogrupy BT był wersją przodków i że haplogrupa A została utworzona przez usunięcie jednej zasady nukleinowej .
Ale według Crucianiego i in. 2011 region otaczający marker M91 jest mutacyjnym hotspotem podatnym na powtarzające się mutacje. Jest zatem możliwe, że odcinek 8T haplogrupy A może być stanem przodków M91, a odcinek 9T haplogrupy BT może być stanem pochodnym, który powstał przez wstawienie 1T. To wyjaśniałoby, dlaczego podklady A1b i A1a-T, najgłębsze gałęzie haplogrupy A, mają odcinek 8T. Ponadto Cruciani i in. 2011 ustalił, że marker P97, który jest również używany do identyfikacji haplogrupy A, posiadał stan przodków w haplogrupie A, ale stan pochodny w haplogrupie BT .
Drzewa filogenetyczne
To drzewo filogenetyczne podkladów haplogrup jest oparte na drzewie Y-Chromosom Consortium (YCC), drzewie haplogrup ISOGG Y-DNA i późniejszych opublikowanych badaniach.
Adam z chromosomem Y
- A0 (dawniej A1b) (P305, V148, V149, V154, V164, V166, V172, V173, V177, V190, V196, V223, V225, V229, V233, V239)
-
A1 (A1a-T wg Cruciani 2011) (L985, L989, L990, L1002, L1003, L1004, L1009, L1013, L1053, V161, V168, V171, V174, V203, V238, V241, V250, V238, V241, V 250 )
- A1a (M31, P82, V4, V14, V15, V25, V26, V28, V30, V40, V48, V53, V57, V58, V63, V76, V191, V201, V204, V214, V215, V236)
-
A1b (A2-T według Crucianiego 2011) (P108, V221)
-
A1b1 (L419)
-
A1b1a (V50, V82, V198, V224)
-
A1b1a1 dawniej A2 (M14, M23, L968/M29/P3/PN3, M71, M135, M141, M206, M276/P247, M277/P248, MEH1, P4, P5, P36.1, strona 71, strona 87, strona 95)
-
A1b1a1a (M6, M196)
-
A1b1a1a1 (M212)
- A1b1a1a1a dawniej A2a (M114)
- A1b1a1a1b dawniej A2b (P28)
- A1b1a1a1c dawniej A2c (P262)
-
A1b1a1a1 (M212)
-
A1b1a1a (M6, M196)
-
A1b1a1 dawniej A2 (M14, M23, L968/M29/P3/PN3, M71, M135, M141, M206, M276/P247, M277/P248, MEH1, P4, P5, P36.1, strona 71, strona 87, strona 95)
-
A1b1b dawniej A3 (M32)
- A1b1b1 dawniej A3a (M28, M59)
-
A1b1b2 dawniej A3b (M144, M190, M220, P289)
-
A1b1b2a dawniej A3b1 (M51, P100, P291)
- A1b1b2a1 dawniej A3b1a (P71, P102)
-
A1b1b2b dawniej A3b2 (M13, M127, M202, M219, M305):
- A1b1b2b1 (M118)
-
A1b1b2a dawniej A3b1 (M51, P100, P291)
-
A1b1a (V50, V82, V198, V224)
- BT (M42, M94, M139, M299, M60, M181/strona 32, P85, P90, P97, strona 65.1/SRY1532.1/SRY10831.1, V21, V29, V31, V59, V64, V102, V187, V202, V216, V235)
-
A1b1 (L419)
Zobacz też
Genetyka
Podklady Y-DNA A
- A-M114
- A-M118
- A-M13
- A-M171
- A-M28
- A-M31
- A-M32
- A-M51
- A-M6
- A-P28
Drzewo szkieletowe Y-DNA
Źródła tabel konwersji
- Capelli, Krystian; Wilson, James F.; Richards, Martin; Stumpf, Michael PH; i in. (luty 2001). „Przeważnie rdzenne dziedzictwo ojcowskie ludów mówiących po austronezyjsku w wyspiarskiej Azji Południowo-Wschodniej i Oceanii” . American Journal of Human Genetics . 68 (2): 432–443. doi : 10.1086/318205 .
- Młotek, Michael F.; Karafet, Tatiana M.; Redd, Alan J.; Jarjanazi, Hamdi; i in. (1 lipca 2001). „Hierarchiczne wzorce globalnej różnorodności ludzkich chromosomów Y” . Biologia molekularna i ewolucja . 18 (7): 1189–1203. doi : 10.1093/oxfordjournals.molbev.a003906 .
- Jobling, Mark A.; Tyler-Smith, Chris (2000), „Nowe zastosowania nowych haplotypów”, Trends in Genetics , 16 (8): 356–62, doi : 10.1016 / S0168-9525 (00) 02057-6 , PMID 10904265
- Kaladjieva, Luba; Calafell, Francesc; Praca, Mark A; Angelicheva, Dora; i in. (luty 2001). „Wzory różnorodności genetycznej między i wewnątrz grup u Romów Vlax, ujawnione przez chromosom Y i mitochondrialne linie DNA” . Europejski Dziennik Genetyki Człowieka . 9 (2): 97–104. doi : 10.1038/sj.ejhg.5200597 .
- Karafet, Tatiana; Xu, Liping; Du, Ruofu; Wang, William; i in. (wrzesień 2001). „Historia populacji ojcowskiej Azji Wschodniej: źródła, wzorce i procesy mikroewolucyjne” . American Journal of Human Genetics . 69 (3): 615–628. doi : 10.1086/323299 .
- Semino, O.; Passarino, G; Oefner, PJ; Lin, AA; i in. (2000), „The Genetic Legacy of Paleolithic Homo sapiens sapiens in Extant Europes: AY Chromosome Perspective”, Science , 290 (5494): 1155–9, Bibcode : 2000Sci... 290.1155S , doi : 10.1126/science.290.5494. 1155 , PMID 11073453
- Su, Bing; Xiao, Junhua; Underhill, Piotr; Deka, Ranjan; i in. (grudzień 1999). „Dowody dotyczące chromosomu Y na migrację współczesnych ludzi na północ do Azji Wschodniej podczas ostatniej epoki lodowcowej” . American Journal of Human Genetics . 65 (6): 1718–1724. doi : 10.1086/302680 .
- Underhill, Peter A.; Shen, Peidong; Lin, Alicja A.; Jin, Li; i in. (listopad 2000). „Zmienność sekwencji chromosomu Y i historia populacji ludzkich”. Genetyka przyrody . 26 (3): 358–361. doi : 10.1038/81685 .