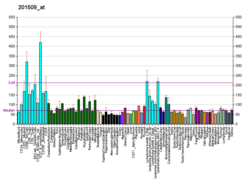
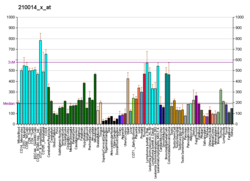
IDH3B
| Identyfikatory | |||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| IDH3B | |||||||||||||||||||||||||||||||||||||||||||||||
| , H-IDHB, RP46, dehydrogenaza izocytrynianowa 3 (NAD(+)) beta, dehydrogenaza izocytrynianowa (NAD(+)) 3 beta, dehydrogenaza izocytrynianowa (NAD(+)) 3 niekatalityczna podjednostka beta Identyfikatory | |||||||||||||||||||||||||||||||||||||||||||||||
| zewnętrzne | |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| Wikidane | |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
Dehydrogenaza izocytrynianowa [NAD] podjednostka beta, mitochondrialna jest enzymem , który u ludzi jest kodowany przez gen IDH3B .
Dehydrogenazy izocytrynianowe (IDH) katalizują oksydacyjną dekarboksylację izocytrynianu do 2- oksoglutaranu . Enzymy te należą do dwóch odrębnych podklas, z których jedna wykorzystuje NAD (+) jako akceptor elektronów , a druga NADP(+). Zgłoszono pięć dehydrogenaz izocytrynianowych: trzy dehydrogenazy izocytrynianowe zależne od NAD(+), które lokalizują się w macierzy mitochondrialnej, oraz dwie NADP (+), z których jedna jest mitochondrialna a drugi głównie cytozolowy . Zależne od NAD (+) dehydrogenazy izocytrynianowe katalizują allosterycznie regulowany etap ograniczający szybkość cyklu kwasu trikarboksylowego . Każdy izozym jest heterotetramerem , który składa się z dwóch podjednostek alfa , jednej podjednostki beta i jednej podjednostki gamma. Białko kodowane przez ten gen jest podjednostką beta jednego izoenzymu dehydrogenazy izocytrynianowej zależnej od NAD(+). Dla tego genu opisano trzy alternatywne splicingowe warianty transkryptu kodujące różne izoformy. [dostarczone przez RefSeq, lipiec 2008]
Struktura
IDH3 jest jednym z trzech izozymów dehydrogenazy izocytrynianowej, pozostałe dwa to IDH1 i IDH2 i są kodowane przez jeden z pięciu genów dehydrogenazy izocytrynianowej, którymi są IDH1 , IDH2 , IDH3A , IDH3B i IDH3G . Geny IDH3A , IDH3B i IDH3G kodują podjednostki IDH3, który jest heterotetramerem złożonym z dwóch podjednostek α 37 kDa (IDH3α), jednej podjednostki β 39 kDa (IDH3β) i jednej podjednostki γ 39 kDa (IDH3γ), każdy z odrębnym punkty izoelektryczne . Dopasowanie ich sekwencji aminokwasowych ujawnia ~ 40% identyczności między IDH3α i IDH3β, ~ 42% identyczności między IDH3α i IDH3γ i jeszcze bliższą identyczność 53% między IDH3β i IDH3γ, co daje ogólną identyczność 34% i 23% podobieństwo we wszystkich trzy typy podjednostek. Warto zauważyć, że Arg 88 w IDH3α jest niezbędny dla aktywności katalitycznej IDH3, podczas gdy równoważne Arg99 w IDH3β i Arg97 w IDH3γ są w dużej mierze zaangażowane w allosteryczną regulację enzymu przez ADP i NAD. Zatem możliwe jest, że te podjednostki powstały w wyniku duplikacji genów wspólnego genu przodków, a oryginalna katalityczna reszta Arg została dostosowana do funkcji allosterycznych w podjednostkach β i γ. Podobnie, Asp181 w IDH3α jest niezbędny do katalizy, podczas gdy równoważny Asp192 w IDH3β i Asp190 w IDH3γ wzmacniają wiązanie NAD i Mn2 + . Ponieważ oksydacyjna dekarboksylacja katalizowana przez IDH3 wymaga wiązania NAD, Mn 2+ i substratu izocytrynian, wszystkie trzy podjednostki biorą udział w reakcji katalitycznej. Ponadto badania enzymu w sercu świni wykazały, że dimery αβ i αγ stanowią dwa miejsca wiązania dla każdego z jego ligandów , w tym izocytrynianu, Mn 2+ i NAD, w jednym tetramerze IDH3.
izoformy
Gen IDH3B zawiera 12 eksonów i koduje dwie izoformy o alternatywnym splicingu : IDH3β1 (349 reszt ) i IDH3β2 (354 reszt). Te izoformy są tkankowo specyficzne i mają optymalne pH odpowiadające pH tkanek docelowych. IDH3β1, przy optymalnym pH 8,0, ulega ekspresji w mózgu i nerkach , podczas gdy IDH3β2, przy optymalnym pH 7,6, ulega ekspresji w sercu i mięśniach szkieletowych .
Funkcjonować
Jako dehydrogenaza izocytrynianowa, IDH3 katalizuje odwracalną dekarboksylację oksydacyjną izocytrynianu z wytworzeniem α-ketoglutaranu (α-KG) i CO2 jako części cyklu TCA w metabolizmie glukozy. Ten etap pozwala również na jednoczesną redukcję NAD+ do NADH, który jest następnie wykorzystywany do generowania ATP poprzez łańcuch transportu elektronów . Warto zauważyć, że IDH3 opiera się na NAD + jako akceptorze elektronów , w przeciwieństwie do NADP+, takich jak IDH1 i IDH2. Aktywność IDH3 jest regulowana potrzebami energetycznymi komórki: gdy komórka potrzebuje energii, IDH3 jest aktywowany przez ADP; a kiedy energia nie jest już potrzebna, IDH3 jest hamowany przez ATP i NADH. Ta allosteryczna regulacja pozwala IDH3 działać jako krok ograniczający szybkość w cyklu TCA. Zaobserwowano, że w komórkach IDH3 i jego podjednostki lokalizują się w mitochondriach .
Znaczenie kliniczne
Homozygotyczne mutacje powodujące utratę funkcji genu IDH3B powiązano z barwnikowym zwyrodnieniem siatkówki , neurodegeneracją pręcików i czopków w siatkówce , prowadzącą do ślepoty.
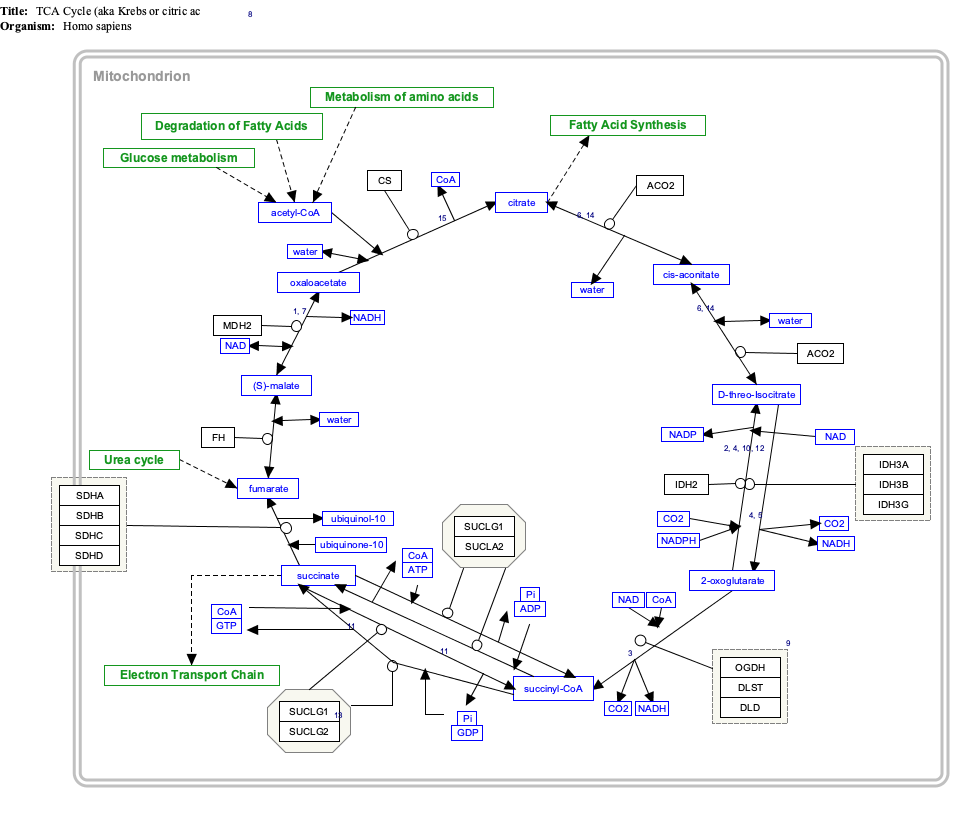
Interaktywna mapa szlaków
Kliknij geny, białka i metabolity poniżej, aby przejść do odpowiednich artykułów.
Zobacz też
Dalsza lektura
- Kim YO, Koh HJ, Kim SH i in. (2000). „Identyfikacja i charakterystyka funkcjonalna nowej, tkankowo specyficznej izoformy podjednostki beta dehydrogenazy izocytrynianowej zależnej od NAD (+)” . J. Biol. chemia . 274 (52): 36866–75. doi : 10.1074/jbc.274.52.36866 . PMID 10601238 .
- Weiss C, Zeng Y, Huang J i in. (2000). „Bovine NAD +-zależna dehydrogenaza izocytrynianowa: alternatywny splicing i zależna od tkanki ekspresja podjednostki 1”. Biochemia . 39 (7): 1807–16. doi : 10.1021/bi991691i . PMID 10677231 .
- Deloukas P, Matthews LH, Ashurst J i in. (2002). „Sekwencja DNA i analiza porównawcza ludzkiego chromosomu 20” . Natura . 414 (6866): 865–71. Bibcode : 2001Natur.414..865D . doi : 10.1038/414865a . PMID 11780052 .
- Strausberg RL, Feingold EA, Grouse LH i in. (2003). „Generowanie i wstępna analiza ponad 15 000 pełnej długości sekwencji cDNA człowieka i myszy” . proc. Natl. Acad. nauka USA . 99 (26): 16899–903. Bibcode : 2002PNAS...9916899M . doi : 10.1073/pnas.242603899 . PMC 139241 . PMID 12477932 .
- Ota T, Suzuki Y, Nishikawa T i in. (2004). „Kompletne sekwencjonowanie i charakterystyka 21 243 pełnej długości ludzkich cDNA” . Nat. Genet . 36 (1): 40–5. doi : 10.1038/ng1285 . PMID 14702039 .
- Gerhard DS, Wagner L, Feingold EA i in. (2004). „Stan, jakość i ekspansja projektu cDNA pełnej długości NIH: Kolekcja genów ssaków (MGC)” . Genom Res . 14 (10B): 2121–7. doi : 10.1101/gr.2596504 . PMC 528928 . PMID 15489334 .
- Kil IS, Park JW (2005). „Regulacja mitochondrialnej aktywności dehydrogenazy izocytrynianowej zależnej od NADP + przez glutationylację” . J. Biol. chemia . 280 (11): 10846–54. doi : 10.1074/jbc.M411306200 . PMID 15653693 .
- Rual JF, Venkatesan K, Hao T i in. (2005). „W kierunku mapy w skali proteomu sieci interakcji ludzkiego białka z białkiem”. Natura . 437 (7062): 1173-8. Bibcode : 2005Natur.437.1173R . doi : 10.1038/natura04209 . PMID 16189514 . S2CID 4427026 .
- Lim J, Hao T, Shaw C i in. (2006). „Sieć interakcji białko-białko dla ludzkich dziedzicznych ataksji i zaburzeń zwyrodnienia komórek Purkinjego” . komórka . 125 (4): 801–14. doi : 10.1016/j.cell.2006.03.032 . PMID 16713569 . S2CID 13709685 .
- Soundar S, O'hagan M, Fomulu KS, Colman RF (2006). „Identyfikacja asparaginianów wiążących Mn2 + z podjednostek alfa, beta i gamma ludzkiej dehydrogenazy izocytrynianowej zależnej od NAD” . J. Biol. chemia . 281 (30): 21073–81. doi : 10.1074/jbc.M602956200 . PMID 16737955 .
- Bzymek KP, Colman RF (2007). „Rola alfa-Asp181, beta-Asp192 i gamma-Asp190 w charakterystycznych podjednostkach ludzkiej dehydrogenazy izocytrynianowej specyficznej dla NAD”. Biochemia . 46 (18): 5391–7. doi : 10.1021/bi700061t . PMID 17432878 .