Zmienność genetyczna człowieka
Ludzka zmienność genetyczna to różnice genetyczne w populacjach i pomiędzy nimi . W populacji ludzkiej może istnieć wiele wariantów danego genu ( alleli ), co nazywa się polimorfizmem .
Nie ma dwóch identycznych genetycznie ludzi. Nawet bliźnięta monozygotyczne (które rozwijają się z jednej zygoty) mają rzadkie różnice genetyczne z powodu mutacji zachodzących podczas rozwoju i zmienności liczby kopii genów . Różnice między osobnikami, nawet blisko spokrewnionymi, są kluczem do takich technik, jak pobieranie genetycznych odcisków palców .
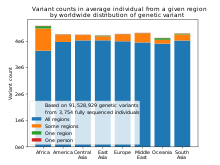
Ludzki genom ma całkowitą długość około 3,2 miliarda par zasad (pz) w 46 chromosomach DNA, a także nieco poniżej 17 000 pz DNA w mitochondriach komórkowych . W 2015 roku typową różnicę między genomem jednostki a genomem odniesienia oszacowano na 20 milionów par zasad (czyli 0,6% całości). Od 2017 roku było łącznie 324 milionów znanych wariantów zsekwencjonowanych ludzkich genomów .
Mówiąc porównawczo, ludzie są genetycznie jednorodnym gatunkiem. Chociaż niewielka liczba wariantów genetycznych występuje częściej w niektórych regionach geograficznych lub u osób pochodzących z tych regionów, ta zmienność odpowiada za niewielką część (~ 15%) zmienności ludzkiego genomu. Większość zmienności występuje w obrębie członków każdej populacji ludzkiej. Dla porównania makaki rezus wykazują 2,5-krotnie większą różnorodność sekwencji DNA w porównaniu z ludźmi.
Brak nieciągłości w odległościach genetycznych między populacjami ludzkimi, brak odrębnych gałęzi w gatunku ludzkim i uderzająca jednorodność istot ludzkich na całym świecie implikują, że nie ma naukowych podstaw do wnioskowania o rasach lub podgatunkach u ludzi, a dla większości cech istnieje znacznie większe zróżnicowanie wewnątrz populacji niż między nimi. Mimo to współczesne badania genetyczne wykazały znaczne średnie różnice genetyczne między populacjami ludzkimi pod względem cech, takich jak kolor skóry, wymiary ciała, trawienie laktozy i skrobi, przystosowanie do dużych wysokości i predyspozycje do rozwoju określonych chorób. Największa różnorodność występuje w obrębie i wśród populacji w Afryce i stopniowo maleje wraz ze wzrostem odległości od kontynentu afrykańskiego, zgodnie z teorią pochodzenia człowieka poza Afryką .
Badanie zmienności genetycznej człowieka ma znaczenie ewolucyjne i zastosowania medyczne. Może pomóc naukowcom zrekonstruować i zrozumieć wzorce migracji ludzi w przeszłości. W medycynie badanie zmienności genetycznej człowieka może być ważne, ponieważ niektóre allele chorobotwórcze występują częściej w pewnych grupach populacji. Na przykład mutacja anemii sierpowatej częściej występuje u osób wywodzących się z niektórych populacji Afryki Subsaharyjskiej, południowoeuropejskiej, arabskiej i indyjskiej, ze względu na presję ewolucyjną ze strony komarów przenoszących malarię w tych regionach.
Nowe odkrycia pokazują, że każdy człowiek ma średnio 60 nowych mutacji w porównaniu do swoich rodziców.
Przyczyny zmienności
Przyczyny różnic między osobnikami obejmują niezależny asortyment , wymianę genów (crossing over i rekombinację) podczas rozmnażania (poprzez mejozę ) oraz różne zdarzenia mutacyjne .
Istnieją co najmniej trzy powody, dla których istnieje zmienność genetyczna między populacjami. Dobór naturalny może dawać przewagę adaptacyjną jednostkom w określonym środowisku, jeśli allel zapewnia przewagę konkurencyjną. Allele poddane selekcji prawdopodobnie wystąpią tylko w tych regionach geograficznych, w których dają przewagę. Drugim ważnym procesem jest dryf genetyczny , będący efektem przypadkowych zmian w puli genowej, w warunkach, w których większość mutacji jest neutralna (to znaczy nie wydają się mieć żadnego pozytywnego ani negatywnego selektywnego wpływu na organizm). Wreszcie, małe populacje migrantów wykazują różnice statystyczne – zwane efektem założyciela – od ogółu populacji, z których pochodzą; kiedy ci migranci osiedlają się na nowych obszarach, ich populacja potomna zazwyczaj różni się od populacji pochodzenia: dominują inne geny i jest mniej zróżnicowana genetycznie.
U ludzi główną przyczyną jest dryf genetyczny . Szeregowe efekty założycielskie i mała wielkość populacji w przeszłości (zwiększająca prawdopodobieństwo dryfu genetycznego) mogły mieć istotny wpływ na neutralne różnice między populacjami. [ potrzebne źródło ] Drugą główną przyczyną zmienności genetycznej jest wysoki stopień neutralności większości mutacji . Wydaje się, że niewielka, ale znacząca liczba genów przeszła niedawno naturalną selekcję, a te presje selekcyjne są czasami specyficzne dla jednego regionu.
Miary zmienności
Zmienność genetyczna wśród ludzi występuje w wielu skalach, od poważnych zmian w ludzkim kariotypie do zmian pojedynczych nukleotydów . Aberracje chromosomowe są wykrywane w 1 na 160 urodzeń żywych ludzi. Poza zaburzeniami chromosomów płciowych większość przypadków aneuploidii kończy się śmiercią rozwijającego się płodu ( poronienie ); najczęstsze dodatkowe autosomalne wśród żywych urodzeń to 21 , 18 i 13 .
Różnorodność nukleotydów to średni odsetek nukleotydów, które różnią się między dwoma osobnikami. Od 2004 r. Różnorodność ludzkich nukleotydów oszacowano na 0,1% do 0,4% par zasad . W 2015 roku projekt 1000 genomów , który zsekwencjonował tysiąc osobników z 26 populacji ludzkich, stwierdził, że „typowy [indywidualny] genom różni się od referencyjnego genomu ludzkiego w 4,1 miliona do 5,0 milionów miejsc… wpływając na 20 milionów zasad sekwencji”; ta ostatnia liczba odpowiada 0,6% całkowitej liczby par zasad. Prawie wszystkie (>99,9%) z tych miejsc to niewielkie różnice, albo polimorfizmy pojedynczych nukleotydów, albo krótkie insercje lub delecje ( indele ) w sekwencji genetycznej, ale zmiany strukturalne odpowiadają za większą liczbę par zasad niż SNP i indele.
Od 2017 r. Baza danych polimorfizmu pojedynczego nukleotydu ( dbSNP ), która zawiera listę SNP i innych wariantów, zawiera listę 324 milionów wariantów znalezionych w zsekwencjonowanych ludzkich genomach.
Polimorfizmy pojedynczych nukleotydów
pojedynczego nukleotydu (SNP) to różnica w pojedynczym nukleotydzie między członkami jednego gatunku, która występuje u co najmniej 1% populacji. Wśród 2504 osób scharakteryzowanych przez projekt 1000 genomów było 84,7 miliona SNP. SNP są najczęstszym rodzajem zmienności sekwencji, oszacowano w 1998 r., że stanowią 90% wszystkich wariantów sekwencji. Inne warianty sekwencji to wymiany pojedynczych zasad, delecje i insercje. SNP występują średnio co około 100 do 300 zasad i dlatego są głównym źródłem heterogeniczności.
Funkcjonalny lub niesynonimowy SNP to taki, który wpływa na niektóre czynniki, takie jak składanie genów lub informacyjny RNA , a tym samym powoduje różnicę fenotypową między członkami gatunku. Około 3% do 5% ludzkich SNP jest funkcjonalnych (patrz International HapMap Project ). Neutralne lub synonimiczne SNP są nadal przydatne jako markery genetyczne w badaniach asocjacyjnych całego genomu , ze względu na ich samą liczbę i stabilne dziedziczenie przez pokolenia.
Kodujący SNP to taki, który występuje w genie. Istnieje 105 ludzkich referencyjnych SNP, które powodują przedwczesne kodony stop w 103 genach. Odpowiada to 0,5% kodujących SNP. Występują z powodu duplikacji segmentowej w genomie. Te SNP powodują utratę białka, jednak wszystkie te allele SNP są powszechne i nie są oczyszczane w selekcji negatywnej .
Zmienność strukturalna
Zmienność strukturalna to zmiana struktury chromosomu organizmu . Zmiany strukturalne, takie jak zmienność liczby kopii i delecje , inwersje , insercje i duplikacje , odpowiadają za znacznie większą zmienność genetyczną człowieka niż różnorodność pojedynczych nukleotydów. Stwierdzono to w 2007 roku na podstawie analizy diploidalnych pełnych sekwencji genomów dwóch ludzi: Craiga Ventera i Jamesa D. Watsona . To dodało do dwóch haploidalnych sekwencje, które były połączeniem sekwencji pochodzących od wielu osobników, opublikowanych odpowiednio przez Human Genome Project i Celera Genomics .
Według projektu 1000 genomów typowy człowiek ma od 2100 do 2500 odmian strukturalnych, w tym około 1000 dużych delecji, 160 wariantów liczby kopii, 915 insercji Alu, 128 insercji L1, 51 insercji SVA, 4 NUMT i 10 inwersji .
Kopiuj odmianę numeru
Zmienność liczby kopii (CNV) to różnica w genomie spowodowana usunięciem lub powieleniem dużych obszarów DNA na pewnym chromosomie. Szacuje się, że 0,4% genomów niespokrewnionych ludzi różni się liczbą kopii. Uwzględniając zmienność liczby kopii, szacuje się, że zmienność genetyczna między ludźmi wynosi co najmniej 0,5% (podobieństwo 99,5%). Odmiany liczby kopii są dziedziczone, ale mogą również powstać podczas rozwoju.
Wizualna mapa z regionami o dużej zmienności genomowej zespołu referencyjnego współczesnego człowieka w stosunku do 50-tysięcznego neandertalczyka została zbudowana przez Pratasa i in.
Epigenetyka
epigenetyczna to zmienność znaczników chemicznych, które przyczepiają się do DNA i wpływają na sposób odczytywania genów. Znaczniki, „zwane oznaczeniami epigenetycznymi, działają jak przełączniki kontrolujące sposób odczytywania genów”. W przypadku niektórych alleli stan epigenetyczny DNA i związany z nim fenotyp mogą być dziedziczone z pokolenia na pokolenie .
Zmienność genetyczna
Zmienność genetyczna jest miarą tendencji poszczególnych genotypów w populacji do różnicowania się (różnienia się) od siebie. Zmienność różni się od różnorodności genetycznej , która jest wielkością zmienności obserwowanej w określonej populacji. Zmienność cechy to stopień, w jakim cecha ta zmienia się w odpowiedzi na wpływy środowiskowe i genetyczne .
kliny
W biologii klin jest kontinuum gatunków , populacji, odmian lub form organizmów, które wykazują stopniowe różnice fenotypowe i/lub genetyczne na obszarze geograficznym, zazwyczaj w wyniku heterogeniczności środowiskowej. W badaniach naukowych nad zmiennością genetyczną człowieka, klina genu może być rygorystycznie zdefiniowana i poddana pomiarom ilościowym.
Haplogrupy
W badaniu ewolucji molekularnej haplogrupa to grupa podobnych haplotypów , które mają wspólnego przodka z mutacją polimorfizmu pojedynczego nukleotydu (SNP). Badanie haplogrup dostarcza informacji o pochodzeniu przodków sprzed tysięcy lat.
Najczęściej badanymi ludzkimi haplogrupami są haplogrupy chromosomu Y (Y-DNA) i haplogrupy mitochondrialnego DNA (mtDNA) , z których obie można wykorzystać do zdefiniowania populacji genetycznych. Y-DNA jest przekazywane wyłącznie w patrylinearnej , z ojca na syna, podczas gdy mtDNA jest przekazywane w linii matrylinearnej , z matki na córkę lub syna. Y-DNA i mtDNA mogą zmieniać się przez przypadkowe mutacje w każdym pokoleniu.
Zmienna liczba powtórzeń tandemowych
Zmienna liczba powtórzeń tandemowych (VNTR) to zmiana długości powtórzeń tandemowych . Powtórzenie tandemowe to sąsiednie powtórzenie krótkiej sekwencji nukleotydowej . Powtórzenia tandemowe występują na wielu chromosomach , a ich długość jest różna u poszczególnych osób. Każdy wariant działa jako odziedziczony allel , więc są one używane do identyfikacji osobistej lub rodzicielskiej. Ich analiza jest przydatna w badaniach genetycznych i biologicznych, kryminalistyce i pobieraniu odcisków palców DNA .
Krótkie powtórzenia tandemowe (około 5 par zasad) nazywane są mikrosatelitami , a dłuższe minisatelitami .
Historia i rozmieszczenie geograficzne
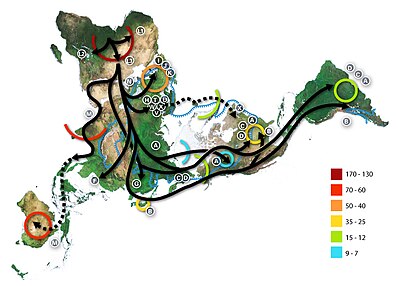
Niedawne afrykańskie pochodzenie współczesnych ludzi
Niedawny paradygmat afrykańskiego pochodzenia współczesnych ludzi zakłada rozproszenie nieafrykańskich populacji anatomicznie współczesnych ludzi po 70 000 lat temu. Rozproszenie w Afryce nastąpiło znacznie wcześniej, co najmniej 130 000 lat temu. Teoria „poza Afryką” wywodzi się z XIX wieku jako wstępna sugestia w książce Karola Darwina „ Pochodzenie człowieka” , ale pozostawała spekulacją aż do lat 80 . archaicznych okazów .
Według badania z 2000 r. Dotyczącego zmienności sekwencji chromosomu Y, ludzkie chromosomy Y śledzą pochodzenie z Afryki, a potomkowie pochodnej linii opuścili Afrykę i ostatecznie zostali zastąpieni archaicznymi ludzkimi chromosomami Y w Eurazji. Badanie pokazuje również, że mniejszość współczesnych populacji w Afryce Wschodniej i Khoisan to potomkowie najbardziej przodków patrylinearów anatomicznie współczesnych ludzi, którzy opuścili Afrykę 35 000 do 89 000 lat temu. Innym dowodem potwierdzającym tę teorię jest to, że różnice w pomiarach czaszki zmniejszają się wraz z odległością od Afryki w tym samym tempie, co spadek różnorodności genetycznej. Różnorodność genetyczna człowieka zmniejsza się w rodzimych populacjach wraz z odległością migracji od Afryki i uważa się, że jest to spowodowane wąskich gardeł podczas migracji ludzi, które są zdarzeniami, które tymczasowo zmniejszają wielkość populacji.
Badanie klastrów genetycznych z 2009 r., W ramach którego genotypowano 1327 markerów polimorficznych w różnych populacjach afrykańskich, zidentyfikowano sześć klastrów przodków. Grupowanie ściśle korespondowało z pochodzeniem etnicznym, kulturą i językiem. sekwencjonowania całego genomu populacji świata z 2018 r. zaobserwowano podobne skupiska wśród populacji w Afryce. Przy K = 9 odrębne komponenty przodków definiowały populacje mówiące w języku afroazjatyckim zamieszkujące Afrykę Północną i Afrykę Północno-Wschodnią ; populacje mówiące po nilo -saharyjsku w Afryce Północno-Wschodniej i Afryce Wschodniej ; populacje Ari w północno-wschodniej Afryce ; populacje posługujące się językiem nigeryjsko -kongijskim w Afryce Środkowo-Zachodniej, Afryce Zachodniej , Afryce Wschodniej i Afryce Południowej ; populacje Pigmejów w Afryce Środkowej ; oraz Khoisan w Afryce Południowej.
Genetyka populacji
Ze względu na wspólne pochodzenie wszystkich ludzi tylko niewielka liczba wariantów ma duże różnice w częstotliwości między populacjami. Jednak niektóre rzadkie warianty w światowej populacji ludzkiej występują znacznie częściej w co najmniej jednej populacji (ponad 5%).
Powszechnie przyjmuje się, że pierwsi ludzie opuścili Afrykę, a zatem musieli przejść przez wąskie gardło populacji przed ich dywergencją afrykańsko-euroazjatycką około 100 000 lat temu (około 3000 pokoleń). Gwałtowna ekspansja wcześniej małej populacji ma dwa ważne skutki dla rozmieszczenia zmienności genetycznej. Po pierwsze, tak zwany efekt założyciela występuje, gdy populacje założycielskie przynoszą tylko podzbiór zmienności genetycznej z populacji swoich przodków. Po drugie, w miarę jak założyciele stają się coraz bardziej oddaleni geograficznie, prawdopodobieństwo, że dwie osoby z różnych populacji założycielskich połączą się w pary, maleje. Efektem tego kojarzenia selektywnego jest zmniejszenie przepływu genów między grupami geograficznymi i zwiększenie dystansu genetycznego między grupami. [ potrzebne źródło ]
Ekspansja ludzi z Afryki wpłynęła na rozmieszczenie zmienności genetycznej na dwa inne sposoby. Po pierwsze, mniejsze populacje (założycielskie) doświadczają większego dryfu genetycznego z powodu zwiększonych fluktuacji neutralnych polimorfizmów. Po drugie, prawdopodobieństwo przeniesienia nowych polimorfizmów, które powstały w jednej grupie do innych grup, było mniejsze, ponieważ przepływ genów był ograniczony. [ potrzebne źródło ]
Populacje w Afryce mają zwykle mniejszy stopień nierównowagi powiązań niż populacje poza Afryką, częściowo z powodu większych populacji ludzkich w Afryce w ciągu historii ludzkości, a częściowo z powodu liczby współczesnych ludzi, którzy opuścili Afrykę, aby skolonizować resztę świat wydaje się być stosunkowo niski. W przeciwieństwie do tego, populacje, które w przeszłości uległy dramatycznemu zmniejszeniu lub gwałtownej ekspansji, oraz populacje utworzone przez mieszankę wcześniej oddzielnych grup przodków, mogą mieć niezwykle wysoki poziom nierównowagi powiązań
Dystrybucja zmienności

Rozmieszczenie wariantów genetycznych w populacjach ludzkich i pomiędzy nimi jest niemożliwe do zwięzłego opisania ze względu na trudność zdefiniowania „populacji”, kliniczną naturę zmienności i heterogeniczność całego genomu (Long i Kittles 2003). Ogólnie jednak średnio 85% zmienności genetycznej występuje w populacjach lokalnych, ~7% między lokalnymi populacjami na tym samym kontynencie, a ~8% zmienności występuje między dużymi grupami żyjącymi na różnych kontynentach. Niedawne pochodzenie afrykańskie Teoria dla ludzi przewidywałaby, że w Afryce istnieje znacznie większa różnorodność niż gdzie indziej i że różnorodność powinna się zmniejszać, im dalej od Afryki pobierana jest próbka populacji.
Zmienność fenotypowa
Afryka Subsaharyjska ma największą różnorodność genetyczną człowieka i wykazano, że to samo odnosi się do zmienności fenotypowej kształtu czaszki. Fenotyp jest połączony z genotypem poprzez ekspresję genów . Różnorodność genetyczna zmniejsza się płynnie wraz z odległością migracji od tego regionu, który według wielu naukowców jest źródłem współczesnego człowieka, a spadek ten odzwierciedla spadek zmienności fenotypowej. Pomiary czaszki są przykładem atrybutu fizycznego, którego zmienność wewnątrz populacji zmniejsza się wraz z odległością od Afryki.
Rozmieszczenie wielu cech fizycznych przypomina rozmieszczenie zmienności genetycznej w populacjach ludzkich i pomiędzy nimi ( Amerykańskie Stowarzyszenie Antropologów Fizycznych 1996; Keita i Kittles 1997). Na przykład ~90% różnic w kształtach ludzkich głów występuje w obrębie grup kontynentalnych, a ~10% oddziela grupy, z większą zmiennością kształtu głowy wśród osobników, których przodkowie pochodzili z Afryki (Relethford 2002).
Znaczącym wyjątkiem od powszechnego rozkładu cech fizycznych w obrębie grup i pomiędzy nimi jest kolor skóry . Około 10% różnic w kolorze skóry występuje w grupach, a ~90% między grupami (Relethford 2002). Ten rozkład koloru skóry i jego wzorce geograficzne - ludzie, których przodkowie mieszkali głównie w pobliżu równika, mieli ciemniejszą skórę niż ci, których przodkowie żyli głównie na wyższych szerokościach geograficznych - wskazują, że ta cecha była pod silną presją selekcyjną . Wydaje się, że ciemniejsza skóra jest silnie wybierana w regionach równikowych, aby zapobiegać poparzeniom słonecznym, rakowi skóry itp fotoliza kwasu foliowego i uszkodzenie gruczołów potowych.
Zrozumienie, w jaki sposób różnorodność genetyczna w populacji ludzkiej wpływa na różne poziomy ekspresji genów, jest aktywnym obszarem badań. Podczas gdy wcześniejsze badania koncentrowały się na związku między zmiennością DNA a ekspresją RNA, nowsze wysiłki charakteryzują kontrolę genetyczną różnych aspektów ekspresji genów, w tym stanów chromatyny, translacji i poziomów białek. Badanie opublikowane w 2007 roku wykazało, że 25% genów wykazywało różne poziomy ekspresji genów między populacjami pochodzenia europejskiego i azjatyckiego. Uważano, że główną przyczyną tej różnicy w ekspresji genów są SNP w regionach regulatorowych genów DNA. Inne badanie opublikowane w 2007 roku wykazało, że około 83% genów ulegało ekspresji na różnych poziomach u poszczególnych osób i około 17% między populacjami pochodzenia europejskiego i afrykańskiego.
Wskaźnik fiksacji Wrighta jako miara zmienności
Genetyk populacji Sewall Wright opracował wskaźnik fiksacji (często w skrócie F ST ) jako sposób pomiaru różnic genetycznych między populacjami. Ta statystyka jest często używana w taksonomii do porównywania różnic między dowolnymi dwiema populacjami poprzez pomiar różnic genetycznych między populacjami i między populacjami dla poszczególnych genów lub dla wielu genów jednocześnie. Często podaje się, że wskaźnik fiksacji u ludzi wynosi około 0,15. Przekłada się to na szacunkowe 85% zmienności mierzonej w całej populacji ludzkiej w obrębie osobników z tej samej populacji, a około 15% zmienności występuje między populacjami. Szacunki te sugerują, że dowolne dwie osoby z różnych populacji mogą być bardziej podobne do siebie niż do członka własnej grupy. „Wspólna historia ewolucyjna żyjących ludzi zaowocowała wysokim pokrewieństwem wśród wszystkich żyjących ludzi, na co wskazuje na przykład bardzo niski wskaźnik fiksacji (F ST ) wśród żywych populacji ludzkich.” Richard Lewontin , który potwierdził te proporcje, doszedł do wniosku, że ani „rasa”, ani „podgatunki” nie są odpowiednimi ani użytecznymi sposobami opisywania populacji ludzkich.
Sam Wright uważał, że wartości > 0,25 reprezentują bardzo dużą zmienność genetyczną, a F ST wynoszący 0,15–0,25 reprezentuje dużą zmienność. Jednak około 5% zmienności ludzkiej występuje między populacjami na kontynentach, dlatego w niektórych badaniach stwierdzono wartości F ST między kontynentalnymi grupami ludzi (lub rasami) na poziomie zaledwie 0,1 (lub prawdopodobnie niższym), co sugeruje bardziej umiarkowane poziomy genetyczne zmiana. Graves (1996) sprzeciwił się temu, że F ST nie powinny być używane jako wskaźnik statusu podgatunku, ponieważ statystyka służy do pomiaru stopnia zróżnicowania między populacjami, chociaż patrz także Wright (1978).
Jeffrey Long i Rick Kittles podają obszerną krytykę zastosowania F ST do populacji ludzkich w swoim artykule z 2003 r. „Różnorodność genetyczna człowieka i brak ras biologicznych”. Stwierdzają, że liczba 85% jest myląca, ponieważ sugeruje, że wszystkie populacje ludzkie zawierają średnio 85% całej różnorodności genetycznej. Twierdzą, że podstawowy model statystyczny błędnie zakłada równe i niezależne historie zmienności dla każdej dużej populacji ludzkiej. Bardziej realistycznym podejściem jest zrozumienie, że niektóre grupy ludzkie są rodzicielskie w stosunku do innych grup i że te grupy reprezentują parafiletykę grupy do swoich grup pochodzenia. Na przykład, zgodnie z niedawną teorią pochodzenia afrykańskiego, populacja ludzka w Afryce jest parafiletyczna w stosunku do wszystkich innych grup ludzkich, ponieważ reprezentuje grupę przodków, z której wywodzą się wszystkie populacje nieafrykańskie, ale co więcej, grupy nieafrykańskie wywodzą się tylko z niewielkiej niereprezentatywna próba tej afrykańskiej populacji. Oznacza to, że wszystkie grupy spoza Afryki są bliżej spokrewnione ze sobą i z niektórymi grupami afrykańskimi (prawdopodobnie z Afryki Wschodniej) niż z innymi, a ponadto migracja z Afryki stanowiła genetyczne wąskie gardło , przy czym większość różnorodności, która istniała w Afryce, nie została przeniesiona z Afryki przez grupy emigrantów. W tym scenariuszu populacje ludzkie nie mają równego poziomu zmienności lokalnej, ale raczej zmniejszają się, im dalej od Afryki żyje jakakolwiek populacja. Long i Kittles stwierdzili, że zamiast 85% ludzkiej różnorodności genetycznej we wszystkich populacjach ludzkich, około 100% ludzkiej różnorodności występuje w pojedynczej populacji afrykańskiej, podczas gdy tylko około 70% ludzkiej różnorodności genetycznej istnieje w populacji pochodzącej z Nowej Gwinei. Long i Kittles argumentowali, że nadal tworzy to globalną populację ludzką, która jest genetycznie jednorodna w porównaniu z innymi populacjami ssaków.
Archaiczna domieszka
Anatomicznie współcześni ludzie krzyżowali się z neandertalczykami w okresie środkowego paleolitu . W maju 2010 r. Neanderthal Genome Project przedstawił dowody genetyczne na to, że miało miejsce krzyżowanie się i że niewielka, ale znacząca część, około 2-4% domieszki neandertalczyka, jest obecna w DNA współczesnych mieszkańców Eurazji i Oceanii, a prawie nieobecna w subsaharyjskiej populacje afrykańskie.
, że od 4% do 6% genomu Melanezyjczyków (reprezentowanych przez mieszkańców Papui-Nowej Gwinei i wysp Bougainville) pochodzi od denisowian – nieznanego wcześniej hominina, który jest bliżej spokrewniony z neandertalczykami niż z sapiens. Prawdopodobnie został wprowadzony podczas wczesnej migracji przodków Melanezyjczyków do Azji Południowo-Wschodniej. Ta historia interakcji sugeruje, że Denisowianie kiedyś zamieszkiwali wschodnią Azję.
W ten sposób Melanezyjczycy wyłaniają się jako jedna z najbardziej archaicznych populacji z domieszkami, z domieszką związaną z denisowianami / neandertalczykami wynoszącą ~ 8%.
W badaniu opublikowanym w 2013 roku Jeffrey Wall z University of California przeanalizował dane dotyczące całej sekwencji genomu i stwierdził wyższe wskaźniki introgresji u Azjatów w porównaniu z Europejczykami. Młotek i in. przetestował hipotezę, że współczesne genomy afrykańskie mają sygnatury przepływu genów z archaicznymi przodkami człowieka i znalazł dowody na archaiczną domieszkę w genomach niektórych grup afrykańskich, co sugeruje, że niewielkie ilości przepływu genów były szeroko rozpowszechnione w czasie i przestrzeni podczas ewolucji anatomicznie współczesnych ludzi .
Badanie opublikowane w 2020 roku wykazało, że populacje Joruba i Mende w Afryce Zachodniej wywodzą od 2% do 19% ich genomu z jeszcze niezidentyfikowanej archaicznej populacji homininów, która prawdopodobnie rozeszła się przed podziałem współczesnych ludzi i przodków neandertalczyków i denisowian , potencjalnie czyniąc te grupy najbardziej archaicznymi zmieszanymi populacjami ludzkimi, jakie dotychczas zidentyfikowano.
Kategoryzacja ludności świata
Nowe dane dotyczące zmienności genetycznej człowieka ponownie rozpaliły debatę na temat możliwych biologicznych podstaw kategoryzacji ludzi na rasy. Większość kontrowersji dotyczy tego, jak interpretować dane genetyczne i czy oparte na nich wnioski są słuszne. Niektórzy badacze twierdzą, że samozidentyfikowana rasa może być używana jako wskaźnik pochodzenia geograficznego dla niektórych zagrożeń dla zdrowia i leków .
Chociaż różnice genetyczne między grupami ludzkimi są stosunkowo niewielkie, te różnice w niektórych genach, takich jak duffy , ABCC11 , SLC24A5 , zwane markerami informacji o przodkach (AIM), można jednak wykorzystać do wiarygodnego umiejscowienia wielu osób w szerokich grupach geograficznych. Na przykład komputerowe analizy setek polimorficznych loci pobranych w globalnie rozproszonych populacjach ujawniły istnienie klastrów genetycznych, które z grubsza są powiązane z grupami, które historycznie zajmowały duże regiony kontynentalne i subkontynentalne (Rosenberg i in. 2002; Bamshad i in. 2003).
Niektórzy komentatorzy argumentowali, że te wzorce zmienności stanowią biologiczne uzasadnienie stosowania tradycyjnych kategorii rasowych. Twierdzą, że skupienia kontynentalne odpowiadają z grubsza podziałowi istot ludzkich na mieszkańców Afryki Subsaharyjskiej ; Europejczycy , mieszkańcy Azji Zachodniej , Azji Środkowej , Azji Południowej i Afryki Północnej ; mieszkańcy Azji Wschodniej , Azji Południowo-Wschodniej , Polinezyjczycy i rdzenni Amerykanie ; i inni mieszkańcy ul Oceania (Melanezyjczycy, Mikronezyjczycy i australijscy Aborygeni) (Risch i in. 2002). Inni obserwatorzy nie zgadzają się, twierdząc, że te same dane podważają tradycyjne pojęcia grup rasowych (King i Motulsky 2002; Calafell 2003; Tishkoff i Kidd 2004). Wskazują na przykład, że główne populacje uważane za rasy lub podgrupy w ramach ras niekoniecznie tworzą własne skupiska.
Kategorie rasowe są również podważane przez ustalenia, że warianty genetyczne, które są ograniczone do jednego regionu, są na ogół rzadkie w tym regionie, warianty, które są powszechne w regionie, zwykle występują na całym świecie, a większość różnic między jednostkami, niezależnie od tego, czy pochodzą one z w tym samym regionie lub w różnych regionach, wynikają z globalnych wariantów. Nie znaleziono żadnych wariantów genetycznych, które są ustalone na kontynencie lub głównym regionie i nie występują nigdzie indziej.
Co więcej, ponieważ zmienność genetyczna człowieka jest kliniczna, wiele osób należy do dwóch lub więcej grup kontynentalnych. Zatem genetycznie oparte „pochodzenie biogeograficzne” przypisane danej osobie będzie na ogół szeroko rozpowszechnione i będzie mu towarzyszyć spora niepewność (Pfaff i in. 2004).
W wielu częściach świata grupy mieszały się w taki sposób, że wiele osobników ma stosunkowo niedawnych przodków z bardzo odległych regionów. Chociaż analizy genetyczne dużej liczby loci mogą dać oszacowanie odsetka przodków danej osoby pochodzących z różnych populacji kontynentalnych (Shriver i in. 2003; Bamshad i in. 2004), szacunki te mogą zakładać fałszywą odrębność populacji rodzicielskich, ponieważ grupy ludzkie wymieniały się partnerami ze skali lokalnej na kontynentalną w całej historii (Cavalli-Sforza i in. 1994; Hoerder 2002). Nawet przy dużej liczbie markerów informacje potrzebne do oszacowania proporcji domieszek osobników lub grup są ograniczone, a szacunki zazwyczaj będą miały szerokie przedziały ufności (Pfaff et al. 2004).
Grupowanie genetyczne
Dane genetyczne można wykorzystać do wnioskowania o strukturze populacji i przypisania jednostek do grup, które często odpowiadają ich samodzielnie określonemu pochodzeniu geograficznemu. Jorde i Wooding (2004) argumentowali, że „Analiza wielu loci daje obecnie dość dokładne oszacowania podobieństwa genetycznego między osobnikami, a nie populacjami. Grupowanie osobników jest skorelowane z pochodzeniem geograficznym lub przodkami”. Jednak identyfikacja według pochodzenia geograficznego może szybko się załamać, biorąc pod uwagę wspólne pochodzenie historyczne między jednostkami w przeszłości.
Analiza autosomalnych danych SNP z Międzynarodowego Projektu HapMap (faza II) i próbek Panelu Różnorodności Genomu Ludzkiego CEPH została opublikowana w 2009 roku. Badanie 53 populacji pobranych z danych HapMap i CEPH (1138 niepowiązanych osobników) zasugerowało, że dobór naturalny może kształtować ludzki genom znacznie wolniej niż wcześniej sądzono, a czynniki takie jak migracja w obrębie kontynentów i między kontynentami silniej wpływają na rozmieszczenie odmian genetycznych. Podobne badanie opublikowane w 2010 roku wykazało mocne dowody obejmujące cały genom na rzecz selekcji ze względu na zmiany w ekoregionie, diecie i sposobach utrzymania, szczególnie w związku z ekoregionami polarnymi, żerowaniem i dietą bogatą w korzenie i bulwy. W badaniu z 2016 r. analiza głównych składowych danych dotyczących całego genomu była w stanie odzyskać znane wcześniej cele do pozytywnej selekcji (bez wcześniejszego zdefiniowania populacji), a także szereg nowych genów kandydujących.
Antropologia sądowa
Antropolodzy sądowi mogą ocenić pochodzenie szczątków szkieletowych, analizując morfologię szkieletu, a także, jeśli to możliwe, używając markerów genetycznych i chemicznych. Chociaż oceny te nigdy nie są pewne, dokładność analiz morfologii szkieletu w określaniu prawdziwego pochodzenia oszacowano na 90%.

Przepływ genów i domieszki
Przepływ genów między dwiema populacjami zmniejsza średni dystans genetyczny między populacjami, tylko całkowicie odizolowane populacje ludzkie nie doświadczają przepływu genów, a większość populacji ma ciągły przepływ genów z innymi sąsiednimi populacjami, które tworzą rozkład kliniczny obserwowany dla większości zmienności genetycznych. Kiedy przepływ genów zachodzi między dobrze zróżnicowanymi populacjami genetycznymi, wynik określa się jako „domieszkę genetyczną”.
Mapowanie domieszek to technika stosowana do badania, w jaki sposób warianty genetyczne powodują różnice w częstości występowania chorób między populacjami. Niedawne populacje domieszek, które śledzą swoje pochodzenie na wielu kontynentach, dobrze nadają się do identyfikacji genów cech i chorób, które różnią się częstością występowania między populacjami rodzicielskimi. Populacje afroamerykańskie były przedmiotem licznych badań genetycznych populacji i mapowania domieszek, w tym badań złożonych cech genetycznych, takich jak liczba białych krwinek, wskaźnik masy ciała, rak prostaty i choroby nerek.
Przeprowadzono analizę zmienności fenotypowej i genetycznej, w tym koloru skóry i statusu społeczno-ekonomicznego, w populacji Republiki Zielonego Przylądka, która ma dobrze udokumentowaną historię kontaktów między Europejczykami a Afrykanami. Badania wykazały, że wzór domieszek w tej populacji był uwarunkowany płcią (obejmujący głównie kojarzenia Europejczyków i Afrykanek) i istnieje istotna interakcja między statusem społeczno-ekonomicznym a kolorem skóry, niezależnie od pochodzenia. Inne badanie pokazuje zwiększone ryzyko choroby przeszczep przeciwko gospodarzowi po przeszczepie z powodu wariantów genetycznych w ludzkim antygenie leukocytarnym (HLA) i białkach innych niż HLA.
Zdrowie
Różnice w częstości alleli przyczyniają się do różnic grupowych w częstości występowania niektórych chorób monogenowych i mogą przyczyniać się do różnic w częstości występowania niektórych powszechnych chorób. W przypadku chorób monogenowych częstość alleli sprawczych zwykle najlepiej koreluje z pochodzeniem, czy to rodzinnym (na przykład zespół Ellisa-Van Crevelda wśród amiszów z Pensylwanii ), etnicznym ( choroba Taya-Sachsa wśród populacji Żydów aszkenazyjskich ), czy geograficznym (hemoglobinopatie wśród ludzie, których przodkowie mieszkali w regionach malarycznych). W zakresie, w jakim pochodzenie odpowiada grupom lub podgrupom rasowym lub etnicznym, częstość występowania chorób monogenowych może różnić się między grupami sklasyfikowanymi według rasy lub pochodzenia etnicznego, a pracownicy służby zdrowia zazwyczaj biorą te wzorce pod uwagę przy stawianiu diagnoz.
Nawet w przypadku powszechnych chorób obejmujących liczne warianty genetyczne i czynniki środowiskowe badacze wskazują na dowody sugerujące udział alleli o różnym rozmieszczeniu z efektami od małych do umiarkowanych. Często cytowane przykłady to nadciśnienie (Douglas i in. 1996), cukrzyca (Gower i in. 2003), otyłość (Fernandez i in. 2003) oraz rak prostaty (Platz i in. 2000). Jednak w żadnym z tych przypadków nie wykazano, aby zmienność alleliczna w genie podatności odpowiadała za znaczną część różnicy w częstości występowania choroby między grupami, a rola czynników genetycznych w generowaniu tych różnic pozostaje niepewna (Mountain i Risch 2004).
Z drugiej strony niektóre inne odmiany są korzystne dla człowieka, ponieważ zapobiegają niektórym chorobom i zwiększają szansę przystosowania się do środowiska. Na przykład mutacja w CCR5 , który chroni przed AIDS . Gen CCR5 jest nieobecny na powierzchni komórki z powodu mutacji. Bez genu CCR5 na powierzchni HIV nie mają się do czego przyczepić i związać. Dlatego mutacja w genie CCR5 zmniejsza ryzyko indywidualnego ryzyka zachorowania na AIDS. Mutacja w CCR5 jest również dość powszechna na niektórych obszarach, przy czym ponad 14% populacji w Europie jest nosicielem mutacji i około 6–10% w Azji i Afryce Północnej .
Oprócz mutacji, wiele genów, które mogły pomagać ludziom w starożytności, nęka ludzi dzisiaj. Na przykład podejrzewa się, że geny, które pozwalają ludziom wydajniej przetwarzać żywność, to te, które sprawiają, że ludzie są dziś podatni na otyłość i cukrzycę.
Neil Risch z Uniwersytetu Stanforda zaproponował, że samozidentyfikowana rasa/grupa etniczna może być ważnym środkiem kategoryzacji w Stanach Zjednoczonych ze względów zdrowia publicznego i polityki. Artykuł Noaha Rosenberga z 2002 r. zawiera podobne twierdzenie: „Struktura populacji ludzkiej jest istotna w różnych kontekstach epidemiologicznych. W wyniku różnic w częstości zarówno genetycznych, jak i niegenetycznych czynników ryzyka, częstości występowania chorób i fenotypów takich jak niepożądana reakcja na lek różni się w zależności od populacji. Ponadto informacje o populacji pacjenta mogą dostarczyć lekarzom informacji o ryzyku, gdy bezpośrednie przyczyny choroby są nieznane”. Jednak w 2018 r Noah Rosenberg opublikował badanie kwestionujące genetycznie esencjalistyczne idee różnic zdrowotnych między populacjami, stwierdzające, że warianty środowiskowe są bardziej prawdopodobną przyczyną
Projekty genomu
Projekty dotyczące genomu ludzkiego to przedsięwzięcia naukowe mające na celu określenie lub badanie struktury ludzkiego genomu . Human Genome Project był przełomowym projektem genomu.
Zobacz też
- Archeogenetyka
- Chimera (genetyka)
- Genealogiczny test DNA
- Genetyka ewolucyjna człowieka
- Izolacja na odległość
- Hipoteza multiregionalna
- Neuroróżnorodność
- Rasa i genetyka
- Niedawna hipoteza pojedynczego pochodzenia
- Haplogrupy chromosomu Y w populacjach świata
Regionalny
- Projekt 1000 genomów
- Afrykańska domieszka w Europie
- Genetyczna historia Europy
- Historia genetyczna rdzennych ludów obu Ameryk
- Genetyczna historia Azji Południowej
- Genetyczna historia Wysp Brytyjskich
Projektowanie
Dalsza lektura
- Rasa, pochodzenie etniczne (październik 2005). „Wykorzystanie kategorii rasowych, etnicznych i przodków w badaniach genetyki człowieka” . American Journal of Human Genetics . 77 (4): 519–32. doi : 10.1086/491747 . PMC 1275602 . PMID 16175499 .
- Altmüller J, Palmer LJ, Fischer G, Scherb H, Wjst M (listopad 2001). „Skanowanie całego genomu złożonych chorób ludzkich: trudno znaleźć prawdziwe powiązanie” . American Journal of Human Genetics . 69 (5): 936–50. doi : 10.1086/324069 . PMC 1274370 . PMID 11565063 .
- Aoki K (2002). „Dobór płciowy jako przyczyna zmienności koloru ludzkiej skóry: ponowna analiza hipotezy Darwina”. Annals of Human Biology . 29 (6): 589–608. doi : 10.1080/0301446021000019144 . PMID 12573076 . S2CID 22703861 .
- Bamshad M, Wooding S, Salisbury BA, Stephens JC (sierpień 2004). „Dekonstrukcja związku między genetyką a rasą”. Recenzje przyrody. Genetyka . 5 (8): 598–609. doi : 10.1038/nrg1401 . PMID 15266342 . S2CID 12378279 . przedruk-zip
- Bamshad M, Wooding SP (luty 2003). „Podpisy doboru naturalnego w ludzkim genomie”. Recenzje przyrody. Genetyka . 4 (2): 99–111. doi : 10.1038/nrg999 . PMID 12560807 . S2CID 13722452 .
- Cann RL, Stoneking M, Wilson AC (1987). „DNA mitochondrialne i ewolucja człowieka”. Natura . 325 (6099): 31–36. Bibcode : 1987Natur.325...31C . doi : 10.1038/325031a0 . PMID 3025745 . S2CID 4285418 .
- Cardon LR, Abecasis GR (marzec 2003). „Używanie bloków haplotypów do mapowania loci cech złożonych człowieka” (PDF) . Trendy w genetyce . 19 (3): 135–40. doi : 10.1016/S0168-9525(03)00022-2 . PMID 12615007 .
- Cavalli-Sforza LL, Feldman MW (marzec 2003). „Zastosowanie podejść genetyki molekularnej do badania ewolucji człowieka”. Genetyka przyrody . 33 Dodatek (3s): 266–75. doi : 10.1038/ng1113 . PMID 12610536 . S2CID 8314161 .
- Collins FS (listopad 2004). „Co wiemy, a czego nie wiemy o„ rasie ”,„ pochodzeniu etnicznym ”, genetyce i zdrowiu u zarania ery genomu” . Genetyka przyrody . 36 (11 Dodatek): S13–15. doi : 10.1038/ng1436 . PMID 15507997 . S2CID 26968169 .
- Collins FS, Green ED, Guttmacher AE, Guyer MS (kwiecień 2003). „Wizja przyszłości badań nad genomiką”. Natura . 422 (6934): 835–47. Bibcode : 2003Natur.422..835C . doi : 10.1038/natura01626 . PMID 12695777 . S2CID 205209730 .
- Ebersberger I, Metzler D, Schwarz C, Pääbo S (czerwiec 2002). „Porównanie całego genomu sekwencji DNA między ludźmi i szympansami” . American Journal of Human Genetics . 70 (6): 1490–97. doi : 10.1086/340787 . PMC 379137 . PMID 11992255 .
- Edwards AW (sierpień 2003). „Różnorodność genetyczna człowieka: błąd Lewontina”. BioEseje . 25 (8): 798–801. doi : 10.1002/bies.10315 . PMID 12879450 .
- Foster MW, Sharp RR (październik 2004). „Poza rasą: w kierunku perspektywy całego genomu na populacje ludzkie i zmienność genetyczną”. Recenzje przyrody. Genetyka . 5 (10): 790–96. doi : 10.1038/nrg1452 . PMID 15510170 . S2CID 25764082 .
- Foster MW, Sharp RR, Freeman WL, Chino M, Bernsten D, Carter TH (czerwiec 1999). „Rola przeglądu społeczności w ocenie ryzyka związanego z badaniami nad zmiennością genetyczną człowieka” . American Journal of Human Genetics . 64 (6): 1719–27. doi : 10.1086/302415 . PMC 1377916 . PMID 10330360 .
- Gabriel SB, Schaffner SF, Nguyen H, Moore JM, Roy J, Blumenstiel B, Higgins J, DeFelice M, Lochner A, Faggart M, Liu-Cordero SN, Rotimi C, Adeyemo A, Cooper R, Ward R, Lander ES, Daly MJ, Altshuler D (czerwiec 2002). „Struktura bloków haplotypów w ludzkim genomie”. nauka . 296 (5576): 2225–29. Bibcode : 2002Sci...296.2225G . doi : 10.1126/science.1069424 . PMID 12029063 . S2CID 10069634 .
- Harding RM, Healy E, Ray AJ, Ellis NS, Flanagan N, Todd C, Dixon C, Sajantila A, Jackson IJ, Birch-Machin MA, Rees JL (kwiecień 2000). „Dowody na zmienne ciśnienia selektywne w MC1R” . American Journal of Human Genetics . 66 (4): 1351–61. doi : 10.1086/302863 . PMC 1288200 . PMID 10733465 .
- Ingman M, Kaessmann H, Pääbo S, Gyllensten U (grudzień 2000). „Zmienność genomu mitochondrialnego i pochodzenie współczesnych ludzi”. Natura . 408 (6813): 708–13. Bibcode : 2000Natur.408..708I . doi : 10.1038/35047064 . PMID 11130070 . S2CID 52850476 .
- Międzynarodowe Konsorcjum Hapmap (grudzień 2003). „Międzynarodowy projekt HapMap” . Natura . 426 (6968): 789–96. Bibcode : 2003Natur.426..789G . doi : 10.1038/natura02168 . hdl : 2027.42/62838 . PMID 14685227 . S2CID 4387110 .
- Międzynarodowe Konsorcjum Hapmap (czerwiec 2004). „Integracja etyki i nauki w międzynarodowym projekcie HapMap” . Recenzje przyrody. Genetyka . 5 (6): 467–75. doi : 10.1038/nrg1351 . PMC 2271136 . PMID 15153999 .
- Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC, Baldwin J i in. (luty 2001). „Wstępne sekwencjonowanie i analiza ludzkiego genomu” . Natura . 409 (6822): 860–921. Bibcode : 2001Natur.409..860L . doi : 10.1038/35057062 . PMID 11237011 .
- Jorde LB, Bamshad M, Rogers AR (luty 1998). „Wykorzystanie mitochondrialnych i jądrowych markerów DNA do rekonstrukcji ewolucji człowieka” (PDF) . BioEseje . 20 (2): 126–36. doi : 10.1002/(SICI)1521-1878(199802)20:2<126::AID-BIES5>3.0.CO;2-R . PMID 9631658 . S2CID 17203268 . Zarchiwizowane od oryginału (PDF) w dniu 28 listopada 2007 r . Źródło 28 października 2007 .
- Jorde LB, Watkins WS, Bamshad MJ, Dixon ME, Ricker CE, Seielstad MT, Batzer MA (marzec 2000). „Dystrybucja różnorodności genetycznej człowieka: porównanie danych mitochondrialnych, autosomalnych i chromosomu Y” . American Journal of Human Genetics . 66 (3): 979–88. doi : 10.1086/302825 . PMC 1288178 . PMID 10712212 .
- Jorde LB, Watkins WS, Kere J, Nyman D, Eriksson AW (2000). „Mapowanie genów w izolowanych populacjach: nowe role dla starych przyjaciół?”. Dziedziczność człowieka . 50 (1): 57–65. doi : 10.1159/000022891 . PMID 10545758 . S2CID 26960216 .
- Kaessmann H, Heissig F, von Haeseler A, Pääbo S (maj 1999). „Zmienność sekwencji DNA w niekodującym regionie o niskiej rekombinacji na ludzkim chromosomie X”. Genetyka przyrody . 22 (1): 78–81. doi : 10.1038/8785 . PMID 10319866 . S2CID 9153915 .
- Kaessmann H, Wiebe V, Weiss G, Pääbo S (luty 2001). „Sekwencje DNA małp człekokształtnych ujawniają zmniejszoną różnorodność i ekspansję u ludzi”. Genetyka przyrody . 27 (2): 155–56. doi : 10.1038/84773 . PMID 11175781 . S2CID 19384784 .
- Keita SO, Kittles RA (1997). „Trwałość myślenia rasowego i mit różnic rasowych”. amerykański antropolog . 99 (3): 534–44. doi : 10.1525/aa.1997.99.3.534 .
- Znaki J. (1995). Różnorodność biologiczna człowieka: geny, rasa i historia . Transakcja Aldine . ISBN 978-0-202-02033-4 .
- Mountain JL, Risch N (listopad 2004). „Ocena wkładu genetycznego w różnice fenotypowe między grupami„ rasowymi ”i„ etnicznymi ”” . Genetyka przyrody . 36 (11 Dodatek): S48–53. doi : 10.1038/ng1456 . PMID 15508003 .
- Pääbo S (styczeń 2003). „Mozaika, która jest naszym genomem” . Natura . 421 (6921): 409–12. Bibcode : 2003Natur.421..409P . doi : 10.1038/natura01400 . PMID 12540910 .
- Ramachandran S, Deshpande O, Roseman CC, Rosenberg NA, Feldman MW, Cavalli-Sforza LL (listopad 2005). „Wsparcie wynikające ze związku odległości genetycznej i geograficznej w populacjach ludzkich dla seryjnego efektu założyciela pochodzącego z Afryki” . Proceedings of the National Academy of Sciences of the United States of America . 102 (44): 15942–47. Bibcode : 2005PNAS..10215942R . doi : 10.1073/pnas.0507611102 . PMC 1276087 . PMID 16243969 .
- Relethford JH (sierpień 2002). „Podział globalnej różnorodności genetycznej człowieka na podstawie kraniometrii i koloru skóry”. American Journal of Physical Anthropology . 118 (4): 393–98. CiteSeerX 10.1.1.473.5972 . doi : 10.1002/ajpa.10079 . PMID 12124919 . S2CID 8717358 .
- Sankar P, Cho MK (listopad 2002). „Genetyka. Ku nowemu słownictwu zmienności genetycznej człowieka” . nauka . 298 (5597): 1337–38. doi : 10.1126/science.1074447 . PMC 2271140 . PMID 12434037 .
- Sankar P, Cho MK, Condit CM, Hunt LM, Koenig B, Marshall P, Lee SS, Spicer P (czerwiec 2004). „Badania genetyczne i różnice zdrowotne” . JAMA . 291 (24): 2985–89. doi : 10.1001/jama.291.24.2985 . PMC 2271142 . PMID 15213210 .
- Serre D, Pääbo S (wrzesień 2004). „Dowody na gradienty różnorodności genetycznej człowieka w obrębie kontynentów i między nimi” . Badania genomu . 14 (9): 1679–85. doi : 10.1101/gr.2529604 . PMC 515312 . PMID 15342553 .
- Templeton AR (1998). „Rasy ludzkie: perspektywa genetyczna i ewolucyjna”. amerykański antropolog . 100 (3): 632–50. doi : 10.1525/aa.1998.100.3.632 .
- Weissa KM (1998). „Pogodzenie się z ludzką zmiennością”. Roczny przegląd antropologii . 27 : 273–300. doi : 10.1146/annurev.anthro.27.1.273 .
- Weiss KM, Terwilliger JD (październik 2000). „Ile chorób potrzeba do zmapowania genu za pomocą SNP?”. Genetyka przyrody . 26 (2): 151–57. doi : 10.1038/79866 . PMID 11017069 . S2CID 685795 .
- Yu N, Jensen-Seaman MI, Chemnick L, Kidd JR, Deinard AS, Ryder O, Kidd KK, Li WH (sierpień 2003). „Niska różnorodność nukleotydów u szympansów i bonobo” . Genetyka . 164 (4): 1511–18. doi : 10.1093/genetics/164.4.1511 . PMC 1462640 . PMID 12930756 .
- Zietkiewicz E, Yotova V, Gehl D, Wambach T, Arrieta I, Batzer M, Cole DE, Hechtman P, Kaplan F, Modiano D, Moisan JP, Michalski R, Labuda D (listopad 2003). „Haplotypy w segmencie DNA dystrofiny wskazują na mozaikowe pochodzenie różnorodności współczesnego człowieka” . American Journal of Human Genetics . 73 (5): 994–1015. doi : 10.1086/378777 . PMC 1180505 . PMID 14513410 .
- Pennisi E (grudzień 2007). „Przełom roku. Zmienność genetyczna człowieka” . nauka . 318 (5858): 1842–43. doi : 10.1126/science.318.5858.1842 . PMID 18096770 .
- Ramachandran S, Tang H, Gutenkunst RN, Bustamante CD (2010). „Genetyka i genomika struktury populacji ludzkiej”. W Speicher MR, Antonarakis SE, Motulsky AG (red.). Vogel and Motulsky's Human Genetics: Problems and Approaches (wyd. 4). Springera . ISBN 978-3-540-37653-8 .