Motyw rozpoznawania RNA
| Motyw rozpoznawania RNA. (inaczej domena RRM, RBD lub RNP) | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| Identyfikatory | |||||||||||
| Symbol | RRM_1 | ||||||||||
| Pfam | PF00076 | ||||||||||
| Klan Pfamów | RRM/CL0221 RRM | ||||||||||
| InterPro | IPR000504 | ||||||||||
| PROZYT | PDOC00030 | ||||||||||
| SCOP2 | 1sxl / ZAKRES / SUPFAM | ||||||||||
| |||||||||||
Motyw rozpoznawania RNA, RNP-1, jest przypuszczalną domeną wiążącą RNA składającą się z około 90 aminokwasów, o których wiadomo, że wiążą jednoniciowe RNA. Stwierdzono go w wielu eukariotycznych .
Największą grupą białek wiążących jednoniciowy RNA jest rodzina eukariotycznych motywów rozpoznawania RNA (RRM), która zawiera ośmioaminokwasową sekwencję konsensusową RNP-1.
Białka RRM mają różnorodne preferencje i funkcje wiązania RNA i obejmują heterogenne jądrowe rybonukleoproteiny ( hnRNP ), białka biorące udział w regulacji alternatywnego splicingu (SR, U2AF2 , Sxl), składniki białkowe małych jądrowych rybonukleoprotein (U1 i U2 snRNP ) oraz białka regulujące stabilność i translację RNA ( PABP , La, Hu). RRM w heterodimerycznym współczynniku splicingu U2 Współczynnik pomocniczy snRNP wydaje się mieć dwie domeny podobne do RRM ze specjalistycznymi cechami rozpoznawania białek. Motyw pojawia się również w kilku białkach wiążących jednoniciowy DNA.
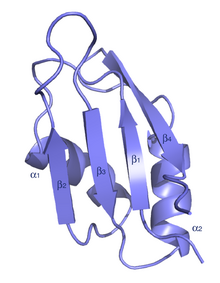
Typowy RRM składa się z czterech antyrównoległych nici beta i dwóch alfa-helis ułożonych w fałd beta-alfa-beta-beta-alfa-beta z łańcuchami bocznymi ułożonymi w stos z zasadami RNA. W niektórych przypadkach podczas wiązania RNA obecna jest trzecia helisa. Przegląd RRM znajduje się w wielu publikacjach.
Białka ludzkie zawierające tę domenę
A2BP1 ; ACF ; BOLL ; BRUNOL4 ; BRUNOL5; BRUNOL6; CCBL2; CGI-96; CIRBP ; CNOT4 ; CPEB2; CPEB3; CPEB4; CPSF7 ; CSTF2 ; CSTF2T ; CUGBP1 ; CUGBP2 ; D10S102; DAZ1 ; DAZ2 ; DAZ3 ; DAZ4; DAZAP1 ; DAZL ; DNAJC17; DND1; EIF3S4; EIF3S9; EIF4B ; EIF4H ; ELAVL1 ; ELAVL2 ; ELAVL3 ; ELAVL4 ; ENOX1; ENOX2 ; EWSR1 ; FUS ; FUSIP1 ; G3BP; G3BP1 ; G3BP2 ; GRSF1; HNRNPL ; HNRPA0; HNRPA1 ; HNRPA2B1 ; HNRPA3 ; HNRPAB ; HNRPC ; HNRPCL1; HNRPD ; HNRPDL ; HNRPF ; HNRPH1 ; HNRPH2 ; HNRPH3 ; HNRPL; HNRPLL; HNRPM; HNRPR; HRNBP1; HSU53209; HTATSF1 ; IGF2BP1 ; IGF2BP2 ; IGF2BP3 ; LARP7; MKI67IP ; MSI1 ; MSI2 ; MSSP-2; MTFSD; MYEF2 ; NCBP2; NCL ; NOL8 ; NIE ; P14; PABPC1 ; PABPC1L; PABPC3 ; PABPC4 ; PABPC5 ; PABPN1 ; POLDIP3 ; PPARGC1; PPARGC1A ; PPARGC1B ; PPIE ; PPIL4 ; PPRC1 ; PSPC1; PTBP1 ; PTBP2 ; PUF60 ; Rajd ; RALYL ; RAVER1 ; RAVER2; RBM10 ; RBM11 ; RBM12 ; RBM12B; RBM14 ; RBM15 ; RBM15B; RBM16; RBM17 ; RBM18; RBM19 ; RBM22 ; RBM23 ; RBM24; RBM25 ; RBM26 ; RBM27; RBM28 ; RBM3 ; RBM32B; RBM33 ; RBM34 ; RBM35A; RBM35B; RBM38; RBM39 ; RBM4 ; RBM41; RBM42; RBM44; RBM45; RBM46; RBM47 ; RBM4B; RBM5 ; RBM7 ; RBM8A ; RBM9 ; RBMS1 ; RBMS2; RBMS3 ; RBMX ; RBMX2; RBMXL2; RBMY1A1 ; RBMY1B; RBMY1E; RBMY1F; RBMY2FP; RBPMS ; RBPMS2; RDBP ; RNPC3; RNPC4; RNPS1 ; Pręt1; SAFB ; SAFB2 ; SART3 ; SETD1A ; SF3B14 ; SF3B4 ; SFPQ ; MSSF1 ; MSSF10 ; MSSF11 ; MSSF12 ; MSSF15; MSSF2 ; MSSF2B; MSSF3 ; MSSF4 ; MSSF5 ; MSSF6 ; MSSF7 ; MSSF9 ; SLIRP; SLTM; SNRP70 ; SNRPA ; SNRPB2 ; SPEN ; SR140 ; SRRP35; SSB ; SYNCRIP ; TAF15 ; TARDBP ; THOC4 ; TIA1 ; TIAL1 ; TNRC4; TNRC6C; TRA2A ; TRSPAP1; TUT1; U1SNRNPBP; U2AF1 ; U2AF2 ; UHMK1 ; ZCRB1; ZNF638 ; ZRSR1 ; ZRSR2 ;
Linki zewnętrzne
- Klasa motywu motywu liniowego eukariotycznego LIG_ULM_U2AF65_1